BLAT (bioinformatyka)
| Deweloperzy | Jim Kent z UCSC |
|---|---|
| Magazyn | |
| Typ | Narzędzie bioinformatyczne |
| Licencja | bezpłatnie do użytku niekomercyjnego , do użytku komercyjnego , źródło dostępne |
| Strona internetowa |
BLAT ( narzędzie do dopasowywania podobne do BLAST ) to algorytm dopasowywania sekwencji parami , który został opracowany przez Jima Kenta z University of California Santa Cruz (UCSC) na początku XXI wieku w celu pomocy w składaniu i adnotacji ludzkiego genomu . Został zaprojektowany przede wszystkim w celu skrócenia czasu potrzebnego do dopasowania milionów mysich odczytów genomowych i wyrażonych znaczników sekwencji do sekwencji ludzkiego genomu. Narzędzia do dopasowywania tamtych czasów nie były w stanie wykonać tych operacji w sposób, który pozwoliłby na regularną aktualizację zestawu ludzkiego genomu. W porównaniu z wcześniej istniejącymi narzędziami, BLAT był ~500 razy szybszy przy wykonywaniu mRNA / DNA i ~50 razy szybszy przy dopasowaniu białko /białko.
Przegląd
BLAT jest jednym z wielu algorytmów opracowanych do analizy i porównywania sekwencji biologicznych, takich jak DNA, RNA i białka, z głównym celem wnioskowania o homologii w celu odkrycia biologicznej funkcji sekwencji genomowych. Nie ma gwarancji znalezienia matematycznie optymalnego dopasowania między dwiema sekwencjami, jak to robią klasyczne programowania dynamicznego Needlemana-Wunscha i Smitha-Watermana ; raczej najpierw próbuje szybko wykryć krótkie sekwencje, które z większym prawdopodobieństwem będą homologiczne, a następnie dopasowuje i dalej rozszerza regiony homologiczne. Jest podobny do heurystycznej rodziny algorytmów BLAST, ale każde narzędzie próbowało poradzić sobie z problemem dopasowania sekwencji biologicznych w szybki i skuteczny sposób, próbując różnych technik algorytmicznych.
Zastosowania BLAT
BLAT można stosować do dopasowywania sekwencji DNA, jak również białek i sekwencji nukleotydów poddanych translacji (mRNA lub DNA). Został zaprojektowany tak, aby działał najlepiej w sekwencjach o dużym podobieństwie. Wyszukiwanie DNA jest najskuteczniejsze w przypadku naczelnych, a wyszukiwanie białek jest skuteczne w przypadku kręgowców lądowych. Ponadto zapytania dotyczące białek lub sekwencji po translacji są bardziej skuteczne w identyfikowaniu odległych dopasowań i analizie międzygatunkowej niż zapytania o sekwencje DNA. Typowe zastosowania BLAT obejmują:
- Dopasowanie wielu sekwencji mRNA do zespołu genomu w celu wywnioskowania ich współrzędnych genomowych;
- Dopasowanie sekwencji białka lub mRNA z jednego gatunku do bazy danych sekwencji z innego gatunku w celu określenia homologii. Pod warunkiem, że te dwa gatunki nie są zbyt rozbieżne, wyrównanie międzygatunkowe jest ogólnie skuteczne w przypadku BLAT. Jest to możliwe, ponieważ BLAT nie wymaga doskonałych dopasowań, ale raczej akceptuje niedopasowania w wyrównaniach;
- BLAT można zastosować do dopasowania dwóch sekwencji białkowych. Nie jest to jednak narzędzie z wyboru do tego typu wyrównań. BLASTP, narzędzie Standard Protein BLAST , jest bardziej wydajne w dopasowywaniu białek do białek;
- Wyznaczanie rozmieszczenia regionów egzonowych i intronowych genu;
- Wykrywanie członków rodziny genów określonego zapytania genowego;
- Wyświetlanie sekwencji kodującej białko określonego genu.
BLAT ma na celu znalezienie dopasowań między sekwencjami o długości co najmniej 40 zasad, które mają ≥95% identyczności nukleotydów lub ≥80% identyczności translowanych białek.
Proces
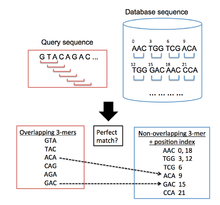
BLAT służy do znajdowania regionów w docelowej bazie danych genomu, które są podobne do badanej sekwencji zapytania. Ogólny proces algorytmiczny, po którym następuje BLAT, jest podobny do BLAST , ponieważ najpierw wyszukuje krótkie segmenty w bazie danych i sekwencje zapytań, które mają określoną liczbę pasujących elementów. Te nasiona dopasowania są następnie rozciągane w obu kierunkach sekwencji w celu utworzenia par o wysokiej punktacji. Jednak BLAT wykorzystuje inne podejście do indeksowania niż BLAST, co pozwala mu szybko skanować bardzo duże genomowe i białkowe bazy danych w poszukiwaniu podobieństw do sekwencji zapytania. Robi to, przechowując w pamięci indeksowaną listę ( tabelę mieszającą ) docelowej bazy danych, co znacznie skraca czas potrzebny na porównanie sekwencji zapytań z docelową bazą danych. Indeks ten jest tworzony na podstawie współrzędnych wszystkich nienakładających się k-merów (słów z k literami) w docelowej bazie danych, z wyjątkiem k-merów o dużej liczbie powtórzeń. Następnie BLAT tworzy listę wszystkich nakładających się k-merów z sekwencji zapytania i wyszukuje je w docelowej bazie danych, tworząc listę trafień, w których występują dopasowania między sekwencjami (Rysunek 1 ilustruje ten proces).
Etap wyszukiwania
Istnieją trzy różne strategie stosowane w celu poszukiwania kandydujących regionów homologicznych:
- Pierwsza metoda wymaga pojedynczych doskonałych dopasowań między zapytaniem a sekwencjami bazy danych, tj. dwa k-merowe słowa są dokładnie takie same. To podejście nie jest uważane za najbardziej praktyczne. Dzieje się tak, ponieważ mały rozmiar k-meru jest niezbędny do osiągnięcia wysokiego poziomu czułości, ale zwiększa to liczbę fałszywych trafień dodatnich, zwiększając w ten sposób ilość czasu spędzanego na etapie dopasowywania algorytmu.
- Druga metoda pozwala na co najmniej jedno niedopasowanie między dwoma k-merowymi słowami. Zmniejsza to liczbę fałszywych alarmów, umożliwiając większe rozmiary k-merów, które są mniej kosztowne obliczeniowo w obsłudze niż te wytwarzane poprzednią metodą. Ta metoda jest bardzo skuteczna w identyfikacji małych regionów homologicznych.
- Trzecia metoda wymaga wielu idealnych dopasowań, które znajdują się blisko siebie. Jak pokazuje Kent, jest to bardzo skuteczna technika zdolna do uwzględnienia małych insercji i delecji w regionach homologicznych.
Podczas dopasowywania nukleotydów BLAT wykorzystuje trzecią metodę wymagającą dwóch doskonałych dopasowań słów o rozmiarze 11 (11-merów). Podczas dopasowywania białek wersja BLAT określa zastosowaną metodologię wyszukiwania: gdy używana jest wersja klient/serwer, BLAT wyszukuje trzy idealne dopasowania 4-merowe; gdy używana jest wersja samodzielna, BLAT wyszukuje jedną idealną 5-mer między zapytaniem a sekwencjami bazy danych.
BLAT kontra BLAST
Niektóre różnice między BLAT i BLAST przedstawiono poniżej:
- BLAT indeksuje bazę danych genomu/białka, zachowuje indeks w pamięci, a następnie skanuje sekwencję zapytania w poszukiwaniu dopasowań. Z drugiej strony BLAST buduje indeks sekwencji zapytań i przeszukuje bazę danych w poszukiwaniu dopasowań. Wariant BLAST o nazwie MegaBLAST indeksuje 4 bazy danych, aby przyspieszyć wyrównanie.
- BLAT może rozciągać się na wiele doskonałych i prawie idealnych dopasowań (domyślnie są to 2 idealne dopasowania o długości 11 do wyszukiwania nukleotydów i 3 idealne dopasowania o długości 4 do wyszukiwania białek), podczas gdy BLAST rozciąga się tylko wtedy, gdy jedno lub dwa dopasowania występują blisko siebie.
- BLAT łączy każdy obszar homologiczny między dwiema sekwencjami w jedno większe dopasowanie, w przeciwieństwie do BLAST, który zwraca każdy obszar homologiczny jako oddzielne dopasowanie lokalne. Wynikiem BLAST jest lista eksonów z każdym dopasowaniem rozciągającym się tuż za końcem eksonu. Jednak BLAT prawidłowo umieszcza każdą zasadę mRNA w genomie, używając każdej zasady tylko raz i może być wykorzystany do identyfikacji granic intron -egzon (tj. miejsc splicingowych ).
- BLAT jest mniej czuły niż BLAST.
Użycie programu
BLAT może być używany jako internetowy program serwer-klient lub jako samodzielny program.
Serwer-klient
Dostęp do internetowej aplikacji BLAT można uzyskać z witryny UCSC Genome Bioinformatics Site. Tworzenie indeksu jest stosunkowo powolną procedurą. Dlatego każdy zespół genomu używany przez internetowy BLAT jest powiązany z serwerem BLAT, aby mieć wstępnie obliczony indeks dostępny do dopasowania. Te internetowe serwery BLAT przechowują indeks w pamięci, aby użytkownicy mogli wprowadzać sekwencje zapytań.
Po przesłaniu/wklejeniu sekwencji zapytania do pola wyszukiwania użytkownik może wybrać różne parametry, takie jak genom gatunku, który ma być docelowy (obecnie dostępnych jest ponad 50 gatunków) oraz wersję tego genomu (na przykład ludzki genom ma cztery zestawy do wyboru), typ zapytania (tj. czy sekwencja odnosi się do DNA, białka itp.) i ustawienia wyjściowe (tj. jak sortować i wizualizować dane wyjściowe). Użytkownik może następnie uruchomić wyszukiwanie, przesyłając zapytanie lub używając wyszukiwania BLAT „Szczęśliwy traf”.
Bhagwat i in. dostarczyć protokoły krok po kroku, jak używać BLAT do:
- Mapowanie sekwencji mRNA/cDNA na sekwencję genomową;
- Zmapuj sekwencję białka do genomu;
- Przeprowadź wyszukiwanie homologii.
Wejście
BLAT może obsługiwać długie sekwencje baz danych, jednak jest bardziej efektywny w przypadku krótkich sekwencji zapytań niż długich sekwencji zapytań. Kent zaleca maksymalną długość zapytania wynoszącą 200 000 zasad. Przeglądarka UCSC ogranicza sekwencje zapytań do mniej niż 25 000 liter (tj. nukleotydów ) w przypadku wyszukiwania DNA i mniej niż 10 000 liter (tj. aminokwasów ) w przypadku wyszukiwania białek i sekwencji po translacji.
Genom wyszukiwania BLAT dostępny na stronie internetowej UCSC akceptuje sekwencje zapytań jako tekst (wycięty i wklejony do pola zapytania) lub przesłane jako pliki tekstowe. BLAT Search Genome może akceptować jednocześnie wiele sekwencji tego samego typu, maksymalnie do 25. W przypadku wielu sekwencji całkowita liczba nukleotydów nie może przekraczać 50 000 w przypadku wyszukiwania DNA lub 25 000 liter w przypadku wyszukiwania białek lub sekwencji po translacji. Przykład przeszukiwania docelowej bazy danych za pomocą sekwencji zapytania DNA przedstawiono na rysunku 2.
Wyjście
Wyszukiwanie BLAT zwraca listę wyników uporządkowanych malejąco na podstawie wyniku. Zwracane są następujące informacje: wynik dopasowania, region sekwencji zapytania, który pasuje do sekwencji bazy danych, rozmiar sekwencji zapytania, poziom identyczności jako procent dopasowania oraz chromosom i pozycja, w której sekwencja zapytania mapy do. Bhagwat i in. opisać sposób obliczania miar „Wynik” i „Tożsamość” BLAT.
Dla każdego wyniku wyszukiwania użytkownik otrzymuje łącze do przeglądarki genomu UCSC, aby mógł zwizualizować dopasowanie na chromosomie. Jest to główna zaleta internetowego BLAT w porównaniu z samodzielnym BLAT. Użytkownik jest w stanie uzyskać informacje biologiczne związane z dopasowaniem, takie jak informacje o genie, do którego może pasować zapytanie. Użytkownik otrzymuje również łącze do przeglądania dopasowania sekwencji zapytania do zestawu genomu. Dopasowania między zapytaniem a zestawem genomu są niebieskie, a granice dopasowań mają jaśniejszy kolor. Te granice eksonów wskazują miejsca składania. Wynik wyszukiwania „Szczęśliwy traf” zwraca wyrównanie o najwyższej punktacji dla pierwszej sekwencji zapytania na podstawie opcji sortowania danych wyjściowych wybranej przez użytkownika.
Samodzielny
Samodzielny BLAT jest bardziej odpowiedni do uruchamiania wsadowego i bardziej wydajny niż internetowy BLAT. Jest bardziej wydajny, ponieważ jest w stanie przechowywać genom w pamięci, w przeciwieństwie do aplikacji internetowej, która przechowuje tylko indeks w pamięci.
Licencja
Zarówno źródło, jak i prekompilowane pliki binarne BLAT są swobodnie dostępne do użytku akademickiego i osobistego. Komercyjna licencja na samodzielny BLAT jest dystrybuowana przez Kent Informatics, Inc.
Zobacz też
- BLAST Podstawowe narzędzie do wyszukiwania lokalnego wyrównania
- Oprogramowanie do dopasowywania sekwencji