Hak AT
| AT-hook | |||||||||
|---|---|---|---|---|---|---|---|---|---|
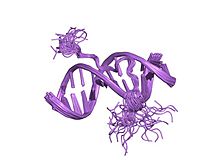 roztwór struktury kompleksu drugiej domeny wiążącej dna ludzkiego hmg-i(y) związanej z dodekamerem DNA zawierającym miejsce prdii promotora interferonu-beta, nmr, 35 struktur
| |||||||||
| Identyfikatory | |||||||||
| Symbol | AT_hak | ||||||||
| Pfam | PF02178 | ||||||||
| InterPro | IPR017956 | ||||||||
| MĄDRY | AT_hak | ||||||||
| SCOP2 | 2eze / SCOPe / SUPFAM | ||||||||
| |||||||||

Hak AT to motyw wiążący DNA obecny w wielu białkach, w tym w białkach grupy o wysokiej ruchliwości (HMG) , białkach roślinnych wiążących DNA i białku hBRG1, centralnej ATPazie ludzkiego przełączania /niefermentującej sacharozy (SWI/ SNF) kompleks przebudowy .
Motyw ten składa się z konserwatywnej, palindromicznej, rdzeniowej sekwencji prolina - arginina - glicyna - arginina - prolina , chociaż niektóre haczyki AT zawierają tylko jedną prolinę w sekwencji rdzeniowej. Haczyki AT obejmują również zmienną liczbę dodatnio naładowanych lizyny i argininy po obu stronach sekwencji rdzenia. Hak AT wiąże się z mniejszym rowkiem DNA bogatego w adeninę - tyminę (AT), stąd AT w nazwie. Reszta nazwy wywodzi się od przewidywanego asparaginy / asparaginianu w najwcześniejszych hakach AT zgłoszonych w 1990 r. W 1997 r. Badania strukturalne z wykorzystaniem NMR wykazały, że haczyk AT związany z DNA przyjął kształt półksiężyca lub haczyka wokół mniejszego rowka docelowej nici DNA (na zdjęciu po prawej). Białka HMGA zawierają trzy haczyki AT, chociaż niektóre białka zawierają ich aż 30. Optymalne sekwencje wiążące dla białek haczyków AT to powtórzenia postaci (ATAA) n lub (TATT) n , chociaż optymalne sekwencje wiążące dla sekwencji rdzeniowej haka AT to AAAT i AATT.