Logo sekwencji

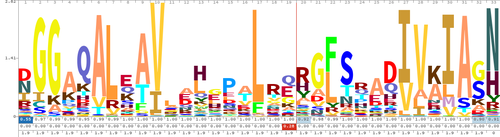
W bioinformatyce logo sekwencji jest graficzną reprezentacją zachowania sekwencji nukleotydów (w nici DNA / RNA ) lub aminokwasów (w sekwencjach białek ) . Logo sekwencji jest tworzone ze zbioru dopasowanych sekwencji i przedstawia sekwencję konsensusową oraz różnorodność sekwencji. Logo sekwencji są często używane do przedstawiania właściwości sekwencji, takich jak miejsca wiązania białek w DNA lub jednostki funkcjonalne w białkach.
Przegląd
Logo sekwencyjne składa się ze stosu liter w każdej pozycji. Względne rozmiary liter wskazują ich częstotliwość w sekwencjach. Całkowita wysokość liter przedstawia zawartość informacyjną pozycji w bitach.
Tworzenie logo
Aby utworzyć logo sekwencji, pokrewne sekwencje DNA, RNA lub białek lub sekwencje DNA, które mają wspólne konserwatywne miejsca wiązania, są dopasowywane tak, że najbardziej konserwatywne części tworzą dobre dopasowania. Następnie można utworzyć logo sekwencji z zachowanego dopasowania wielu sekwencji . Logo sekwencji pokaże, jak dobrze reszty są konserwowane w każdej pozycji: im większa liczba reszt, tym wyższe będą litery, ponieważ lepsza jest konserwacja w tej pozycji. Różne reszty w tej samej pozycji są skalowane zgodnie z ich częstotliwością. Wysokość całego stosu pozostałości jest informacją mierzoną w bity . Logo sekwencji można wykorzystać do przedstawienia konserwatywnych miejsc wiązania DNA , w których wiążą się czynniki transkrypcyjne .
Treść informacji (oś y) pozycji jest określona przez:
- dla aminokwasów
- dla kwasów nukleinowych
gdzie niepewnością (czasami nazywaną entropią Shannona pozycji ja
Tutaj jest względną częstotliwością zasady aminokwasu w pozycji i [ wymagane wyjaśnienie ] to poprawka na małą próbkę dla . Wysokość litery w kolumnie jest dany przez
Przybliżenie dla poprawki na małą próbkę, jest podane przez:
gdzie dla nukleotydów, 20 dla aminokwasów i sekwencji w dopasowaniu.
Logo konsensusu
Logo konsensusu to uproszczona odmiana logo sekwencji, którą można osadzić w formacie tekstowym. Podobnie jak logo sekwencji, logo konsensusu jest tworzone ze zbioru dopasowanych sekwencji białek lub DNA/RNA i przekazuje informacje o zachowaniu każdej pozycji motywu sekwencji lub dopasowania sekwencji . Jednak logo konsensusu wyświetla tylko informacje o ochronie, a nie wyraźnie informacje o częstotliwości każdego nukleotydu lub aminokwasu na każdej pozycji. Zamiast stosu składającego się z kilku znaków, oznaczającego względną częstotliwość każdego znaku, logo konsensusu przedstawia stopień zachowania każdej pozycji przy użyciu wysokości znaku konsensusu na tej pozycji.
Zalety i wady
Główną i oczywistą zaletą logo konsensusu w porównaniu z logo sekwencyjnym jest ich zdolność do osadzania jako tekstu w dowolnym formacie RTF obsługującym edytor/przeglądarkę, a zatem w manuskryptach naukowych. Jak opisano powyżej, logo konsensusu jest skrzyżowaniem logo sekwencji i sekwencji konsensusu . W rezultacie, w porównaniu do logo sekwencji, logo konsensusu pomija informacje (względny wkład każdego znaku w zachowanie tej pozycji w motywie/ustawieniu). W związku z tym logo sekwencyjne powinno być stosowane preferencyjnie, gdy tylko jest to możliwe. To powiedziawszy, potrzeba uwzględnienia figur graficznych w celu wyświetlenia logo sekwencji utrwaliła stosowanie sekwencji konsensusowych w manuskryptach naukowych, mimo że nie przekazują one informacji zarówno o zachowaniu, jak i częstotliwości. Logo konsensusu stanowi zatem ulepszenie w stosunku do sekwencji konsensusu, ilekroć informacja o motywie / dopasowaniu musi być ograniczona do tekstu.
Rozszerzenia
Ukryte modele Markowa (HMM) uwzględniają nie tylko zawartość informacyjną wyrównanych pozycji w dopasowaniu, ale także wstawienia i usunięcia. W logo sekwencji HMM używanym przez Pfam , dodano trzy wiersze, aby wskazać częstotliwość zajętości (obecności) i wkładania, a także oczekiwaną długość wkładania.
Zobacz też
Linki zewnętrzne
- Utwórz logo sekwencji w przeglądarce, offline
- Logo sekwencji GitHub w natywnym JavaScript
- Jak czytać logo sekwencji .
- Zalecenia dotyczące tworzenia logo sekwencji .
- Erill, I., „Delikatne wprowadzenie do treści informacji w miejscach wiązania czynników transkrypcyjnych”, Eprint
- Czym jest (w) logo sekwencji?












