Wewnętrzne miejsce wejścia rybosomu wirusa zapalenia wątroby typu A (IRES)
| Wewnętrzne miejsce wejścia rybosomu wirusa zapalenia wątroby typu A (IRES) | |
|---|---|
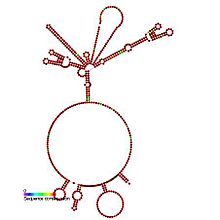 Przewidywana struktura drugorzędowa i konserwacja sekwencji IRES_HepA
| |
| identyfikatorów | |
| Symbol | IRES_HepA |
| Alt. Symbolika | HepA_IRES |
| Rfam | RF00228 |
| Inne dane | |
| typ RNA | Cis-reg ; IRES |
| Domeny | Wirusy |
| IŚĆ | GO: 0043022 |
| WIĘC | SO: 0000243 |
| Struktury PDB | PDBe |
Ta rodzina reprezentuje wewnętrzne miejsce wejścia rybosomu (IRES) wirusa zapalenia wątroby typu A. HAV IRES to sekwencja o długości 450 nukleotydów zlokalizowana w 5' UTR ( region nieulegający translacji) o długości 735 nt genomu RNA wirusa zapalenia wątroby typu A. Elementy IRES umożliwiają niezależną od czapeczki i końca translację mRNA w komórce gospodarza. IRES osiąga to poprzez pośredniczenie w wewnętrznej inicjacji translacji poprzez rekrutację rybosomalnego kompleksu preinicjacyjnego 40S bezpośrednio do kodonu inicjacyjnego i eliminuje wymóg eukariotycznego czynnika inicjacji , eIF4F.
Historia i tło
IRES został po raz pierwszy odkryty w genomie RNA picornaviridae przez Normana Sonenberga w 1988 r. Początkowo zidentyfikowany 5' UTR wirusa polio (PV), który kierował wewnętrzną inicjacją syntezy białek, nazwano lądowiskiem rybosomu (RLP). Termin ten został wkrótce potem zastąpiony przez wewnętrzne miejsce wejścia rybosomu, które jest również częściej używane dzisiaj. W 1988 roku tylko wirus zapalenia mózgu i mięśnia sercowego ( EMCV ) i PV były używane do zademonstrowania zdolności IRES. Badania identyfikujące IRES w HAV zostały przeprowadzone w 1993 roku przez Michaela J. Glassa, Xi-Yu Jia i Donalda F. Summersa. Ich badania wykazały, że IRES HAV znajdował się poniżej nukleotydu 45 i obejmował aż do nukleotydu 734. Niezależna od czapeczki wewnętrzna inicjacja syntezy białek różni się od normalnej komórkowej zależnej od czapeczki inicjacji translacji. eIF4 (eukariotyczny czynnik inicjacji 4) jako cały kompleks składający się z eIF4A, eIF4B, eIF4E i eIF4G. eIF4F jest używany w odniesieniu do kompleksu, który obejmuje eIF4E, 4G i 4A. W zależnej od czapeczki inicjacji translacji eIF4F wiąże się z czapeczką m7G na końcu 5' UTR, rekrutując 40S małej podjednostki rybosomu i skanuje w dół w poszukiwaniu kodonu startowego AUG. Wykazano, że inicjacja wewnętrzna niezależna od czapeczki zakłóca część eIF4 lub cały kompleks.
Funkcjonować
Niepowleczony w cytoplazmie genom HAV inicjuje translację niezależnie od czapeczki 5' w celu syntezy swoich białek wirusowych. Struktura drugorzędowa HAV IRES jest zarówno konieczna, jak i wystarczająca, aby genom mógł rekrutować rybosom i zainicjować translację. Rybosom komórki gospodarza rozpoznaje IRES i wejdzie bezpośrednio w sekwencję, zamiast skanować od końca 5'. Genom HAV nie koduje białek, które mają zdolność wyłączania białek gospodarza. Dlatego HAV IRES musi konkurować z mRNA z czapeczką m7G komórki gospodarza. Niestety inicjacja translacji przez HAV IRES nie jest tak wydajna, jak typowa czapeczka m7G komórki gospodarza. Chociaż struktura HAV IRES ma powinowactwo do eIF4F, jego powinowactwo nie jest tak wysokie, jak mRNA z czapeczką komórki gospodarza. Skutkuje to dłuższym okresem wymaganym do osiągnięcia maksymalnego wydalenia wirusa. Powoduje to również śmierć komórki występującą tylko w odpowiedzi immunologicznej gospodarza, a nie w wyniku lizy.
Tłumienie IRES
Ponieważ eIF4F odgrywa istotną rolę w inicjacji HAV IRES, jest celem tłumienia IRES. Rozszczepienie eIF4G , rusztowania białkowego eIF4F, przez specyficzne dla sekwencji proteazy, proteazę 2A lub L-proteazę, spowoduje wysoce zahamowaną aktywność HAV IRES. Te proteazy są kodowane przez innych przedstawicieli rodziny picornaviridae rodzina. Na przykład wirus pryszczycy (FMDV) koduje te proteazy, aby hamować translację komórkowego mRNA, jednocześnie umożliwiając translację wirusowego RNA. Wymóg nienaruszonego eIF4G do inicjacji IRES jest specyficzny dla HAV IRES wśród innych pikornawirusów. Białko I wiążące eIF4E (4E-BP1) będzie również interferować z białkiem eIF4G. 4E-BP1 działa poprzez sekwestrację eIF4E, co w ten sposób hamuje jego asocjację z eIF4G i powoduje inaktywację HAV IRES (1). Inną metodą inaktywacji aktywności eIF4F jest działanie analogu kapu m7GpppG, który celuje w eIF4E, a następnie jest w stanie zapobiec jego połączeniu z zakończonymi czapeczką końcami 5' mRNA. Dokładny mechanizm, w którym ten analog kapu zakłóca IRES, nie jest jasny, ale sugeruje się, że wiązanie tego analogu z eIF4E powoduje zmianę konformacyjną eIF4G, co zakłóca normalną funkcję eIF4G.
dehydrogenaza gliceraldehydo-3-fosforanowa (GAPDH)
Dehydrogenaza aldehydu 3-fosforanowego gliceryny ( GAPDH ) jest enzymem komórkowym biorącym zazwyczaj udział w glikolizie . Wiadomo, że GAPDH wiąże się z nakładającymi się miejscami w obrębie pętli macierzystej IIIa w obrębie HAV IRES. Pętla-pętla IIIa zawiera delecję nukleotydu UU wewnątrz sekwencji 5 nukleotydów, która wzmacnia aktywność IRES. Skuteczne wiązanie GAPDH z tym regionem zdestabilizuje strukturę drugorzędową, którą tworzy IRES, hamując zdolność IRES do wykonywania translacji niezależnej od czapeczki.
La supresja białka
Białko komórki gospodarza powszechnie i wyłącznie w komórkach eukariotycznych, białko La wiąże się bezpośrednio z określonymi regionami HAV IRES podczas translacji mRNA, jak również replikacji RNA . W badaniu z 2008 roku zaobserwowano, że cytoplazmatyczny La zmniejsza inicjację HAV IRES. Jednak w 2014 roku nowsze badanie wykazało skuteczne hamowanie (in vivo) białka La jako proponowaną metodę hamowania translacji i replikacji HAV IRES, co oznacza, że najprawdopodobniej odgrywa ono integralną rolę w translacji i replikacji HAV.
Amantadyna
Amantadyna , tricykliczna amina symetryczna , jest sprawdzonym supresorem, który specyficznie hamuje zależną od HAV IRES translację HAV RNA. Eksperyment z 2005 roku wykazał, że amantadyna tłumiła translację HAV IRES i nie wyzwalała odpowiedzi na interferon, co wskazuje na obiecujące przeciwwirusowe zastosowanie amantadyny. Na wirusa grypy A , jego podstawową metodą działania jako środka przeciwwirusowego jest zapobieganie odsłanianiu genomu wirusa, co hamuje translację i replikację, w których pośredniczy HAV IRES. Skuteczność amantadyny wynika z lokalizacji IRES w regionie 5'NTR, który ma wysokie powinowactwo do leków przeciwwirusowych, co czyni ją skutecznym celem. Odkryto również, że białko M2 wirusa grypy typu A może być kolejnym realnym celem potencjalnego środka przeciwwirusowego.
Stwierdzono, że wszystkie pikornawirusy zawierają IRES. Istnieją cztery klasy IRES w picornaviridae , w zakresie od 270-450 nt. Wśród pikornawirusów wiele UTR 5' zawiera również dodatkowe elementy strukturalne w górę, które mogą wspomagać replikację genomu wirusa. Wiele pikornawirusów IRES pozwala również wielu wirusom blokować inicjację zależną od czapeczki, co skutkuje wyłączeniem syntezy białek komórki gospodarza. Cztery klasy to entero-/rinowirus IRES, kardio-/aftowirus IRES, HAV IRES, HCV-podobny pikornawirus IRES. Te IRES są podzielone na kategorie według ich sekwencji nukleotydowych, ale mają wspólne podobieństwo strukturalne, ponieważ to struktura RNA ma zdolność wewnętrznej rekrutacji maszynerii translacyjnej. Elementy entero-/rinowirusa IRES mają wspólne motywy strukturalne z HAV IRES. HAV IRES, entero-/rinowirus i kardio-/aptowirus IRES mają około 450 nt, ale znacznie różnią się strukturą. Kardiowirus, EMCV i aptowirus, wirus pryszczycy (FMDV) mają w około 50% identyczne elementy IRES. Pikornawirus podobny do HCV IRES zawiera największą różnicę w elementach IRES z pozostałych trzech klas. Istnieje wiele różnych picornaviridae , które mają wysoce konserwatywne elementy IRES podobne do HCV, z których przewiduje się, że niektóre z nich nadal będą identyfikowane. Należy zauważyć, że aktywność HAV IRES różni się od pozostałych trzech klas specyficznym wymogiem dla nienaruszonego eIF4G. Inne pikornawirusy kodują białka, które rozszczepią eIF4G w celu zwiększenia aktywności IRES.