BŁYSK
| Deweloperzy | Stevena Salzberga i Arthura Delchera |
|---|---|
| Wersja stabilna | 3.02 / 9 maja 2006
|
| Dostępne w | C++ |
| Typ | Narzędzie bioinformatyczne |
| Licencja | Oprogramowanie Open Source z certyfikatem OSI na licencji artystycznej |
| Strona internetowa |
W bioinformatyce GLIMMER (Gene Locator and Interpolated Markov ModelER) jest używany do znajdowania genów w prokariotycznym DNA . „Jest skuteczny w znajdowaniu genów w bakteriach , archeonach , wirusach , zwykle znajdując 98-99% wszystkich stosunkowo długich genów kodujących białka ”. GLIMMER był pierwszym systemem, który wykorzystał interpolowany model Markowa do identyfikacji regionów kodujących. Oprogramowanie GLIMMER jest open source i jest utrzymywane przez Stevena Salzberga , Arta Delchera i ich współpracowników z Centrum Biologii Obliczeniowej na Uniwersytecie Johnsa Hopkinsa . Oryginalne algorytmy i oprogramowanie GLIMMER zostały zaprojektowane przez Arta Delchera, Simona Kasifa i Stevena Salzberga i zastosowane do adnotacji genomu bakterii we współpracy z Owenem White'em .
Wersje
GLIMMER 1.0
Pierwsza wersja GLIMMER „tj. GLIMMER 1.0” została wydana w 1998 roku i została opublikowana w artykule Identyfikacja genów drobnoustrojów przy użyciu interpolowanego modelu Markowa . Modele Markowa wykorzystano do identyfikacji genów drobnoustrojów w GLIMMER 1.0. GLIMMER uwzględnia lokalne zależności sekwencji kompozycji, co czyni GLIMMER bardziej elastycznym i potężniejszym w porównaniu z modelem Markowa o stałym porządku .
Porównanie interpolowanego modelu Markowa zastosowanego w programie GLIMMER z modelem Markowa piątego rzędu dokonano w pracy Microbial gene identification using interpolated Markov models . „Algorytm GLIMMER znalazł 1680 genów z 1717 genów z adnotacjami u Haemophilus influenzae, gdzie model Markowa piątego rzędu znalazł 1574 geny. GLIMMER znalazł 209 dodatkowych genów, które nie zostały uwzględnione w 1717 genach z adnotacjami, gdzie model Markowa piątego rzędu znalazł 104 geny”.
GLIMMER 2.0
Druga wersja GLIMMERa, tj. GLIMMER 2.0 została wydana w 1999 roku i została opublikowana w artykule Udoskonalona identyfikacja drobnoustrojów za pomocą GLIMMER . Ten artykuł zawiera znaczące ulepszenia techniczne, takie jak użycie interpolowanego modelu kontekstu zamiast interpolowanego modelu Markowa i rozwiązanie nakładających się genów, co poprawia dokładność GLIMMER.
Interpolowane modele kontekstowe są używane zamiast interpolowanego modelu Markowa, co daje elastyczność wyboru dowolnej bazy. W interpolowanym modelu Markowa rozkład prawdopodobieństwa bazy wyznaczany jest na podstawie bezpośrednio poprzedzających baz. Jeśli bezpośrednio poprzedzająca zasada jest nieistotną aminokwasów , interpolowany model Markowa nadal uwzględnia poprzedzającą zasadę w celu określenia prawdopodobieństwa danej zasady, podczas gdy jako interpolowany model kontekstu, który był używany w GLIMMER 2.0, może ignorować nieistotne zasady. Fałszywie pozytywne prognozy zostały zwiększone w GLIMMER 2.0, aby zmniejszyć liczbę fałszywie negatywnych prognoz. Nakładające się geny są również rozwiązywane w GLIMMER 2.0.
Różne porównania między GLIMMER 1.0 i GLIMMER 2.0 zostały dokonane w artykule Ulepszona identyfikacja drobnoustrojów za pomocą GLIMMER , który pokazuje poprawę w późniejszej wersji. „Czułość GLIMMER 1.0 waha się od 98,4 do 99,7% ze średnią 99,1%, podczas gdy GLIMMER 2.0 ma zakres czułości od 98,6 do 99,8% ze średnią 99,3%. GLIMMER 2.0 jest bardzo skuteczny w znajdowaniu genów o dużej gęstości. pasożyt Trypanosoma brucei , odpowiedzialny za wywoływanie śpiączki afrykańskiej , jest identyfikowany przez GLIMMER 2.0"
GLIMMER 3.0
Trzecia wersja GLIMMER, „GLIMMER 3.0”, została wydana w 2007 roku i została opublikowana w artykule Identyfikacja genów bakteryjnych i DNA endosymbiontu za pomocą Glimmera . W tym artykule opisano kilka głównych zmian wprowadzonych w systemie GLIMMER, w tym ulepszone metody identyfikacji regionów kodujących i kodonów startowych . Ocena ORF w GLIMMER 3.0 odbywa się w odwrotnej kolejności, tj. zaczynając od kodonu stop i przesuwając się z powrotem w kierunku kodonu start. Skanowanie wsteczne pomaga dokładniej zidentyfikować kodującą część genu, która jest zawarta w oknie kontekstowym IMM. GLIMMER 3.0 poprawia również dane generowanego zestawu treningowego, porównując długą ORF z uniwersalnym rozkładem aminokwasów w bardzo różnych genomach bakteryjnych. -Wyjście ORF 39%.
GLIMMER 3.0 zmniejsza liczbę fałszywie pozytywnych prognoz, które zostały zwiększone w GLIMMER 2.0, aby zmniejszyć liczbę fałszywie negatywnych prognoz. „GLIMMER 3.0 ma dokładność przewidywania miejsca startu na poziomie 99,5% dla dopasowań 3'5', podczas gdy GLIMMER 2.0 ma 99,1% dla dopasowań 3'5'. GLIMMER 3.0 wykorzystuje nowy algorytm do skanowania regionów kodujących, nowy moduł wykrywania miejsca startu i architekturę, która integruje wszystkie przewidywania genów w całym genomie”.
Fundacja Teoretyczna i Biologiczna
Projekt GLIMMER pomógł wprowadzić i spopularyzować wykorzystanie modeli o zmiennej długości w biologii obliczeniowej i bioinformatyce, które następnie zastosowano do wielu problemów, takich jak klasyfikacja białek i inne. Modelowanie o zmiennej długości zostało pierwotnie zapoczątkowane przez teoretyków informacji, a następnie zostało pomysłowo zastosowane i spopularyzowane w kompresji danych (np. kompresji Ziva-Lempela). Przewidywanie i kompresja są ze sobą ściśle powiązane przy użyciu minimalnej długości opisu . Podstawową ideą jest stworzenie słownika wyrazów częstych (motywów w sekwencjach biologicznych). Intuicja mówi, że często pojawiające się motywy będą prawdopodobnie najbardziej predykcyjne i informacyjne. W GLIMMER model interpolowany jest modelem mieszanym prawdopodobieństw tych stosunkowo powszechnych motywów. Podobnie jak w przypadku rozwoju HMM w biologii obliczeniowej, na autorów projektu GLIMMER koncepcyjnie wpłynęło wcześniejsze zastosowanie innego wariantu interpolowanych modeli Markowa do rozpoznawania mowy przez badaczy takich jak Fred Jelinek (IBM) i Eric Ristad (Princeton). Algorytm uczenia się w GLIMMER różni się od tych wcześniejszych podejść.
Dostęp
GLIMMER można pobrać ze strony głównej The Glimmer (wymaga kompilatora C++ ). Alternatywnie, wersja online jest hostowana przez NCBI [1] .
Jak to działa
- GLIMMER wyszukuje przede wszystkim długie ORFS . Otwarta ramka odczytu może nakładać się na dowolną inną otwartą ramkę odczytu, która zostanie rozwiązana przy użyciu techniki opisanej w podsekcji. Korzystając z tych długich ORFS i podążając za określonym rozkładem aminokwasów, GLIMMER generuje dane zestawu treningowego .
- Korzystając z tych danych treningowych, GLIMMER trenuje wszystkie sześć modeli kodowania DNA Markowa od zera do ośmiu rzędów, a także trenuje model niekodującego DNA
- GLIMMER próbuje obliczyć prawdopodobieństwa na podstawie danych. Na podstawie liczby obserwacji GLIMMER określa, czy użyć modelu Markowa o stałym rzędzie , czy interpolowanego modelu Markowa.
- Jeśli liczba obserwacji jest większa niż 400, GLIMMER używa modelu Markowa o stałym rzędzie, aby uzyskać tam prawdopodobieństwa.
- Jeśli liczba obserwacji jest mniejsza niż 400, GLIMMER używa interpolowanego modelu Markowa, który jest pokrótce wyjaśniony w następnym podrozdziale.
- GLIMMER uzyskuje wynik dla każdego wygenerowanego długiego ORF przy użyciu wszystkich sześciu kodujących modeli DNA, a także przy użyciu niekodującego modelu DNA.
- Jeśli wynik uzyskany w poprzednim kroku jest większy niż określony próg, GLIMMER przewiduje, że jest to gen.
Kroki wyjaśnione powyżej opisują podstawową funkcjonalność GLIMMER. W GLIMMER wprowadzono różne ulepszenia, a niektóre z nich opisano w poniższych podsekcjach.
System GLIMMER
System GLIMMER składa się z dwóch programów. Pierwszy program o nazwie build-imm, który pobiera wejściowy zestaw sekwencji i wyprowadza interpolowany model Markowa w następujący sposób.
Obliczane jest prawdopodobieństwo dla każdej podstawy, tj. A,C,G,T dla wszystkich k-merów dla 0 ≤ k ≤ 8. Następnie dla każdego k-meru GLIMMER oblicza wagę. Prawdopodobieństwo nowej sekwencji jest obliczane w następujący sposób.
gdzie n to długość sekwencji to oligomer w pozycji x. 8 -order interpolowany wynik modelu Markowa jest obliczany
„gdzie jest wagą k -meru w pozycji x-1 w sekwencji S i jest oszacowaniem uzyskanym z danych treningowych prawdopodobieństwa położenia podstawy na pozycji x w modelu rzędu "
Prawdopodobieństwo podstawy i poprzednie podstawy, oblicza się w następujący sposób
Wartość związaną z x miara pewności co do dokładności tej wartości jako oszacowanie prawdziwego prawdopodobieństwa. GLIMMER wykorzystuje dwa kryteria do określenia. Pierwszym z nich jest częstotliwości, w którym liczba wystąpień ciągu kontekstu treningowych przekracza określoną wartość progową, a następnie jest ustawione na 1,0. Bieżąca domyślna wartość progu to 400, co daje 95% pewności. Gdy nie ma wystarczającej liczby przykładowych wystąpień łańcucha kontekstu, build-imm stosuje dodatkowe kryteria w celu określenia danego ciągu kontekstowego porównaj obserwowane częstotliwości następującej podstawy , fa , z wcześniej obliczonymi interpolowanymi prawdopodobieństwami modelu Markowa przy użyciu następnego krótszego kontekstu ja , , S . Używając , build-imm określ, jakie jest prawdopodobieństwo, że cztery zaobserwowane częstotliwości są zgodne z wartościami IMM z następnego krótszego
Drugi program o nazwie glimmer używa tego IMM do identyfikacji domniemanego genu w całym genomie. GLIMMER identyfikuje wszystkie otwarte ramki odczytu , które uzyskały wynik wyższy niż próg i sprawdza, czy geny nie nakładają się. Rozwiązywanie nakładających się genów wyjaśniono w następnym podrozdziale.
Równania i wyjaśnienia użytych powyżej terminów zaczerpnięto z artykułu „Identyfikacja genów drobnoustrojów za pomocą interpolowanych modeli Markowa
Rozwiązywanie nakładających się genów
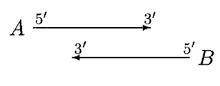
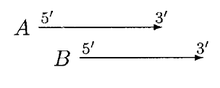
W GLIMMER 1.0, gdy dwa geny A i B zachodzą na siebie, punktowany jest obszar nakładania się. Jeśli A jest dłuższe niż B i jeśli A uzyska wyższy wynik w obszarze nakładania się, a przeniesienie miejsca początkowego B nie rozwiąże problemu nakładania się, wówczas B zostaje odrzucone.
GLIMMER 2.0 zapewnił lepsze rozwiązanie problemu nakładania się. W GLIMMER 2.0, gdy dwa potencjalne geny A i B nakładają się, punktowany jest region nakładania się. Załóżmy, że gen A ma wyższy wynik, rozważane są cztery różne orientacje.
W powyższym przypadku przeniesienie miejsc początkowych nie usuwa nakładania się. Jeśli A jest znacznie dłuższe niż B, to B jest odrzucane lub oba A i B nazywane są genami, z wątpliwym nakładaniem się.
W powyższym przypadku przesunięcie B może rozwiązać nakładanie się, A i B można nazwać genami nienakładającymi się, ale jeśli B jest znacznie krótszy niż A, to B jest odrzucane.
W powyższym przypadku przesunięcie A może rozwiązać nakładanie się. A jest przesuwane tylko wtedy, gdy nakładanie się jest niewielką częścią A, w przeciwnym razie B zostanie odrzucone.
W powyższym przypadku zarówno A, jak i B mogą zostać przesunięte. Najpierw przesuwamy początek B, aż region zachodzenia uzyska wyższy wynik dla B. Następnie przesuwamy początek A, dopóki nie uzyska wyższego wyniku. Następnie ponownie B i tak dalej, aż albo nakładanie się zostanie wyeliminowane, albo nie będzie można wykonać dalszych ruchów.
Powyższy przykład zaczerpnięto z artykułu „Identifying Bacterial Gens and Endosymbiont DNA with Glimmer”
Miejsca wiązania rybosomów
miejsca wiązania rybosomu (RBS) może być wykorzystany do znalezienia prawdziwej pozycji miejsca startu. Wyniki GLIMMER są przekazywane jako dane wejściowe do programu RBSfinder w celu przewidywania miejsc wiązania rybosomów. GLIMMER 3.0 integruje program RBSfinder z samą funkcją przewidywania genów.
Oprogramowanie ELPH (które w pracy określono jako wysoce skuteczne w identyfikacji RBS) jest używane do identyfikacji RBS i jest dostępne na tej stronie internetowej . Algorytm próbkowania Gibbsa służy do identyfikacji wspólnego motywu w dowolnym zestawie sekwencji. Te wspólne motywów i ich długość są podawane jako dane wejściowe do ELPH. Następnie ELPH oblicza macierz wagi pozycji (PWM), która zostanie wykorzystana przez GLIMMER 3 do oceny każdego potencjalnego RBS znalezionego przez RBSfinder. Powyższy proces ma miejsce, gdy mamy znaczną ilość genów treningowych. Jeśli liczba genów treningowych jest niewystarczająca, GLIMMER 3 może samodzielnie wygenerować zestaw prognoz genów, które można wykorzystać jako dane wejściowe do ELPH. ELPH oblicza teraz PWM i ten PWM może być ponownie użyty na tym samym zestawie genów, aby uzyskać dokładniejsze wyniki dla miejsc startowych. Ten proces można powtarzać dla wielu iteracji, aby uzyskać bardziej spójne wyniki PWM i przewidywania genów.
Wydajność
Glimmer wspiera wysiłki związane z adnotacją genomu w szerokim zakresie gatunków bakterii, archeonów i wirusów. W zakrojonym na dużą skalę wysiłku readnotacji w DNA Data Bank of Japan (DDBJ, który odzwierciedla Genbank ). Kosuge i in. (2006) zbadali metody znajdowania genów stosowane dla 183 genomów. Zgłosili, że spośród tych projektów Glimmer był wyszukiwarką genów w 49%, a następnie GeneMark w 12%, a inne algorytmy były używane w 3% lub mniej projektów. (Poinformowali również, że 33% genomów używało „innych” programów, co w wielu przypadkach oznaczało, że nie mogli zidentyfikować metody. Wyłączając te przypadki, Glimmer był używany w 73% genomów, dla których metody można było jednoznacznie zidentyfikować. ) Glimmer został użyty przez DDBJ do ponownego przypisania wszystkich genomów bakteryjnych w Międzynarodowych Bazach Danych Sekwencji Nukleotydów. Jest również używany przez tę grupę do opisywania wirusów. Glimmer jest częścią potoku adnotacji bakteryjnych w National Center for Biotechnology Information (NCBI), które utrzymuje również serwer sieciowy dla Glimmera, podobnie jak witryny w Niemczech i Kanadzie.
Według Google Scholar, od początku 2011 r. oryginalny artykuł Glimmera (Salzberg et al., 1998) był cytowany 581 razy, a artykuł Glimmer 2.0 (Delcher et al., 1999) był cytowany 950 razy.
Linki zewnętrzne
- Strona główna Glimmer w CCB, Johns Hopkins University , z której można pobrać oprogramowanie.




![{\displaystyle \operatorname {IMM_{k}(S_{x})=Y_{k}(S_{x-1})\cdot P_{k}(S_{x})+[1-{Y_{k}(S_{x-1})]\cdot IMM_{k-1}(S_{x})}} }](https://wikimedia.org/api/rest_v1/media/math/render/svg/2d303028692d57e04cae6e304ca63cd7db9b1181)



![{\displaystyle \operatorname {P_{i}(S_{x})=P(s_{x}|S_{x,j})={\frac {f(S_{x,j})}{\sum _{b\in {[acgt]}}\operatorname {f(S_{x,i},b)} }}} }](https://wikimedia.org/api/rest_v1/media/math/render/svg/0ed6988a29b7c1f3de8c00efa5f2bd50e895dc06)