Ekrany nokautowe CRISPR-Cas9 obejmujące cały genom
Ekrany nokautowe całego genomu CRISPR-Cas9 mają na celu wyjaśnienie związku między genotypem a fenotypem poprzez ablację ekspresji genów w skali całego genomu i badanie wynikających z tego zmian fenotypowych. Podejście to wykorzystuje CRISPR-Cas9 , połączony z bibliotekami pojedynczych przewodnich RNA (sgRNA) , które są zaprojektowane tak, aby celować w każdy gen w genomie. W ostatnich latach badanie przesiewowe CRISPR całego genomu stało się potężnym narzędziem do przeprowadzania badań przesiewowych utraty funkcji na dużą skalę, z niskim poziomem szumów, wysoką wydajnością nokautu i minimalnymi efektami poza celem.

Historia
We wczesnych badaniach nad Caenorhabditis elegans i Drosophila melanogaster przeprowadzono wielkoskalowe, systematyczne ekrany utraty funkcji (LOF) przeprowadzane za pomocą mutagenezy nasycającej , demonstrując potencjał tego podejścia do charakteryzowania szlaków genetycznych i identyfikowania genów o unikalnych i podstawowych funkcjach. Technikę mutagenezy nasycającej zastosowano później w innych organizmach, na przykład danio pręgowanego i myszach.
Ukierunkowane podejścia do eliminacji genów pojawiły się w latach 80. XX wieku wraz z technikami takimi jak rekombinacja homologiczna , trans-cięcie rybozymów i technologie antysensowne .
Do roku 2000 technologia interferencji RNA (RNAi) okazała się szybką, prostą i niedrogą techniką ukierunkowanego knockdownu genów i była rutynowo stosowana do badania funkcji genów in vivo u C. elegans . Rzeczywiście, w ciągu zaledwie kilku lat po jego odkryciu przez Fire et al . (1998), prawie wszystkie z około 19 000 genów C. elegans zostały przeanalizowane przy użyciu knockdown opartego na RNAi .
Produkcja bibliotek RNAi ułatwiła zastosowanie tej technologii na skalę całego genomu, a metody oparte na RNAi stały się dominującym podejściem do badań przesiewowych obejmujących cały genom. [ potrzebne źródło ]
Niemniej jednak podejścia oparte na RNAi do ekranów knockdown całego genomu mają swoje ograniczenia. Po pierwsze, wysokie efekty poza celem powodują problemy z fałszywie dodatnimi obserwacjami. Ponadto, ponieważ RNAi zmniejsza ekspresję genów na poziomie potranskrypcyjnym poprzez celowanie w RNA, badania przesiewowe oparte na RNAi skutkują jedynie częściową i krótkotrwałą supresją genów. Chociaż w niektórych sytuacjach częściowe powalenie może być pożądane, potrzebna była technologia o zwiększonej skuteczności celowania i mniejszej liczbie efektów poza celem. [ potrzebne źródło ]
Od początkowej identyfikacji jako prokariotyczny adaptacyjny układ odpornościowy, bakteryjny typ II skupiony w regularnych odstępach między krótkimi powtórzeniami palindromu (CRISPR) / system Cas9 stał się prostym i skutecznym narzędziem do generowania ukierunkowanych mutacji LOF. Został z powodzeniem zastosowany do edycji ludzkich genomów i zaczął wypierać RNAi jako dominujące narzędzie w badaniach ssaków. W kontekście ekranów z nokautem całego genomu, ostatnie badania wykazały, że ekrany CRISPR/Cas9 są w stanie osiągnąć wysoce wydajne i całkowite wyczerpanie białka oraz przezwyciężyć problemy poza celem obserwowane w przypadku ekranów RNAi. Podsumowując, niedawne pojawienie się CRISPR-Cas9 radykalnie zwiększyło naszą zdolność do wykonywania ekranów LOF na dużą skalę. Wszechstronność i programowalność Cas9, w połączeniu z niskim poziomem szumów, wysoką wydajnością nokautu i minimalnymi efektami poza celem, sprawiły, że CRISPR jest platformą wybieraną przez wielu badaczy zajmujących się celowaniem i edycją genów.
Metody
CRISPR/Cas9 Utrata funkcji
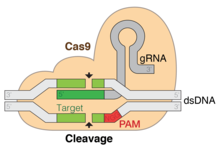
Skupiony , regularnie rozmieszczony system krótkich powtórzeń palindromu (CRISPR) / Cas9 to technologia edycji genów, która może wprowadzać pęknięcia dwuniciowe (DSB) w docelowym locus genomowym. Używając pojedynczego kierującego RNA (sgRNA) , endonukleaza Cas9 może zostać dostarczona do określonej sekwencji DNA, gdzie rozszczepia łańcuch nukleotydowy. Specyficzność sgRNA jest określona przez sekwencję 20-nt, homologiczną do interesującego locus genomowego, a wiązanie z Cas9 odbywa się za pośrednictwem stałego regionu rusztowania sgRNA. Po żądanym miejscu docelowym musi następować bezpośrednio (od 5' do 3') konserwatywny motyw sąsiadujący z protoprzerywnikiem o długości 3 nukleotydów (PAM). W celu naprawy DSB komórka może wykorzystywać wysoce podatne na błędy niehomologiczne łączenie końców lub rekombinację homologiczną . Projektując odpowiednie sgRNA, można wprowadzić do genomu planowane insercje lub delecje. W kontekście badań przesiewowych LOF obejmujących cały genom, celem jest spowodowanie przerwania i nokautu genów. [ potrzebne źródło ]
biblioteki sgRNA
Budowa Biblioteki
Aby przeprowadzić nokaut CRISPR na skalę całego genomu, należy wygenerować kolekcje sgRNA znane jako biblioteki sgRNA lub biblioteki nokautów CRISPR. Pierwszym krokiem w tworzeniu biblioteki sgRNA jest identyfikacja interesujących regionów genomowych w oparciu o znane zasady kierowania sgRNA. Na przykład sgRNA są najskuteczniejsze, gdy celują w regiony kodujące genów, a nie w 5' i 3' UTR . Konserwatywne egzony obecne jako atrakcyjne cele i należy wziąć pod uwagę pozycję względem miejsca startu transkrypcji. Po drugie, wszystkie możliwe miejsca PAM są identyfikowane i wybierane. Należy przeanalizować aktywność w miejscu docelowym i poza celem, podobnie jak zawartość GC i należy unikać rozciągania homopolimeru. Najczęściej stosowana endonukleaza Cas9, pochodząca ze Streptococcus pyogenes , rozpoznaje sekwencję PAM NGG.
Ponadto wydaje się, że określone nukleotydy są preferowane w określonych lokalizacjach. Guanina jest silnie uprzywilejowana w stosunku do cytozyny w pozycji 20 tuż obok motywu PAM, aw pozycji 16 cytozyna jest preferowana w stosunku do guaniny. Wykazano, że dla zmiennego nukleotydu w motywie NGG PAM cytozyna jest preferowana, a tymina nie. Biorąc pod uwagę takie kryteria, biblioteka sgRNA jest projektowana obliczeniowo wokół wybranych miejsc PAM.
Należy utworzyć wiele sgRNA (co najmniej 4–6) przeciwko każdemu pojedynczemu genowi, aby ograniczyć wykrywanie wyników fałszywie dodatnich, i należy uwzględnić sgRNA kontroli negatywnej bez znanych celów. SgRNA są następnie tworzone przez in situ , amplifikowane przez PCR i klonowane do systemu dostarczania wektorów.
Istniejące biblioteki
Opracowanie nowej biblioteki sgRNA jest pracochłonnym i czasochłonnym procesem. W praktyce badacze mogą wybrać istniejącą bibliotekę w zależności od celu eksperymentu i linii komórkowych będących przedmiotem zainteresowania. Od lutego 2020 r. Najczęściej używanymi zasobami do ekranów nokautowych CRISPR obejmujących cały genom były dwie CRISPR Knock-Out (GeCKO) w skali genomu utworzone przez laboratorium Zhang. Dostępne za pośrednictwem Addgene, te biblioteki lentiwirusowe celują odpowiednio w eksony ludzkie i mysie, i oba są dostępne jako system jednowektorowy (gdzie sgRNA i Cas9 są obecne na tym samym plazmidzie) lub jako system dwuwektorowy (gdzie sgRNA i Cas9 są obecne na oddzielnych plazmidach). Każda biblioteka jest dostarczana jako dwie półbiblioteki, co pozwala naukowcom na badanie przesiewowe za pomocą 3 lub 6 sgRNA/gen.
Oprócz GeCKO, za pośrednictwem Addgene wygenerowano i udostępniono szereg innych bibliotek CRISPR. Laboratoria Sabatini & Lander mają obecnie 7 oddzielnych bibliotek ludzkich i mysich, w tym ukierunkowane podbiblioteki dla różnych podpul, takich jak kinazy i geny rybosomalne (Addgene #51043–51048). Co więcej, ulepszenia specyficzności sgRNA zaowocowały bibliotekami „drugiej generacji”, takimi jak biblioteki Brie (Addgene #73632) i Brunello (Addgene #73178) wygenerowane przez laboratoria Doench i Root oraz biblioteka Toronto knockout (TKO) (Addgene # 1000000069) wygenerowany przez laboratorium Moffat.
Wektory lentiwirusowe
Ukierunkowany nokaut genu przy użyciu CRISPR/Cas9 wymaga zastosowania systemu dostarczania w celu wprowadzenia sgRNA i Cas9 do komórki. Chociaż potencjalnie dostępnych jest wiele różnych systemów dostarczania dla CRISPR, ekrany utraty funkcji całego genomu są głównie przeprowadzane przy użyciu wektorów lentiwirusowych trzeciej generacji. Te wektory lentiwirusowe są w stanie skutecznie transdukować szeroki zakres typów komórek i stabilnie integrować się z genomem dzielących się i niedzielących się komórek. Cząsteczki lentiwirusa trzeciej generacji są wytwarzane przez kotransfekcję ludzkich embrionalnych komórek nerki (HEK) 293T za pomocą:
- dwa plazmidy pakujące, jeden kodujący Rev , a drugi Gag i Pol ;
- wymienny plazmid otoczki , który koduje glikoproteinę otoczki innego wirusa (najczęściej białko G wirusa pęcherzykowego zapalenia jamy ustnej (VSV-G));
- jeden lub dwa (w zależności od zastosowanej biblioteki) plazmidy transferowe, kodujące Cas9 i sgRNA oraz markery selekcyjne.
Supernatant zawierający cząstki lentiwirusowe zbiera się, zatęża, a następnie używa do zakażenia komórek docelowych. Dokładny protokół produkcji lentiwirusa będzie się różnić w zależności od celu badawczego i zastosowanej biblioteki. Jeśli stosuje się na przykład system dwóch wektorów, komórki są sekwencyjnie transdukowane Cas9 i sgRNA w procedurze dwuetapowej. Chociaż jest to bardziej złożone, ma to zaletę w postaci wyższego miana wirusa biblioteki sgRNA.
Selekcja fenotypowa
Ogólnie rzecz biorąc, istnieją dwa różne formaty ekranów nokautowych CRISPR obejmujących cały genom: macierzowe i łączone. W ułożonym ekranie każda studzienka zawiera specyficzny i znany sgRNA ukierunkowany na określony gen. Ponieważ sgRNA odpowiedzialne za każdy fenotyp jest znane na podstawie lokalizacji studzienki, fenotypy można identyfikować i analizować bez konieczności sekwencjonowania genetycznego. Ten format pozwala na pomiar bardziej specyficznych fenotypów komórkowych, być może za pomocą fluorescencji lub luminescencji, i pozwala naukowcom na wykorzystanie większej liczby typów bibliotek i metod dostarczania. Jednak w przypadku ekranów LOF na dużą skalę formaty tablicowe są uważane za mało wydajne i kosztowne pod względem zasobów finansowych i materialnych, ponieważ populacje komórek muszą być izolowane i hodowane indywidualnie.
W zbiorczym badaniu przesiewowym komórki hodowane w jednym naczyniu transdukuje się masowo wektorami wirusowymi zawierającymi łącznie całą bibliotekę sgRNA. Aby upewnić się, że ilość komórek zainfekowanych przez więcej niż jedną cząsteczkę zawierającą sgRNA jest ograniczona, stosuje się niską krotność infekcji (MOI) (zwykle 0,3-0,6). Dotychczasowe dowody sugerują, że każdy sgRNA powinien być reprezentowany w co najmniej 200 komórkach. Transdukowane komórki zostaną poddane selekcji, po czym nastąpi selekcja pozytywna lub negatywna pod kątem fenotypu będącego przedmiotem zainteresowania, a sekwencjonowanie genetyczne będzie konieczne do zidentyfikowania zintegrowanych sgRNA.
Sekwencjonowanie i analiza trafień nowej generacji
Po selekcji fenotypowej genomowy DNA jest ekstrahowany z wybranych klonów wraz z kontrolną populacją komórek. W najpowszechniejszych protokołach nokautów całego genomu „biblioteka sekwencjonowania nowej generacji (NGS)” jest tworzona przez dwuetapową reakcję łańcuchową polimerazy (PCR). W pierwszym etapie amplifikuje się region sgRNA, stosując startery specyficzne dla sekwencji integracyjnej lentiwirusa, a w drugim etapie dodaje się sekwencje Illumina i5 i i7. NGS produktów PCR umożliwia identyfikację odzyskanych sgRNA, a do określenia względnej obfitości każdego sgRNA można zastosować etap oznaczania ilościowego.
Ostatnim krokiem na ekranie jest obliczeniowa ocena znacznie wzbogaconych lub zubożonych sgRNA, prześledzenie ich z powrotem do odpowiadających im genów, a następnie określenie, które geny i szlaki mogą być odpowiedzialne za obserwowany fenotyp. Obecnie dostępnych jest kilka algorytmów do tego celu, z których najbardziej popularna to oparta na modelach metoda CRISPR/Cas9 Knockout (MAGeCK) oparta na modelu. Opracowany specjalnie dla ekranów nokautowych CRISPR/Cas9 w 2014 r. MAGeCK wykazał lepszą wydajność w porównaniu z alternatywnymi algorytmami w tamtym czasie i od tego czasu wykazał solidne wyniki i wysoką czułość w różnych warunkach eksperymentalnych. Od 2015 roku algorytm MAGeCK został rozszerzony, aby wprowadzić pomiary kontroli jakości i uwzględnić pomijaną wcześniej skuteczność nokautu sgRNA. Zintegrowano również internetowe narzędzie do wizualizacji (VISPR), umożliwiające użytkownikom interaktywne przeglądanie wyników, analiz i kontroli jakości.
Aplikacje
Mechanizmy sygnalizacji komórkowej
W ostatnich latach ekran CRISPR obejmujący cały genom stał się potężnym narzędziem do badania skomplikowanych sieci sygnalizacji komórkowej. Sygnalizacja komórkowa jest niezbędna dla wielu podstawowych procesów biologicznych, w tym wzrostu komórek, proliferacji, różnicowania i apoptozy .
Jednym z praktycznych przykładów jest identyfikacja genów wymaganych do sygnalizacji proliferacyjnej w komórkach nowotworowych. Komórki transdukuje się biblioteką sgRNA CRISPR i bada pod kątem wzrostu w czasie. Porównując obfitość sgRNA w wybranych komórkach z kontrolą, można zidentyfikować, które sgRNA ulegają wyczerpaniu iz kolei, które geny mogą być odpowiedzialne za defekt proliferacji. Takie ekrany wykorzystano do identyfikacji genów istotnych dla raka w ostrej białaczce szpikowej i nerwiaku niedojrzałym oraz do opisania specyficznych dla nowotworu różnic między liniami komórek nowotworowych.
Identyfikacja syntetycznych śmiercionośnych partnerów
Ukierunkowane terapie przeciwnowotworowe mają na celu ukierunkowanie na określone geny, białka lub środowiska przyczyniające się do wzrostu lub przeżycia komórek nowotworowych. Jednak po okresie przedłużonego leczenia tymi terapiami komórki nowotworowe mogą rozwinąć oporność. Chociaż mechanizmy stojące za opornością na leki przeciwnowotworowe są słabo poznane, potencjalne przyczyny obejmują: zmianę celu, degradację leku, ucieczkę przed apoptozą i zmiany epigenetyczne. Oporność jest dobrze znana i stanowi poważny problem w leczeniu raka. [ potrzebne źródło ]
Aby przezwyciężyć ten problem, można zidentyfikować syntetycznego śmiercionośnego partnera. Ekrany LOF całego genomu przy użyciu CRISPR-Cas9 można wykorzystać do wyszukiwania syntetycznych śmiercionośnych partnerów. W tym celu linię komórkową typu dzikiego i linię komórkową nowotworu zawierającą mutację powodującą oporność transdukuje się biblioteką sgRNA CRISPR. Dwie linie komórkowe są hodowane, a wszelkie niedostatecznie reprezentowane lub martwe komórki są analizowane w celu zidentyfikowania potencjalnych syntetycznych śmiercionośnych genów partnerskich. Niedawne badanie przeprowadzone przez Hinze i in. (2019) wykorzystali tę metodę do zidentyfikowania syntetycznej śmiertelnej interakcji między lekiem stosowanym w chemioterapii asparaginazą a dwoma genami szlaku sygnałowego Wnt, NKD2 i LGR6.
Czynniki zależności gospodarza dla infekcji wirusowej
Ze względu na małe genomy i ograniczoną liczbę zakodowanych białek wirusy wykorzystują białka gospodarza do wejścia, replikacji i transmisji. Identyfikacja takich białek gospodarza, zwanych również czynnikami zależności od gospodarza (HDF), jest szczególnie ważna dla identyfikacji celów terapeutycznych. W ostatnich latach wiele grup z powodzeniem wykorzystało CRISPR/Cas9 całego genomu jako strategię przesiewową w kierunku HDF w infekcjach wirusowych.
Jeden przykład jest dostarczony przez Marceau i in. (2017), których celem było przeanalizowanie czynników gospodarza związanych z zakażeniem dengą i zapaleniem wątroby typu C (HCV) (dwa wirusy z rodziny Flaviviridae ). Stwierdzono, że ELAVL1 , białko wiążące RNA kodowane przez gen ELAVL1, jest krytycznym receptorem dla wejścia HCV i wykazano niezwykłą rozbieżność czynników zależności od gospodarza między dwoma flawiwirusami.
Dalsze zastosowania
Dodatkowe zgłoszone zastosowania ekranów CRISPR całego genomu obejmują badanie: metabolizmu mitochondriów, oporności na toksyny bakteryjne, genetycznych czynników powodujących przerzuty, oporności na leki przeciwnowotworowe, śmierci komórkowej wywołanej wirusem Zachodniego Nilu i sieci genów komórek odpornościowych.
Ograniczenia
Ta sekcja dotyczy w szczególności ekranów CRISPR obejmujących cały genom. Aby zapoznać się z przeglądem ograniczeń CRISPR, zob. Lino i in. (2018)
Biblioteka sgRNA
Ekrany CRISPR całego genomu będą ostatecznie ograniczone właściwościami wybranej biblioteki sgRNA. Każda biblioteka będzie zawierać inny zestaw sgRNA, a średni zasięg na gen może się różnić. Obecnie dostępne biblioteki są raczej ukierunkowane na sgRNA ukierunkowane na wczesne (5') eksony kodujące białka, niż na te ukierunkowane na bardziej funkcjonalne domeny białkowe. Problem ten został podkreślony przez Hinze i in. (2019), którzy zauważyli, że geny związane z wrażliwością na asparaginazę nie uzyskały punktów w całym genomie przesiewowym komórek białaczki opornych na asparaginazę.
Jeśli odpowiednia biblioteka nie jest dostępna, tworzenie i amplifikacja nowej biblioteki sgRNA jest procesem długotrwałym, który może zająć wiele miesięcy. Potencjalne wyzwania obejmują: (i) efektywny projekt sgRNA; (ii) zapewnienie kompleksowego pokrycia sgRNA w całym genomie; (iii) projekt szkieletu wektora lentiwirusowego; (iv) wytwarzanie wystarczających ilości wysokiej jakości lentiwirusa; (v) przezwyciężenie niskiej wydajności transformacji; (vi) właściwe skalowanie hodowli bakteryjnej.
Utrzymanie komórkowego pokrycia sgRNA
Jedną z największych przeszkód dla badań przesiewowych CRISPR całego genomu jest zapewnienie odpowiedniego pokrycia biblioteki sgRNA w całej populacji komórek. Dotychczasowe dowody sugerują, że każdy sgRNA powinien być reprezentowany i utrzymywany w co najmniej 200-300 komórkach.
Biorąc pod uwagę, że standardowy protokół wykorzystuje krotność infekcji ~0,3, a wydajność transdukcji 30-40%, liczba komórek wymaganych do wytworzenia i utrzymania odpowiedniego pokrycia staje się bardzo duża. Na przykład najpopularniejszą biblioteką ludzkiego sgRNA jest biblioteka GeCKO v2 stworzona przez laboratorium Zhang; zawiera 123 411 sgRNA. Badania wykorzystujące tę bibliotekę zwykle transdukują więcej niż 1x108 komórek
Ponieważ CRISPR nadal wykazuje niski poziom szumów i minimalne efekty poza celem, alternatywną strategią jest zmniejszenie liczby sgRNA na gen dla głównego ekranu przesiewowego. Do selekcji trafień stosuje się mniej rygorystyczne wartości odcięcia, a dodatkowe sgRNA są później wykorzystywane w bardziej szczegółowym badaniu wtórnym. To podejście zostało zademonstrowane przez Doench i in . (2016), którzy stwierdzili, że >92% genów odzyskanych przy użyciu standardowego protokołu zostało również odzyskanych przy użyciu mniejszej liczby sgRNA na gen. Sugerują, że ta strategia może być przydatna w badaniach, w których zwiększanie skali jest zbyt kosztowne. [ potrzebne źródło ]
Ograniczenia lentiwirusowe
Wektory lentiwirusowe mają pewne ogólne ograniczenia. Po pierwsze, niemożliwe jest kontrolowanie, gdzie genom wirusa integruje się z genomem gospodarza, a to może wpływać na ważne funkcje komórki. Vannucci i in. zapewniają doskonały przegląd wektorów wirusowych wraz z ich ogólnymi zaletami i wadami. W specyficznym kontekście badań przesiewowych CRISPR obejmujących cały genom, wytwarzanie i transdukcja cząstek lentiwirusowych jest stosunkowo pracochłonne i czasochłonne, łącznie zajmuje około dwóch tygodni. Dodatkowo, ponieważ DNA integruje się z genomem gospodarza, dostarczanie lentiwirusowe prowadzi do długotrwałej ekspresji Cas9, potencjalnie prowadząc do efektów poza celem. [ potrzebne źródło ]
Ekrany w tablicy a ekrany w puli
W ułożonym ekranie każda studzienka zawiera specyficzny i znany sgRNA ukierunkowany na określony gen. Ekrany z macierzami umożliwiają zatem szczegółowe profilowanie pojedynczej komórki, ale są ograniczone wysokimi kosztami i pracochłonnością wymaganą do wyizolowania i hodowli dużej liczby pojedynczych populacji komórek. Konwencjonalne połączone ekrany CRISPR są stosunkowo proste i opłacalne w wykonaniu, ale ograniczają się do badania całej populacji komórek. Oznacza to, że rzadkie fenotypy mogą być trudniejsze do zidentyfikowania i tylko surowe fenotypy mogą być selekcjonowane pod kątem np. przeżycia komórek, proliferacji lub ekspresji genu reporterowego. [ potrzebne źródło ]
Media kulturowe
Wybór pożywki hodowlanej może mieć wpływ na fizjologiczną istotność wyników eksperymentów z hodowlami komórkowymi ze względu na różnice w składzie i stężeniach składników odżywczych. Ostatnio wykazano systematyczne odchylenie w generowanych zestawach danych dla wyciszania genów CRISPR i RNAi (szczególnie dla genów metabolicznych) oraz dla profilowania metabolicznego linii komórek nowotworowych . Na przykład stwierdzono silniejszą zależność od ASNS (syntetazy asparaginy) w liniach komórkowych hodowanych w DMEM , w którym brakuje asparaginy, w porównaniu z liniami komórkowymi hodowanymi w RPMI lub F12 (zawierający asparaginę). Uniknięcie takiego odchylenia można osiągnąć, stosując jednolitą pożywkę dla wszystkich badanych linii komórkowych, a najlepiej, stosując pożywkę wzrostową , która lepiej odzwierciedla fizjologiczne poziomy składników odżywczych. Ostatnio opracowano takie typy pożywek, jak Plasmax i Human Plasma Like Medium (HPLM).
Przyszłe kierunki
CRISPR + pojedyncza komórka RNA-sekw
Powstające technologie mają na celu połączenie połączonych ekranów CRISPR ze szczegółową rozdzielczością masowo równoległego sekwencjonowania RNA pojedynczych komórek (RNA-seq) . Badania wykorzystujące „CRISP-seq”, „CROP-seq” i „PERTURB-seq” wykazały bogate odczyty genomowe, dokładnie identyfikując sygnatury ekspresji genów dla poszczególnych nokautów genów w złożonej puli komórek. Metody te mają dodatkową zaletę tworzenia profili transkrypcyjnych komórek indukowanych sgRNA.