Pirosekwencjonowanie
Pirosekwencjonowanie to metoda sekwencjonowania DNA (określania kolejności nukleotydów w DNA) oparta na zasadzie „sekwencjonowania przez syntezę”, w której sekwencjonowanie odbywa się poprzez wykrywanie nukleotydu wbudowanego przez polimerazę DNA . Pirosekwencjonowanie polega na wykrywaniu światła w oparciu o reakcję łańcuchową uwalniania pirofosforanu . Stąd nazwa pirosekwencjonowanie.
Zasada pirosekwencjonowania została po raz pierwszy opisana w 1993 roku przez Bertila Petterssona, Mathiasa Uhlena i Påla Nyrena poprzez połączenie metody sekwencjonowania w fazie stałej przy użyciu kulek magnetycznych pokrytych streptawidyną z rekombinowaną polimerazą DNA pozbawioną aktywności egzonukleazy 3´do 5´ (korekta) i luminescencji wykrywanie za pomocą enzymu lucyferazy świetlika . Mieszanina trzech enzymów ( polimerazy DNA , sulfurylazy ATP i lucyferazy świetlika ) oraz nukleotydu ( dNTP ) dodaje się do jednoniciowego DNA w celu sekwencjonowania, a po włączeniu nukleotydu dokonuje się pomiaru emitowanego światła. Intensywność światła określa, czy włączono 0, 1 lub więcej nukleotydów, pokazując w ten sposób, ile komplementarnych nukleotydów jest obecnych na nici matrycy. Mieszaninę nukleotydów usuwa się przed dodaniem następnej mieszaniny nukleotydów. Ten proces jest powtarzany dla każdego z czterech nukleotydów, aż do określenia sekwencji DNA jednoniciowej matrycy.
Druga metoda pirosekwencjonowania oparta na roztworach została opisana w 1998 roku przez Mostafę Ronaghi , Mathiasa Uhlena i Påla Nyrena . W tej alternatywnej metodzie wprowadza się dodatkowy enzym apirazę w celu usunięcia nukleotydów, które nie są włączane przez polimerazę DNA. Umożliwiło to powstanie mieszaniny enzymów obejmującej polimerazę DNA , lucyferazę i apirazę należy dodać na początku i zachować przez cały czas trwania procedury, zapewniając w ten sposób prostą konfigurację odpowiednią do automatyzacji. Zautomatyzowany instrument oparty na tej zasadzie został wprowadzony na rynek w następnym roku przez firmę Pyrosequencing.
Trzeci mikroprzepływowy wariant metody pirosekwencjonowania został opisany w 2005 roku przez Jonathana Rothberga i współpracowników firmy 454 Life Sciences . To alternatywne podejście do pirosekwencjonowania opierało się na pierwotnej zasadzie przyłączania DNA przeznaczonego do sekwencjonowania do stałego podłoża i wykazało, że sekwencjonowanie można przeprowadzić w wysoce równoległy sposób przy użyciu mikromacierzy mikrofabrykowanej . Pozwoliło to na wysokowydajne sekwencjonowanie DNA, a na rynek wprowadzono zautomatyzowane urządzenie. Stało się to pierwszym instrumentem do sekwencjonowania nowej generacji, rozpoczynającym nową erę genomicznych , przy gwałtownie spadających cenach sekwencjonowania DNA, co umożliwia sekwencjonowanie całego genomu po przystępnych cenach.
Procedura
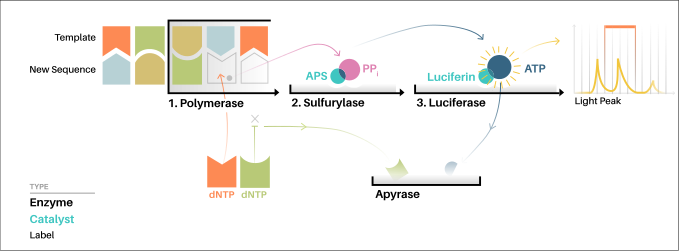
„Sekwencjonowanie przez syntezę” obejmuje pobranie pojedynczej nici DNA do sekwencjonowania, a następnie enzymatyczną syntezę jej komplementarnej nici. Metoda pirosekwencjonowania polega na wykrywaniu aktywności polimerazy DNA (enzymu syntetyzującego DNA) z innym enzymem chemoluminescencyjnym . Zasadniczo metoda umożliwia sekwencjonowanie pojedynczej nici DNA poprzez syntezę komplementarnej nici wzdłuż niej, po jednej parze zasad i wykrywanie, która zasada została faktycznie dodana na każdym etapie. Matryca DNA jest nieruchoma, a roztwory nukleotydów A, C, G i T są kolejno dodawane i usuwane z reakcji. Światło jest wytwarzane tylko wtedy, gdy roztwór nukleotydu uzupełnia pierwszą niesparowaną zasadę matrycy. Sekwencja roztworów, które wytwarzają sygnały chemiluminescencyjne, pozwala na określenie sekwencji matrycy.
W wersji pirosekwencjonowania opartej na roztworze, matryca jednoniciowego DNA ( ssDNA ) jest hybrydyzowana ze starterem do sekwencjonowania i inkubowana z enzymami polimerazy DNA , sulfurylazy ATP , lucyferazy i apyrazy oraz z substratami 5´-fosfosiarczanem adenozyny (APS) i lucyferyna .
- Dodanie jednego z czterech trójfosforanów dezoksynukleotydów ( dNTP ) (dATPαS, który nie jest substratem dla lucyferazy, dodaje się zamiast dATP, aby uniknąć szumu) inicjuje drugi etap. Polimeraza DNA włącza prawidłowe, komplementarne dNTP do matrycy. To włączenie uwalnia pirofosforan (PPi).
- Sulurylaza ATP przekształca PPi w ATP w obecności 5´fosfosiarczanu adenozyny. Ten ATP działa jako substrat dla pośredniczonej przez lucyferazę konwersji lucyferyny do oksylucyferyny, która generuje światło widzialne w ilościach proporcjonalnych do ilości. Światło wytwarzane w reakcji katalizowanej lucyferazą jest wykrywane przez kamerę i analizowane w programie.
- Niewbudowane nukleotydy i ATP są rozkładane przez apyrazę , a reakcja może rozpocząć się ponownie z innym nukleotydem.
Proces ten można przedstawić za pomocą następujących równań:
- PPi + APS → ATP + Siarczan (katalizowany przez ATP-sulfurylazę);
- ATP + lucyferyna + O2 → AMP + PPi + oksylucyferyna + CO2 + hv (katalizowana przez lucyferazę);
Gdzie:
- PPi to pirofosforan
- APS to 5-fosforan adenozyny;
- ATP to trójfosforan adenozyny;
- O2 to cząsteczka tlenu;
- AMP to monofosforan adenozyny;
- CO2 to dwutlenek węgla;
- hv jest lekki.
Ograniczenia
Obecnie ograniczeniem tej metody jest to, że długości poszczególnych odczytów sekwencji DNA mieszczą się w okolicach 300-500 nukleotydów, czyli są krótsze niż 800-1000 uzyskiwane metodami terminacji łańcucha (np. sekwencjonowanie Sangera). Może to utrudnić proces składania genomu , szczególnie w przypadku sekwencji zawierających dużą ilość powtarzalnego DNA . Brak działań korekcyjnych ogranicza dokładność tej metody.
Komercjalizacja
Firma Pyrosequencing AB w Uppsali w Szwecji została założona dzięki kapitałowi podwyższonego ryzyka zapewnionemu przez HealthCap w celu komercjalizacji maszyn i odczynników do sekwencjonowania krótkich odcinków DNA przy użyciu techniki pirosekwencjonowania. Pyrosequencing AB jest notowana na Giełdzie Papierów Wartościowych w Sztokholmie w 1999 r. W 2003 r. Zmieniono jej nazwę na Biotage. Linia biznesowa pirosekwencjonowania została przejęta przez Qiagen w 2008 r. Licencję na technologię pirosekwencjonowania otrzymało 454 Life Sciences . 454 opracował technologię pirosekwencjonowania opartą na macierzach, która pojawiła się jako platforma do sekwencjonowania DNA na dużą skalę , w tym sekwencjonowania genomu i metagenomiki .
Roche ogłosił wycofanie platformy sekwencjonowania 454 w 2013 roku.
Dalsza lektura
- Metzker M. (2005). „Nowe technologie w sekwencjonowaniu DNA” . Badania genomu . 15 (12): 1767–76. doi : 10.1101/gr.3770505 . PMID 16339375 .