Wielokrotne wzmocnienie przemieszczenia
Amplifikacja wielokrotnego przemieszczenia (MDA) to technika amplifikacji DNA . Ta metoda może szybko amplifikować niewielkie ilości próbek DNA do rozsądnej ilości do analizy genomicznej. Reakcja rozpoczyna się od przyłączenia losowych starterów heksamerowych do matrycy: synteza DNA jest przeprowadzana przez enzym o wysokiej wierności , preferencyjnie polimerazę DNA Φ29 . W porównaniu z konwencjonalnym PCR technik amplifikacji, MDA nie wykorzystuje starterów specyficznych dla sekwencji, ale amplifikuje całe DNA, generuje produkty o większej wielkości z mniejszą częstotliwością błędów i działa w stałej temperaturze. MDA jest aktywnie wykorzystywana w amplifikacji całego genomu (WGA) i jest obiecującą metodą do zastosowania w sekwencjonowaniu genomu pojedynczej komórki i badaniach genetycznych opartych na sekwencjonowaniu.
Tło
Wiele przypadków biologicznych i kryminalistycznych obejmujących analizę genetyczną wymaga sekwencjonowania DNA z niewielkich ilości próbek, takich jak DNA z niehodowanych pojedynczych komórek lub śladowych ilości tkanek pobranych z miejsc zbrodni. Konwencjonalne metody amplifikacji DNA oparte na reakcji łańcuchowej polimerazy ( PCR ) wymagają specyficznych dla sekwencji starterów oligonukleotydowych i termostabilnej (zwykle Taq ) polimerazy i może być wykorzystany do wygenerowania znacznych ilości DNA z niewielkich ilości DNA. Nie jest to jednak wystarczające dla nowoczesnych technik wykorzystujących analizę DNA opartą na sekwencjonowaniu. Dlatego konieczna jest bardziej wydajna, niespecyficzna dla sekwencji metoda amplifikacji niewielkich ilości DNA, zwłaszcza w badaniach genomicznych pojedynczych komórek.
Materiały
polimeraza DNA Phi 29
Polimeraza DNA bakteriofaga Φ29 jest enzymem o wysokiej procesowości , który może wytwarzać amplikony DNA większe niż 70 kilopar zasad. Jej wysoka wierność i aktywność korygująca 3'–5' zmniejszają współczynnik błędów amplifikacji do 1 na 10 6-10 7 zasad w porównaniu z konwencjonalną polimerazą Taq z raportowanym poziomem błędów 1 na 9000. Reakcję można przeprowadzić w umiarkowanych warunkach izotermicznych 30°C i dlatego nie wymaga termocyklera . Był aktywnie wykorzystywany w klonowaniu bezkomórkowym, czyli enzymatycznej metodzie amplifikacji DNA in vitro bez hodowli komórkowej i ekstrakcji DNA . Duży fragment Bst jest również używany w MDA, ale generalnie preferowany jest Ф29 ze względu na wystarczającą wydajność produktu i aktywność korekty.
Podkłady heksamerowe
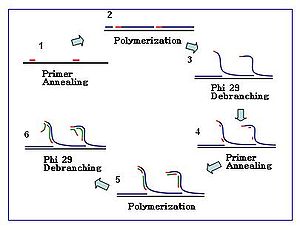
Startery heksamerowe to sekwencje złożone z sześciu przypadkowych nukleotydów . W zastosowaniach MDA startery te są zwykle modyfikowane tiofosforanem na swoim końcu 3 'w celu przekazania odporności na egzonukleazy 3' – 5' polimerazy DNA Ф29 . Reakcje MDA rozpoczynają się od przyłączania takich starterów do matrycy DNA, po którym następuje wydłużanie łańcucha za pośrednictwem polimerazy. Wzdłuż reakcji amplifikacji zachodzi coraz większa liczba zdarzeń przyłączania starterów.
Reakcja
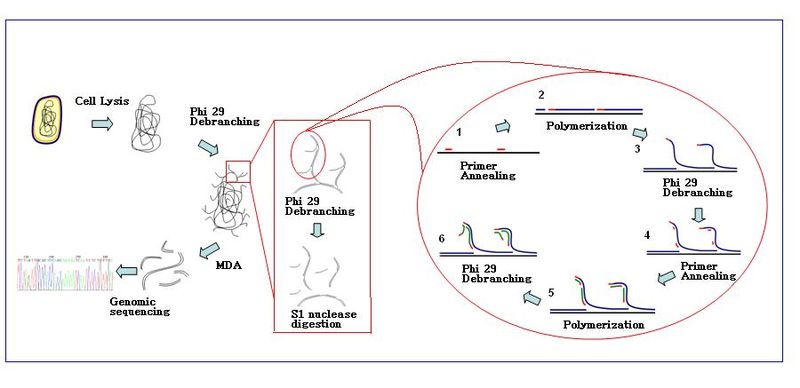
Reakcja amplifikacji rozpoczyna się, gdy wiele heksamerów starterów łączy się z matrycą. Kiedy synteza DNA przechodzi do następnego miejsca startowego, polimeraza wypiera nowo utworzoną nić DNA i kontynuuje jej wydłużanie. Przemieszczenie nici generuje nowo zsyntetyzowany szablon jednoniciowego DNA dla większej liczby starterów do hybrydyzacji. Dalsze wygrzewanie starterów i przemieszczanie nici na nowo zsyntetyzowanej matrycy skutkuje hiper-rozgałęzioną siecią DNA. Usuwanie rozgałęzień sekwencji podczas amplifikacji skutkuje wysoką wydajnością produktów. Aby oddzielić sieć rozgałęzień DNA, nukleazy S1 są używane do rozszczepiania fragmentów w miejscach przemieszczenia. Nacięcia na powstałych fragmentach DNA są naprawiane przez polimerazę DNA I.
Jakość produktu
MDA może wygenerować 1–2 µg DNA z pojedynczej komórki z pokryciem genomu do 99%. Produkty mają również niższy wskaźnik błędów i większe rozmiary w porównaniu z amplifikacją Taq opartą na PCR.
Ogólny przebieg pracy MDA:
- Przygotowanie próbki : Próbki są zbierane i rozcieńczane w odpowiednim buforze reakcyjnym (bez Ca 2+ i Mg 2+ ). Komórki poddaje się lizie buforem alkalicznym .
- Warunek : Reakcję MDA z polimerazą Ф29 przeprowadza się w temperaturze 30°C. Reakcja trwa zwykle około 2,5–3 godzin.
- Koniec reakcji : Inaktywować enzymy w temperaturze 65°C przed pobraniem zamplifikowanych produktów DNA
- Produkty DNA można oczyszczać za pomocą komercyjnego zestawu do oczyszczania.
Zalety
MDA generuje wystarczającą wydajność produktów DNA. Jest to potężne narzędzie do amplifikacji cząsteczek DNA z próbek, takich jak niehodowane mikroorganizmy lub pojedyncze komórki, do ilości wystarczającej do sekwencjonowania . Duży rozmiar produktów DNA amplifikowanych MDA zapewnia również pożądaną jakość próbki do identyfikacji wielkości polimorficznych alleli powtórzeń. Jego wysoka wierność sprawia również, że można go stosować w polimorfizmie pojedynczego nukleotydu (SNP) wykrywanie alleli. Ze względu na przemieszczenie nici podczas amplifikacji, amplifikowany DNA ma wystarczające pokrycie źródłowych cząsteczek DNA, co zapewnia wysokiej jakości produkt do analizy genomicznej. Produkty przemieszczonych nici można następnie klonować do wektorów w celu skonstruowania biblioteki do kolejnych sekwencjonowania .
Ograniczenia
Porzucenie alleli (ADO)
ADO definiuje się jako losowy brak amplifikacji jednego z alleli obecnych w próbce heterozygotycznej . Niektóre badania wykazały, że wskaźnik ADO produktów MDA wynosi 0–60%. Ta wada zmniejsza dokładność genotypowania pojedynczej próbki i błędną diagnozę w innych zastosowaniach związanych z MDA. Wydaje się, że ADO jest niezależne od wielkości fragmentów i odnotowano, że ma podobną częstość w innych technikach jednokomórkowych. Możliwe rozwiązania to zastosowanie różnych warunków lizy lub przeprowadzenie wielu rund amplifikacji z rozcieńczonych produktów MDA od PCR doniesiono, że amplifikacja za pośrednictwem hodowanych komórek daje niższe wskaźniki ADO.
Wzmocnienie preferencyjne
„Amplifikacja preferencyjna” to nadmierna amplifikacja jednego z alleli w porównaniu z drugim. Większość badań dotyczących MDA dotyczyła tego problemu. Obecnie obserwuje się, że błąd amplifikacji jest losowy. Może to wpływać na analizę małych odcinków genomowego DNA w identyfikacji alleli krótkich powtórzeń tandemowych (STR).
Interakcje elementarz-elementarz
Endogenna, niezależna od szablonu interakcja primer-primer wynika z losowego projektu starterów heksamerowych. Jednym z możliwych rozwiązań jest zaprojektowanie heksanukleotydowych starterów z randomizacją, które nie ulegają hybrydyzacji krzyżowej.
Aplikacje
Sekwencjonowanie genomu pojedynczej komórki
Za pomocą MDA zsekwencjonowano pojedyncze komórki niehodowanych bakterii, archeonów i protistów, a także pojedyncze cząsteczki wirusa i pojedyncze zarodniki grzybów .
Zdolność do sekwencjonowania poszczególnych komórek jest również przydatna w zwalczaniu chorób człowieka. Genomy z pojedynczych ludzkich komórek embrionalnych zostały pomyślnie zamplifikowane w celu sekwencjonowania przy użyciu MDA, umożliwiając preimplantacyjną diagnostykę genetyczną (PGD): badania przesiewowe pod kątem genetycznych problemów zdrowotnych we wczesnym stadium zarodka przed implantacją . Choroby o heterogennych właściwościach, takie jak rak , również korzystają ze zdolności sekwencjonowania genomu opartego na MDA do badania mutacji w poszczególnych komórkach.
Produkty MDA z pojedynczej komórki były również z powodzeniem stosowane w eksperymentach hybrydyzacji genomowej z porównawczą macierzą , które zwykle wymagają stosunkowo dużej ilości zamplifikowanego DNA.
Immunoprecypitacja chromatyny
Immunoprecypitacja chromatyny powoduje wytwarzanie złożonych mieszanin stosunkowo krótkich fragmentów DNA, których amplifikacja za pomocą MDA jest trudna bez powodowania błędu w reprezentacji fragmentów. Zaproponowano metodę obejścia tego problemu, która opiera się na konwersji tych mieszanin do kolistych konkatamerów za pomocą ligacji, a następnie MDA za pośrednictwem polimerazy DNA Φ29.
Analiza kryminalistyczna
Śladowe ilości próbek pobranych z miejsc zbrodni mogą zostać zwielokrotnione przez MDA do ilości wystarczającej do kryminalistycznej analizy DNA, która jest powszechnie stosowana do identyfikacji ofiar i podejrzanych.
Zobacz też
- Reakcja łańcuchowa polimerazy
- Amplifikacja całego genomu