Chip do immunoprecypitacji RNA
RIP-chip (RNA immunoprecipitation chip) to technika biologii molekularnej , która łączy immunoprecypitację RNA z mikromacierzą . Celem tej techniki jest określenie, które RNA oddziałują in vivo z konkretnym białkiem wiążącym RNA będącym przedmiotem zainteresowania . Można go również wykorzystać do określenia względnych poziomów ekspresji genów , aby zidentyfikować podzbiory RNA, które mogą być współregulowane, lub zidentyfikować RNA, które mogą mieć powiązane funkcje. Technika ta zapewnia wgląd w potranskrypcyjną regulację genów zachodzącą między RNA a białkami wiążącymi RNA.
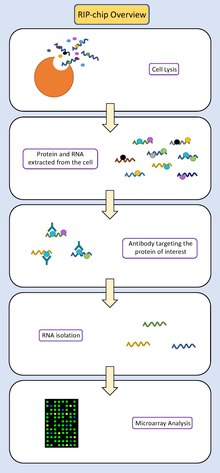
Przegląd procedur
- Zbierz i poddaj lizie interesujące Cię komórki .
- Wyizolować z roztworu wszystkie fragmenty RNA i związane z nimi białka.
- Immunoprecypituj białko będące przedmiotem zainteresowania. Roztwór zawierający RNA związane z białkami przemywa się przez kulki skoniugowane z przeciwciałami . Te przeciwciała są zaprojektowane tak, aby wiązać się z białkiem będącym przedmiotem zainteresowania. Wyciągają białko (i wszelkie fragmenty RNA, które są z nim specyficznie związane) z roztworu zawierającego resztę zawartości komórki.
- Oddzielić RNA związany z białkiem od kompleksu przeciwciało-kulki . Następnie użyj wirówki do oddzielenia RNA związanego z białkiem od cięższych kompleksów przeciwciało-kuleczki, zachowując RNA związany z białkiem i odrzucając kulki.
- Oddziel RNA od białka będącego przedmiotem zainteresowania .
- Wyizolować fragmenty RNA z białka za pomocą wirówki.
- Użyj metody PCR z odwrotną transkrypcją , aby przekształcić fragmenty RNA w cDNA (DNA, które jest komplementarne do fragmentów RNA).
- Oznacz fluorescencyjnie te fragmenty cDNA.
- Przygotuj chip genowy . To mały czip, z którym są związane sekwencje DNA w znanych miejscach. Te sekwencje DNA odpowiadają wszystkim znanym genom w genomie organizmu, z którym pracuje badacz (lub podzbiorowi genów, którymi interesuje się badacz). Zebrane sekwencje cDNA będą komplementarne do niektórych z tych sekwencji DNA, ponieważ cDNA reprezentują podzbiór RNA transkrybowanych z genomu.
- Pozwól fragmentom cDNA na konkurencyjną hybrydyzację z sekwencjami DNA związanymi z chipem .
- Wykrywanie sygnału fluorescencyjnego z cDNA związanego z chipem mówi naukowcom, które geny na chipie uległy hybrydyzacji z cDNA.
Geny zidentyfikowane fluorescencyjnie za pomocą analizy chipów to geny, których RNA oddziałuje z oryginalnym białkiem będącym przedmiotem zainteresowania. Siła sygnału fluorescencyjnego dla określonego genu może wskazywać, ile tego konkretnego RNA było obecne w oryginalnej próbce, co wskazuje na poziom ekspresji tego genu.
Rozwój i podobne techniki
testy zmiany mobilności elektroforetycznej RNA i sieciowanie UV, a następnie RT-PCR, jednak taka selektywna analiza nie może być stosowana, gdy związane RNA nie są jeszcze znane. Aby rozwiązać ten problem, układ RIP łączy immunoprecypitację RNA w celu wyizolowania cząsteczek RNA wchodzących w interakcje z określonymi białkami za pomocą mikromacierzy, która może wyjaśnić tożsamość RNA uczestniczących w tej interakcji. Alternatywy dla chipów RIP obejmują:
- RIP-seq : obejmuje sekwencjonowanie RNA, które zostały ściągnięte przy użyciu sekwencjonowania o wysokiej przepustowości, zamiast analizować je za pomocą mikromacierzy. Autorzy Zhao i in., 2010. połączyli procedurę immunoprecypitacji RNA z sekwencjonowaniem RNA . Używając swoistych przeciwciał (α-Ezh2) dokonali immunoprecypitacji jądrowego RNA wyizolowanego z mysich ES , a następnie zsekwencjonowali ściągnięty RNA przy użyciu platformy sekwencjonowania nowej generacji , Illumina.
- CLIP : Białko wiążące RNA jest sieciowane z RNA za pomocą światła UV przed lizą, po której następuje fragmentacja RNA, immunoprecypitacja, płukanie w wysokiej zawartości soli, SDS-PAGE, transfer błony, trawienie proteinazą, przygotowanie biblioteki cDNA i sekwencjonowanie w celu identyfikacji miejsc bezpośredniego wiązania RNA. CLIP po raz pierwszy połączono z wysokowydajnym sekwencjonowaniem w HITS-CLIP w celu określenia miejsc wiązania Nova-RNA w mózgu myszy oraz w iCLIP , który umożliwił amplifikację obciętych cDNA i wprowadził użycie UMI.
- ChIP-on-chip : Podobna technika, która wykrywa wiązanie białek z genomowym DNA, a nie z RNA.