Dependoparwowirus
| Klasyfikacja wirusów | |
|---|---|
| Dependoparvovirus | |
|
|
| (nierankingowe): | Wirus |
| królestwo : | Monodnaviria |
| Królestwo: | Shotokuvirae |
| Gromada: | Cossaviricota |
| Klasa: | Quintoviricetes |
| Zamówienie: | Piccovirales |
| Rodzina: | Parvoviridae |
| Podrodzina: | Parwowirinae |
| Rodzaj: | Dependoparwowirus |
| Gatunek | |
|
Patrz tekst |
|
Dependoparvovirus (dawniej Dependovirus lub grupa wirusów związanych z adenowirusami ) to rodzaj z podrodziny Parvovirinae z rodziny wirusów Parvoviridae ; są to wirusy grupy II zgodnie z klasyfikacją z Baltimore . Niektóre zależne parwowirusy są również znane jako wirusy związane z adenowirusami, ponieważ nie mogą replikować się w komórce gospodarza bez jednoczesnej infekcji komórki przez wirusa pomocniczego , takiego jak adenowirus , wirus opryszczki lub wirus krowianki .
Gatunek
Istnieje jedenaście uznanych gatunków:
- Adenoparwowirus związany z adenowirusem A
- Adenoparwowirus związany z adenoparwowirusem B
- Zależności parwowirusa Anseriform 1
- Zależności od ptasiego parwowirusa 1
- Zależności parwowirozy mięsożerców 1
- Chiropteran Dependoparvovirus 1
- Zależności parwowirozy płetwonogich 1
- Zależności od parwowirusa gryzoni 1
- Zależności od parwowirusa gryzoni 2
- Zależności od parwowirusa łuskowatego 1
- Zależności od parwowirusa łuskonośnego 2
Wirusologia
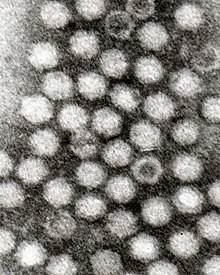
Dependoparwowirusy mają kształt dwudziestościanu , średnica 22 nm składa się z 60 białek w kształcie klina, liczba triangulacji = 1). W każdym kapsomerze obecne są trzy białka (VP1, VP2 i VP3). Każdy kapsyd składa się z 5 białek VP1, 5 VP2 i 50 VP3. Kapsyd nie posiada koperty.
Genom to pojedyncza cząsteczka jednoniciowego DNA o długości 4,7 kilozasad. Ma tylko dwie otwarte ramki odczytu . Otwarta ramka odczytu 3' to strukturalne białko kapsydu, cap, które można łączyć, tworząc dwa RNA, jeden dla białka wirionu 1 (VP1), a drugi ostatecznie tworzy VP2 i VP3. Drugi gen, rep, można złożyć w cztery różne, niestrukturalne białka regulatorowe, które wszystkie pomagają w replikacji genomu. Białka te są nazywane Rep 78, Rep68, Rep 52 i Rep 40 na podstawie ich masy cząsteczkowej.
Dzięki odwróconym powtórzeniom końcowym (ITR) na każdym końcu genomu powstaje drugorzędowa struktura w kształcie litery T. Komplementarne obszary pozostawiają jednoniciową grupę hydroksylową 3' , aby rozpocząć replikację. Ta grupa hydroksylowa 3' jest stosowana jako starter do syntezy nici wiodącej . Tworzone są zarówno dodatnie, jak i ujemne nici sensowne DNA. Podczas replikacji powstają dwuniciowe związki pośrednie; oznacza to, że dwie nici, sens pozytywny i negatywny, zostaną dopasowane.
Zakres gospodarzy
Wirusy te są zdolne do replikacji we wszystkich kręgowcach. Ogranicza ich tylko wirus, którym muszą zarazić, znany również jako wirus pomocniczy. Te wirusy pomocnicze są niezbędne do replikacji zależnego od parwowirusa. Częstym wirusem pomocniczym u ludzi jest adenowirus . [ potrzebne źródło ]
Terapia genowa
Dependoparwowirus nie jest wystarczająco zakaźny, aby wywołać odpowiedź immunologiczną ; sprawia to, że jest dobrym wirusem do wykorzystania jako narzędzie terapii genowej. Terapia genowa jest możliwym sposobem leczenia różnych zaburzeń i chorób o podłożu genetycznym. Obecnie opracowywane są wektory wirusowe do transportu genów do komórek ludzkich. Ponieważ wirus ten nie stymuluje odpowiedzi immunologicznej, może być skutecznie stosowany wielokrotnie bez neutralizacji przed infekcją [ potrzebne źródło ] . Innym powodem, dla którego te wirusy są niezawodnymi wektorami, jest znany punkt insercji genomu. Ten wirus zawsze wstawia swoją zawartość w to samo miejsce na chromosomie 19. [ Potrzebne źródło ] Ta przewidywalność może zmniejszyć szanse na wstawienie się do ważnego obszaru, który mógłby zakłócić normalne funkcjonowanie genu lub zwiększyć ryzyko zachorowania na raka.
W tej chwili jednym z wyzwań związanych z wykorzystaniem tego wirusa jako narzędzia terapeutycznego jest fakt, że genom jest dość mały. Przy mniej niż 5 kb w genomie ilość materiału genetycznego, który może zmieścić się w kapsydzie, jest ograniczona. Obecnie trwają prace nad zwiększeniem ilości informacji, które ten wektor może dostarczyć. Można to osiągnąć dzięki ITR znajdującym się zarówno na końcu 5', jak i 3' genomu. Ponieważ ITR mają tę samą sekwencję, pozostawią odsłonięte komplementarne nici, jeśli zostaną usunięte. Komplementarne nici mogą ulegać rekombinacji i łączyć ze sobą dwa wstawione fragmenty o długości 5 kb.
