MHETaza
Schemat wstążki | |||||||||
| MHetazy MHetazy (). | |||||||||
|---|---|---|---|---|---|---|---|---|---|
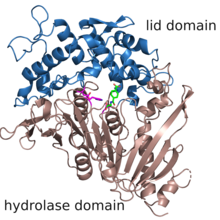 Domena hydrolazy jest pokazana na brązowo, w tym reszty katalityczne na karmazynowo. Domena wieczka jest pokazana na niebiesko. Analog substratu monohydroksyetyloterftalamid jest pokazany na zielono.
| |||||||||
| Identyfikatory | |||||||||
| nr WE | 3.1.1.102 | ||||||||
| Alt. nazwy | Hydrolaza MHET, hydrolaza tereftalanu monohydroksyetylu | ||||||||
| Bazy danych | |||||||||
| IntEnz | Widok IntEnz | ||||||||
| BRENDA | Wpis BRENDY | ||||||||
| ExPASy | Widok NiceZyme | ||||||||
| KEGG | Wpis KEGG | ||||||||
| MetaCyc | szlak metaboliczny | ||||||||
| PRYM | profil | ||||||||
| Struktury PDB | RCSB PDB PDBe PDB suma | ||||||||
| |||||||||
Enzym MHETaza jest hydrolazą , którą odkryto w 2016 roku. Rozszczepia kwas 2-hydroksyetylotereftalowy , produkt degradacji PET przez PETazę , do glikolu etylenowego i kwasu tereftalowego . Ta para enzymów, PETaza i MHETaza, umożliwia bakteriom Ideonella sakaiensis życie na plastikowym PET jako jedynym źródle węgla.
Reakcja chemiczna
Pierwszy enzym szlaku degradacji PET, PETaza , rozszczepia to tworzywo sztuczne na półprodukty MHET (kwas mono-(2-hydroksyetylo)tereftalowy ) i niewielkie ilości BHET (kwas bis-(2-hydroksyetylo)tereftalowy). MHETaza hydrolizuje wiązanie estrowe MHET tworząc kwas tereftalowy i glikol etylenowy .
Oprócz swojego naturalnego substratu MHET, chromogeniczny substrat MpNPT, tereftalan mono-p-nitrofenylu, jest również dobrze hydrolizowany. Można to wykorzystać do pomiaru aktywności enzymatycznej i określenia parametrów kinetycznych. Estry ferulanowe i galusanowe, substraty najbliższych krewnych z rodziny tannaz, nie ulegają konwersji. Estry p-nitrofenylowe alifatycznych kwasów monokarboksylowych, takie jak szeroko stosowany substrat esterazy, octan p-nitrofenylu, również nie ulegają hydrolizie.
Natywny enzym nie jest w stanie działać na BHET, izoftalan mono(2-hydroksyetylu) (MHEI) lub furanian mono(2-hydroksyetylu) (MHEF). MHEI jest prawdopodobnym przemysłowym produktem degradacji PET ze względu na użycie komonomeru izoftalanowego. MHEF jest produktem degradacji PEF przez PETazę. inżynierii białek mają na celu pokonanie tych barier.
Struktura
Struktura MHETazy została rozwiązana w 2019 roku. Pokazuje wspólny fałd nadrodziny hydrolaz alfa / beta . Zgodnie z klasyfikacją w bazie danych ESTHER, MHETaza należy do rodziny tannaz w obrębie bloku X. Rodzina ta zawiera głównie tannazy i esterazy feruloilowe. Enzym składa się z dwóch domen: domena hydrolazy zawiera reszty katalityczne Ser225, His528 i Asp492; domena wieczka dostarcza większość reszt miejsca wiązania substratu.

