Replikacja toczącego się koła
Replikacja toczącego się koła ( RCR ) to proces jednokierunkowej replikacji kwasu nukleinowego , który umożliwia szybką syntezę wielu kopii kolistych cząsteczek DNA lub RNA , takich jak plazmidy , genomy bakteriofagów i kolisty genom RNA wiroidów . Niektóre wirusy eukariotyczne również replikują swoje DNA lub RNA poprzez mechanizm toczącego się koła.
Jako uproszczoną wersję replikacji naturalnego toczącego się koła, opracowano technikę izotermicznej amplifikacji DNA , amplifikację toczącego się koła. Mechanizm RCA jest szeroko stosowany w biologii molekularnej i nanotechnologii biomedycznej , zwłaszcza w dziedzinie biosensingu (jako metoda wzmacniania sygnału).
Okrągła replikacja DNA
toczącego się koła DNA jest inicjowana przez białko inicjatorowe kodowane przez DNA plazmidu lub bakteriofaga, które nacina jedną nić dwuniciowej, kolistej cząsteczki DNA w miejscu zwanym początkiem podwójnej nici lub DSO. Białko inicjatora pozostaje związane z fosforanowym końcem 5' naciętej nici, a wolny koniec hydroksylowy 3' jest uwalniany, aby służyć jako starter do syntezy DNA przez polimerazę DNA III . Używając nienaciętej nici jako szablonu, replikacja przebiega wokół kolistej cząsteczki DNA, wypierając naciętą nić jako jednoniciowy DNA. Przemieszczenie naciętej nici jest przeprowadzane przez kodowaną przez gospodarza helikazę zwaną PcrA (skrót oznaczający zredukowaną kopię plazmidu) w obecności białka inicjującego replikację plazmidu.
Ciągła synteza DNA może wytworzyć wiele jednoniciowych liniowych kopii oryginalnego DNA w ciągłej serii od głowy do ogona zwanej konkatemerem . Te liniowe kopie można przekształcić w dwuniciowe okrągłe cząsteczki w następującym procesie:
Najpierw białko inicjatora wykonuje kolejne nacięcie w DNA, aby zakończyć syntezę pierwszej (wiodącej) nici. Polimeraza RNA i polimeraza DNA III następnie replikują DNA pochodzenia jednoniciowego (SSO), tworząc kolejne dwuniciowe koło. Polimeraza DNA I usuwa starter, zastępując go DNA, a ligaza DNA łączy końce, tworząc kolejną cząsteczkę dwuniciowego kolistego DNA.
Podsumowując, typowa replikacja toczącego się koła DNA składa się z pięciu etapów:
- Okrągły dsDNA zostanie „nacięty”.
- Koniec 3' jest wydłużany przy użyciu „nienaciętego” DNA jako nici wiodącej (szablon); Koniec 5' jest przesunięty.
- Przemieszczony DNA jest nicią opóźnioną i składa się z podwójnej nici poprzez serię fragmentów Okazaki .
- Replikacja zarówno „nienaciętego”, jak i przemieszczonego ssDNA.
- Przesunięty DNA zatacza koło.
Wirusologia
Replikacja wirusowego DNA
Niektóre wirusy DNA replikują swoje informacje genomowe w komórkach gospodarza poprzez replikację toczącego się koła. Na przykład ludzki herpeswirus-6 (HHV-6) (hibv) wykazuje ekspresję zestawu „wczesnych genów”, które, jak się uważa, biorą udział w tym procesie. Powstałe długie konkatamery są następnie cięte między regionami pac-1 i pac-2 genomu HHV-6 przez rybozymy , gdy jest on pakowany w poszczególne wiriony.
Wirus brodawczaka ludzkiego-16 (HPV-16) to kolejny wirus, który wykorzystuje replikację toczną do wytwarzania potomstwa z dużą szybkością. HPV-16 infekuje ludzkie komórki nabłonkowe i ma dwuniciowy kolisty genom. Podczas replikacji, w miejscu początku, heksamer E1 owija się wokół pojedynczej nici DNA i przesuwa się w kierunku od 3' do 5'. W normalnej replikacji dwukierunkowej dwa białka replikacyjne rozpadają się w momencie zderzenia, ale uważa się, że w przypadku HPV-16 heksamer E1 nie rozdziela się, co prowadzi do ciągłej replikacji toczącej się. Uważa się, że ten mechanizm replikacji HPV może mieć fizjologiczne implikacje dla integracji wirusa z chromosomem gospodarza i ewentualnej progresji do raka szyjki macicy.
Ponadto geminiwirus wykorzystuje również replikację toczącego się koła jako mechanizm replikacji. Jest to wirus odpowiedzialny za zniszczenie wielu głównych upraw, takich jak maniok, bawełna, rośliny strączkowe, kukurydza, pomidor i okra. Wirus ma okrągły, jednoniciowy DNA, który replikuje się w komórkach rośliny gospodarza. Cały proces jest inicjowany przez białko inicjujące replikację geminiwirusa, Rep, które jest również odpowiedzialne za zmianę środowiska gospodarza, aby działać jako część mechanizmu replikacji. Rep jest również uderzająco podobny do większości innych białek inicjujących replikację toczenia eubakterii, z obecnością motywów I, II i III na N-końcu. Podczas replikacji toczącego się koła ssDNA geminiwirusa jest przekształcane w dsDNA, a Rep jest następnie przyłączane do dsDNA w początkowej sekwencji TAATATTAC. Po tym, jak Rep, wraz z innymi białkami replikacyjnymi, zwiąże się z dsDNA, tworzy pętlę łodygi, w której DNA jest następnie cięte w sekwencji nanomeru, powodując przemieszczenie nici. To przemieszczenie pozwala widełkom replikacyjnym na postęp w kierunku od 3 'do 5', co ostatecznie daje nową nić ssDNA i konkatameryczną nić DNA.
replikacji DNA bakteriofaga T4 obejmują koliste i rozgałęzione koliste struktury konkatameryczne . Struktury te prawdopodobnie odzwierciedlają mechanizm replikacji toczącego się koła.
Replikacja wirusowego RNA
Niektóre wirusy i wiroidy RNA również replikują swój genom poprzez replikację RNA w toczącym się kole. W przypadku wiroidów istnieją dwa alternatywne szlaki replikacji RNA, po których odpowiednio podążają członkowie rodziny Pospivirodae (replikacja asymetryczna) i Avsunviroidae (replikacja symetryczna).
W rodzinie Pospiviroidae (podobne do PSTVd), kolista nić dodatnia RNA jest przepisywana przez polimerazę RNA gospodarza na nici oligomeryczne ujemne, a następnie nici oligomeryczne nić dodatnie. Te oligomeryczne nici dodatnie są cięte przez RNazę gospodarza i ligowane przez ligazę RNA gospodarza w celu zreformowania monomerycznej kolistej nici dodatniej RNA. Nazywa się to asymetryczną ścieżką replikacji toczącego się koła. Wiroidy z rodziny Avsunviroidae (podobne do ASBVd) replikują swój genom poprzez symetryczną ścieżkę replikacji toczącego się koła. W tej symetrycznej ścieżce oligomeryczne nici minus są najpierw cięte i łączone w celu utworzenia monomerycznych nici minus, a następnie są transkrybowane na oligomeryczne nici plus. Te oligomeryczne nici plus są następnie cięte i łączone w celu zreformowania monomerycznej nici plus. Symetryczna ścieżka replikacji została nazwana, ponieważ obie nici plus i minus są wytwarzane w ten sam sposób.
W rozszczepianiu oligomerycznych nici plus i minus pośredniczy samorozszczepiająca się struktura rybozymu typu młotek obecna w Avsunviroidae, ale taka struktura jest nieobecna w Pospiviroidae.
Wzmocnienie toczącego się koła
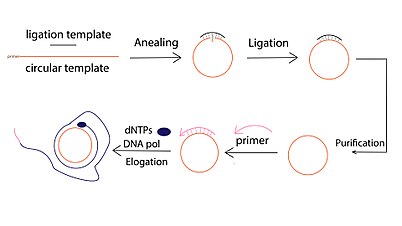
Pochodna forma replikacji toczącego się koła została z powodzeniem zastosowana do amplifikacji DNA z bardzo małych ilości materiału wyjściowego. Ta technika amplifikacji nosi nazwę Rolling Circle Amplification (RCA). W odróżnieniu od konwencjonalnych technik amplifikacji DNA, takich jak reakcja łańcuchowa polimerazy (PCR) , RCA jest izotermiczną techniką amplifikacji kwasów nukleinowych , w której polimeraza w sposób ciągły dodaje pojedyncze nukleotydy do startera połączonego z kolistą matrycą, co skutkuje długim konkamerem ssDNA zawierającym od dziesiątek do setek powtórzeń tandemowych (uzupełniające szablon okrągły).
Istnieje pięć ważnych elementów wymaganych do przeprowadzenia reakcji RCA:
- Polimeraza DNA
- Odpowiedni bufor, który jest kompatybilny z polimerazą.
- Krótki starter DNA lub RNA
- Okrągły szablon DNA
- Trifosforany dezoksynukleotydów (dNTP)
Polimerazy stosowane w RCA to egzopolimeraza DNA Phi29 , Bst i Vent do amplifikacji DNA oraz polimeraza RNA T7 do amplifikacji RNA. Ponieważ polimeraza DNA Phi29 ma najlepszą procesywność i zdolność przemieszczania nici spośród wszystkich wymienionych polimeraz, była najczęściej stosowana w reakcjach RCA. W odróżnieniu od reakcji łańcuchowej polimerazy (PCR), RCA można prowadzić w stałej temperaturze (temperatura pokojowa do 65°C) zarówno w wolnym roztworze, jak i na wierzchu unieruchomionych celów (amplifikacja w fazie stałej).
Reakcja DNA RCA obejmuje zazwyczaj trzy etapy:
- Ligacja z kołową matrycą, którą można przeprowadzić poprzez ligację enzymatyczną za pośrednictwem matrycy (np. ligaza DNA T4) lub ligację bez matrycy przy użyciu specjalnych ligaz DNA (tj. CircLigase).
- Wydłużenie jednoniciowego DNA wywołane starterem . Do hybrydyzacji z tym samym okręgiem można zastosować wiele starterów. W rezultacie można zainicjować wiele zdarzeń amplifikacji, wytwarzając wiele produktów RCA („Multiprimed RCA”).
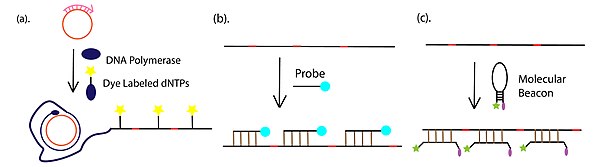
- Wykrywanie i wizualizacja produktu amplifikacji, która jest najczęściej przeprowadzana za pomocą detekcji fluorescencyjnej, z dNTP sprzężonym z fluoroforem, komplementarnymi lub znakowanymi fluorescencyjnie latarniami molekularnymi . Oprócz metod fluorescencyjnych elektroforeza żelowa jest również szeroko stosowana do wykrywania produktu RCA.
RCA wytwarza liniową amplifikację DNA, ponieważ każda kolista matryca rośnie z określoną prędkością przez określony czas. Aby zwiększyć wydajność i osiągnąć amplifikację wykładniczą, jak ma to miejsce w przypadku PCR, zbadano kilka podejść. Jednym z nich jest hiperrozgałęziona amplifikacja toczącego się koła lub HRCA, w której dodaje się i wydłuża startery, które łączą się z oryginalnymi produktami RCA. W ten sposób oryginalny RCA tworzy więcej szablonów, które można wzmacniać. Innym jest amplifikacja koło w kółko lub C2CA, w której produkty RCA są trawione enzymem restrykcyjnym i łączone z nowymi kolistymi matrycami przy użyciu oligo restrykcyjnego, po czym następuje nowa runda RCA z większą ilością kolistych matryc do amplifikacji.
Zastosowania RCA
RCA może wzmocnić pojedyncze zdarzenie wiązania molekularnego ponad tysiąckrotnie, co czyni go szczególnie przydatnym do wykrywania celów o bardzo małej liczebności. Reakcje RCA można przeprowadzać nie tylko w środowisku wolnych roztworów, ale także na powierzchni stałej, takiej jak szkło, mikro- lub nanokulki, płytki z mikrodołkami, urządzenia mikroprzepływowe, a nawet paski papieru. Ta cecha sprawia, że jest to bardzo potężne narzędzie do wzmacniania sygnałów w testach immunologicznych fazy stałej (np. ELISA ). W ten sposób RCA staje się wysoce wszechstronnym narzędziem do wzmacniania sygnału o szerokim zastosowaniu w genomice, proteomice, diagnostyce i bioczujnikach.
Immuno-RCA
Immuno-RCA to metoda izotermicznej amplifikacji sygnału służąca do wykrywania i oznaczania ilościowego białek o wysokiej specyficzności i wysokiej czułości. Technika ta łączy w sobie dwie dziedziny: RCA, która umożliwia amplifikację nukleotydów, oraz test immunologiczny, w którym wykorzystuje się przeciwciała specyficzne dla wewnątrzkomórkowych lub wolnych biomarkerów. W rezultacie immuno-RCA daje specyficzny wzmocniony sygnał (wysoki stosunek sygnału do szumu), dzięki czemu nadaje się do wykrywania, oznaczania ilościowego i wizualizacji markerów białkowych o niskiej liczebności w testach immunologicznych w fazie ciekłej i immunohistochemii .
Immuno-RCA następuje po typowej reakcji immunoadsorbcyjnej w teście ELISA lub immunohistochemicznym barwieniu tkanek. Przeciwciała wykrywające stosowane w reakcji immuno-RCA są modyfikowane poprzez dołączenie oligonukleotydu ssDNA na końcu łańcuchów ciężkich. Zatem sekcja Fab (fragment, wiązanie antygenu) przeciwciała wykrywającego może nadal wiązać się ze specyficznymi antygenami, a oligonukleotyd może służyć jako starter reakcji RCA.
Typowa procedura immuno-RCA, w której pośredniczą przeciwciała, jest następująca:
1. Przeciwciało wykrywające rozpoznaje określony cel białkowy. To przeciwciało jest również przyłączone do startera oligonukleotydowego.
2. Gdy obecny jest kolisty DNA, jest on hybrydyzowany, a starter dopasowuje się do komplementarnej sekwencji kolistego DNA.
3. Komplementarna sekwencja kolistej matrycy DNA jest kopiowana setki razy i pozostaje przyłączona do przeciwciała.
4. Wyjście RCA (wydłużone ssDNA) jest wykrywane za pomocą sond fluorescencyjnych przy użyciu mikroskopu fluorescencyjnego lub czytnika mikropłytek.
Immuno-RCA na bazie aptamerów
Oprócz immuno-RCA, w którym pośredniczy przeciwciało, starter ssDNA RCA można również skoniugować z końcem 3' aptameru DNA. Ogon startera można zamplifikować poprzez amplifikację toczącego się koła. Produkt można wizualizować poprzez oznakowanie reporterem fluorescencyjnym. Proces ten przedstawiono na rysunku po prawej stronie.
Inne zastosowania RCA
Różne pochodne RCA były szeroko stosowane w dziedzinie bioczujników. Na przykład RCA jest z powodzeniem stosowany do wykrywania wirusowego i bakteryjnego DNA z próbek klinicznych, co jest bardzo korzystne dla szybkiej diagnostyki chorób zakaźnych . Został również wykorzystany jako metoda amplifikacji sygnału na chipie w mikromacierzy kwasu nukleinowego (zarówno dla DNA, jak i RNA) .
Oprócz funkcji amplifikacji w zastosowaniach bioczujnikowych, technika RCA może być również stosowana do budowy nanostruktur DNA i hydrożeli DNA. Produkty RCA mogą być również wykorzystywane jako matryce do okresowego składania nanogatunków lub białek, syntezy metalicznych nanodrutów i tworzenia nano-wysp.
Zobacz też
Linki zewnętrzne
- Systemy replikacji DNA używane z małymi kolistymi cząsteczkami DNA Genomes 2 , T. Brown i in., w NCBI Books
- MicrobiologyBytes: Wiroidy i Wirusoidy
- http://mcmanuslab.ucsf.edu/node/246