Okrągły RNA
Okrągły RNA (lub circRNA ) to rodzaj jednoniciowego RNA , który w przeciwieństwie do liniowego RNA tworzy kowalencyjnie zamkniętą ciągłą pętlę. W kolistym RNA końce 3' i 5' normalnie obecne w cząsteczce RNA zostały połączone ze sobą. Ta cecha nadaje kolistemu RNA liczne właściwości, z których wiele dopiero niedawno zidentyfikowano.
Wiele rodzajów kolistego RNA powstaje z genów kodujących białka. Wykazano, że niektóre koliste RNA kodują białka. Niektóre typy kolistego RNA również niedawno wykazały potencjał jako regulatory genów. Biologiczna funkcja większości kolistych RNA jest niejasna.
Ponieważ kolisty RNA nie ma końców 5' ani 3', jest odporny na degradację za pośrednictwem egzonukleazy i prawdopodobnie jest bardziej stabilny niż większość liniowych RNA w komórkach. Okrągły RNA został powiązany z niektórymi chorobami, takimi jak rak.
splicing RNA
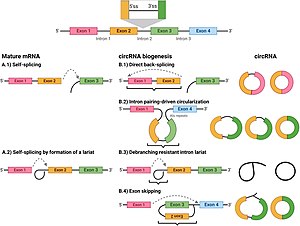
W przeciwieństwie do genów u bakterii , geny eukariotyczne są podzielone przez niekodujące sekwencje zwane intronami . U eukariontów, gdy gen jest transkrybowany z DNA do informacyjnego RNA (mRNA), interweniujące introny są usuwane, pozostawiając tylko egzony w dojrzałym mRNA, które można następnie poddać translacji w celu wytworzenia produktu białkowego. Spliceosom się w jądrze, katalizuje splicing w następujący sposób:
- Spliceosom rozpoznaje intron , który jest otoczony specyficznymi sekwencjami na jego końcach 5' i 3', znanymi odpowiednio jako donorowe miejsce splicingowe (lub miejsce splicingowe 5') i akceptorowe miejsce splicingowe (lub miejsce splicingowe 3') .
- Sekwencja miejsca splicingowego 5' jest następnie poddawana atakowi nukleofilowemu przez następną sekwencję zwaną punktem rozgałęzienia, w wyniku czego powstaje kolista struktura zwana lariatem.
- Następnie wolny ekson 5' atakuje miejsce składania 3', łącząc dwa eksony i uwalniając strukturę znaną jako lariat intronu . Lariat intronu jest następnie rozgałęziony i szybko degradowany.
Splicing alternatywny
Splicing alternatywny to zjawisko, w wyniku którego jeden transkrypt RNA może dawać różne produkty białkowe w oparciu o to, które segmenty są uważane za „introny” i „egzony” podczas zdarzenia splicingu. Chociaż nie jest specyficzna dla ludzi, jest to częściowe wyjaśnienie faktu, że ludzie i inne znacznie prostsze gatunki (takie jak nicienie) mają podobną liczbę genów (w zakresie 20 - 25 tysięcy). Jednym z najbardziej uderzających przykładów alternatywnego splicingu jest DSCAM Drosophila , który może dać początek około 30 tysiącom różnych izoform o alternatywnym splicingu.
Splicing niekanoniczny
Szyfrowanie eksonów
Szyfrowanie egzonów, zwane także tasowaniem eksonów, opisuje zdarzenie, w którym eksony są łączone w „niekanonicznej” (nietypowej) kolejności. Istnieją trzy sposoby, w jakie może wystąpić szyfrowanie eksonów:
- Tandemowa duplikacja egzonu w genomie, która często występuje w nowotworach.
- Trans-splicing , w którym dwa transkrypty RNA łączą się, w wyniku czego powstaje liniowy transkrypt zawierający eksony, które na przykład mogą pochodzić z genów zakodowanych na dwóch różnych chromosomach. Trans-splicing jest bardzo powszechny u C. elegans
- Miejsce dawcy splicingu jest połączone z miejscem akceptora splicingu dalej w górę w pierwotnym transkrypcie, dając transkrypt kolisty.
Pogląd, że zakreślone transkrypty są produktami ubocznymi niedoskonałego splicingu, jest wspierany przez niską liczebność i brak zachowania sekwencji większości circRNA, ale został zakwestionowany.
Elementy Alu wpływają na splicing circRNA
Powtarzające się sekwencje Alu stanowią około 10% ludzkiego genomu. Obecność Alu w flankujących intronach genów kodujących białka, przylegających do pierwszego i ostatniego egzonu tworzącego circRNA, wpływa na powstawanie circRNA. Ważne jest, aby flankujące intronowe elementy Alu były komplementarne, ponieważ umożliwia to parowanie RNA, co z kolei ułatwia syntezę circRNA.
Wpływ edycji RNA na powstawanie circRNA
RNA mogą podlegać modyfikacji zasad poprzez edycję RNA po transkrypcji. Edycja RNA zachodzi głównie w elementach Alu genów kodujących białka. Edycja RNA od A do I w intronowych elementach Alu w górę iw dół, flankujących miejsce splicingu wstecznego (BSS) może zmniejszyć tworzenie się circRNA w ludzkim sercu. W niewydolnym ludzkim sercu dominująca redukcja edycji RNA A-do-I prowadzi do zwiększonego tworzenia circRNA, w czym prawdopodobnie pośredniczy lepsze komplementarne parowanie RNA elementów Alu flankujących miejsce splicingu wstecznego.
Charakterystyka kolistego RNA
Wczesne odkrycia circRNA
Wczesne odkrycia kolistych RNA doprowadziły do przekonania, że nie mają one znaczenia ze względu na ich rzadkość. Te wczesne odkrycia obejmowały analizę genów, takich jak DCC i Sry , oraz niedawne odkrycie ludzkiego niekodującego RNA ANRIL , z których wszystkie wyrażały okrągłe izoformy. Odkryto również geny wytwarzające CircRNA, takie jak ludzki gen ETS-1 , ludzkie i szczurze geny cytochromu P450 , szczurzy gen białka wiążącego androgeny ( Shbg ) oraz ludzki gen dystrofiny.
Identyfikacja circRNA w całym genomie
Zaszyfrowane izoformy i circRNA
W 2012 roku, starając się wstępnie zidentyfikować specyficzne dla raka zdarzenia szyfrowania egzonów, odkryto dużą liczbę eksonów pomieszanych zarówno w komórkach normalnych, jak i nowotworowych. Stwierdzono, że pomieszane izoformy eksonu stanowiły około 10% wszystkich izoform transkryptu w leukocytach , przy czym zidentyfikowano 2748 pomieszanych izoform w embrionalnych komórkach macierzystych HeLa i H9 . Ponadto około 1 na 50 eksprymowanych genów wytwarzało pomieszane izoformy transkryptu przez co najmniej 10% czasu. Testy stosowane do rozpoznawania kolistości obejmowały traktowanie próbek RNazą R , enzymem, który rozkłada liniowe, ale nie koliste RNA, oraz testowanie obecności ogonów poli-A , które nie występują w cząsteczkach kolistych. Ogólnie stwierdzono, że 98% wymieszanych izoform reprezentuje circRNA, stwierdzono, że circRNA znajdują się w cytoplazmie, a circRNA występują w obfitości.
Odkrycie większej obfitości circRNA
W 2013 roku odkryto większą obfitość circRNA. Ludzki fibroblastów traktowano RNazą R w celu wzbogacenia w okrągłe RNA, a następnie kategoryzowano okrągłe transkrypty na podstawie ich obfitości (niska, średnia, wysoka). Stwierdzono, że około 1 na 8 eksprymowanych genów wytwarza wykrywalne poziomy circRNA, w tym te o niskiej liczebności, które były znacznie wyższe niż wcześniej podejrzewano i przypisywano je większej głębokości sekwencjonowania .
Specyficzność tkankowa CircRNA i aktywność antagonistyczna
W tym samym czasie opracowano metodę obliczeniową do wykrywania circRNA, która doprowadziła do wykrycia de novo circRNA u ludzi, myszy i C. elegans oraz ich obszernej walidacji. Często stwierdzano, że ekspresja circRNA jest specyficzna dla tkanki/etapu rozwojowego. Ponadto stwierdzono, że circRNA mają zdolność działania jako antagoniści miRNA, mikroRNA, które zakłócają translację mRNA, czego przykładem jest CDR1as circRNA, który ma miejsca wiązania miRNA (jak widać poniżej).
Dane CircRNA i ENCODE Ribozero RNA-seq
W 2014 r. zidentyfikowano i oznaczono ilościowo ludzkie circRNA na podstawie danych ENCODE Ribozero RNA-seq . Stwierdzono, że większość circRNA to mniejsze izoformy składania i ulegają ekspresji tylko w kilku typach komórek, przy czym 7112 ludzkich circRNA ma ułamki kołowe (ułamek podobieństwa, jaki izoforma ma do transkrypcji tego samego locus) co najmniej 10%. Stwierdzono również, że circRNA nie są bardziej konserwatywne niż ich liniowe kontrole i zgodnie z profilowaniem rybosomów nie ulegają translacji. Jak wcześniej zauważono, circRNA mają zdolność działania jako antagoniści miRNA, co jest również znane jako potencjał do działania jako gąbki mikroRNA. Oprócz CDR1as bardzo niewiele circRNA ma potencjał działania jako gąbki mikroRNA. Ogólnie rzecz biorąc, większość kolistych RNA okazała się nieistotnymi produktami ubocznymi niedoskonałego splicingu.
CircRNA i CIRCexplorer
opracowano CIRCexplorer, narzędzie służące do identyfikacji tysięcy circRNA u ludzi bez danych RNazy R RNA-seq . Stwierdzono, że zdecydowana większość zidentyfikowanych egzonowych kolistych RNA o wysokiej ekspresji została przetworzona z egzonów znajdujących się w środku genów RefSeq , co sugeruje, że tworzenie kolistego RNA jest generalnie sprzężone ze splicingiem RNA. Ustalono, że większość kolistych RNA zawiera wiele, najczęściej od dwóch do trzech eksonów. Stwierdzono, że eksony z circRNA z tylko jednym eksonem w kształcie koła są znacznie dłuższe niż eksony z circRNA z wieloma eksonami w kształcie koła, co wskazuje, że przetwarzanie może preferować pewną długość, aby zmaksymalizować krążenie egzonu (ów). Introny okrągłych eksonów na ogół zawierają duże Alu , które mogą tworzyć odwrócone powtarzające się pary Alu (IRAlus). IRAlus, zbieżne lub rozbieżne, są zestawione w poprzek flankujących intronów circRNA w sposób równoległy z podobnymi odległościami do sąsiednich eksonów. Stwierdzono również, że IRAlus i inne niepowtarzające się, ale uzupełniające się sekwencje sprzyjają tworzeniu kolistego RNA. Z drugiej strony określono, że na wydajność cyrkulacji eksonów wpływa konkurencja parowania RNA, tak że alternatywne parowanie RNA i jego konkurencja prowadzi do alternatywnej cyrkulacji. Wreszcie stwierdzono, że zarówno cyklizacja egzonu, jak i jego regulacja są dynamiczne ewolucyjnie.
Wywołanie circRNA w całym genomie w przypadkach choroby Alzheimera
Laboratorium Cruchaga przeprowadziło pierwsze analizy circRNA na dużą skalę w chorobie Alzheimera (AD) i wykazało rolę circRNA w zdrowiu i chorobie. Stwierdzono, że łącznie 148 circRNA było istotnie powiązanych w wielu zestawach danych ze choroby Alzheimera i oceną klinicznej demencji (CDR) w chwili śmierci po korekcie współczynnika fałszywego odkrycia (FDR). Ekspresja circRNA była niezależna od postaci liniowej i ta ekspresja circRNA była również korygowana proporcjami komórek. Stwierdzono również, że circRNA są współeksprymowane ze znanymi przyczynowymi genami Alzheimera, takimi jak APP i PSEN1 , co wskazuje, że niektóre circRNA są również częścią szlaku przyczynowego. W sumie stwierdzono, że ekspresja circRNA w mózgu wyjaśnia więcej objawów klinicznych choroby Alzheimera niż liczba alleli APOε4, co sugeruje, że circRNA można wykorzystać jako potencjalny biomarker choroby Alzheimera.
Klasy CircRNA
Okrągłe RNA można podzielić na pięć klas:
| Klasy okrągłych RNA | Opis |
| Wiroidy i wirus zapalenia wątroby typu delta (HDV) | W wiroidach i HDV jednoniciowe circRNA są niezbędne w replikacji RNA. Cykliczność pozwala, aby jedno zdarzenie inicjujące doprowadziło do powstania wielu kopii genomowych w procesie znanym również jako replikacja RNA w toczącym się kole . |
| CircRNA z intronów | Cząsteczki kołowe są wytwarzane przez introny wytwarzane ze splicingu spliceosomalnego, splicingu tRNA oraz intronów grupy I i grupy II (samoskładające się rybozymy). Introny grupy I tworzą circRNA poprzez autokatalityczne działanie rybozymów i chociaż można je wykryć in vivo, ich funkcja nie została jeszcze określona. Introny grupy II również generują circRNA in vivo . Okrągłe introny wytwarzane przez eukariotyczny spliceosomalny splicing to koliste lariaty intronów znane jako koliste intronowe RNA (ciRNA). Ze względu na cyrkulację ciRNA mogą uniknąć degradacji i uważa się, że są wysoce nadreprezentowane. Funkcja ciRNA jest obecnie nieznana; spekuluje się jednak, że mogą odgrywać rolę we wzmacnianiu transkrypcji genów, z których są wytwarzane, ponieważ oddziałują z polimerazą RNA II. |
| CircRNA z półproduktów w reakcjach przetwarzania RNA | Są one najpierw łączone z prekursorów w postaci liniowych cząsteczek, a następnie koliste za pomocą ligazy. Są one niezbędne do umożliwienia przegrupowania w kolejności sekwencji RNA i niezbędne w biogenezie permutowanych genów tRNA w niektórych algach i archeonach. |
| Niekodujące circRNA w archeonach | Niektóre gatunki archeonów mają circRNA, które są wytwarzane z wyciętych kolistych intronów tRNA. Uważa się, że krążenie funkcjonalnych niekodujących RNA działa jako mechanizm ochronny przed egzonukleazami i promuje prawidłowe fałdowanie. |
| CircRNA u eukariontów wytwarzane przez splicing wsteczny | Okrągłe RNA wytwarzane przez splicing wsteczny (forma mieszania eksonów) występują, gdy miejsce splicingowe 5 'jest połączone z miejscem splicingowym 3' w górę. Obecnie u ludzi zidentyfikowano ponad 25 000 różnych circRNA. |
Długość circRNA
Niedawne badanie ludzkich circRNA ujawniło, że cząsteczki te składają się zwykle z 1–5 eksonów. Każdy z tych eksonów może być do trzech razy dłuższy niż przeciętnie eksprymowany ekson, co sugeruje, że długość eksonu może odgrywać rolę w podejmowaniu decyzji, które eksony mają zostać zakreślone. 85% egzonów o okrągłym kształcie pokrywa się z egzonami, które kodują białko , chociaż same okrągłe RNA nie wydają się ulegać translacji. Podczas tworzenia circRNA ekson 2 jest często eksonem „akceptorowym” w górę.
Introny otaczające eksony, które zostały wybrane do kolistości, są średnio do trzech razy dłuższe niż te, które nie flankują egzonów przed kołem, chociaż nie jest jeszcze jasne, dlaczego tak jest. W porównaniu z regionami, które nie dają kółek, te introny znacznie częściej zawierają komplementarne odwrócone Alu , przy czym Alu jest najczęstszym transpozonem w genomie. Zaproponowano, że powtarzające się pary zasad Alu mogą umożliwić wzajemne odnajdywanie się miejsc splicingu, ułatwiając w ten sposób cyrkulację.
Introny w circRNA są zatrzymywane ze stosunkowo wysoką częstotliwością (~ 25%), dodając w ten sposób dodatkową sekwencję do dojrzałych circRNA.
Lokalizacja circRNA w komórce
W komórce circRNA znajdują się głównie w cytoplazmie , gdzie liczba okrągłych transkryptów RNA pochodzących z genu może być nawet dziesięciokrotnie większa niż liczba powiązanych liniowych RNA generowanych z tego locus . Nie jest jasne, w jaki sposób koliste RNA opuszczają jądro przez stosunkowo mały por jądrowy . Ponieważ otoczka jądrowa rozpada się podczas mitozy , jedna z hipotez głosi, że cząsteczki opuszczają jądro podczas tej fazy cyklu komórkowego . Jednak niektóre circRNA, takie jak CiRS-7/CDR1as, ulegają ekspresji w tkankach neuronalnych, w których nie występuje podział mitotyczny.
CircRNA są stabilne w porównaniu z liniowymi RNA
CircRNA nie mają poliadenylowanego ogona i dlatego przewiduje się, że będą mniej podatne na degradację przez egzonukleazy. W 2015 roku Enuka i in. zmierzyli okresy półtrwania 60 circRNA i ich liniowych odpowiedników ulegających ekspresji z tego samego genu gospodarza i ujawnili, że mediana okresu półtrwania circRNA komórek sutka (18,8 do 23,7 godzin) jest co najmniej 2,5 razy dłuższa niż mediana okresu półtrwania ich liniowe odpowiedniki (od 4,0 do 7,4 godziny). Ogólnie czas życia cząsteczek RNA określa czas ich odpowiedzi. W związku z tym doniesiono, że circRNA sutka powoli reagują na stymulację czynnikami wzrostu.
Prawdopodobne funkcje kolistego RNA
Ewolucyjna konserwacja mechanizmów i sygnałów cyrkularnych
CircRNA zidentyfikowano u różnych gatunków we wszystkich dziedzinach życia . W 2011 roku Danan i in. zsekwencjonowany RNA z Archaea . Po strawieniu całkowitego RNA za pomocą RNazy R byli w stanie zidentyfikować koliste gatunki, co wskazuje, że circRNA nie są specyficzne dla eukariontów. Jednak te koliste gatunki archeologiczne prawdopodobnie nie powstają poprzez splicing, co sugeruje, że prawdopodobnie istnieją inne mechanizmy generowania kolistego RNA.
Stwierdzono, że CircRNA są w dużej mierze konserwatywne między ludźmi a owcami. Analizując dane sekwencjonowania całkowitego RNA z kory płata ciemieniowego owcy i komórek jednojądrzastych krwi obwodowej, wykazano, że 63% wykrytych circRNA jest homologicznych do znanych ludzkich circRNA.
W bliższym powiązaniu ewolucyjnym porównanie RNA z jąder myszy z RNA z komórki ludzkiej wykazało 69 ortologicznych circRNA. Na przykład zarówno ludzie, jak i myszy kodują geny HIPK2 i HIPK3 , dwie paralogiczne kinazy, które wytwarzają dużą ilość circRNA z jednego określonego eksonu u obu gatunków. Konserwacja ewolucyjna wzmacnia prawdopodobieństwo istotnej i znaczącej roli cyrkulacji RNA.
CDR1as/CiRS-7 jako gąbka miR-7
mikroRNA (miRNA) to małe (~ 21 nt) niekodujące RNA, które hamują translację informacyjnych RNA zaangażowanych w duży, zróżnicowany zestaw procesów biologicznych. Bezpośrednio łączą się w pary z docelowymi informacyjnymi RNA (mRNA) i mogą wyzwalać cięcie mRNA w zależności od stopnia komplementarności.
MikroRNA są pogrupowane w „rodziny nasion”. Członkowie rodziny dzielą nukleotydy 2–7, znane jako region nasion. argonaute to „białka efektorowe”, które pomagają miRNA wykonywać swoją pracę, podczas gdy gąbki mikroRNA to RNA, które „gąbczą” miRNA z określonej rodziny, służąc w ten sposób jako konkurencyjne inhibitory, które hamują zdolność miRNA do wiązania jego cele mRNA, dzięki obecności wielu miejsc wiążących, które rozpoznają określony region nasion. Niektóre koliste RNA mają wiele miejsc wiązania miRNA, co dało wskazówkę, że mogą one działać jako gąbki. Dwa ostatnie artykuły potwierdziły tę hipotezę, szczegółowo badając kolistą gąbkę zwaną CDR1as/CiRS-7, podczas gdy inne grupy nie znalazły bezpośrednich dowodów na to, że koliste RNA działają jak gąbki miRNA, analizując potencjalną interakcję kolistych RNA z białkiem Argonauta (AGO) za pomocą dane z wysokowydajnego sekwencjonowania RNA wyizolowanego przez sieciowanie i immunoprecypitację (HITS-CLIP).
CDR1as/CiRS-7 jest zakodowany w genomie antysensownie do ludzkiego locus CDR1 (genu) (stąd nazwa CDR1as) i celuje w miR-7 (stąd nazwa CiRS-7 – Circular RNA Sponge for miR-7 ). Ma ponad 60 miejsc wiążących miR-7, znacznie więcej niż jakakolwiek znana gąbka liniowego miRNA.
AGO2 jest białkiem Argonaute związanym z miR-7 (patrz wyżej). Chociaż CDR1as/CiRS-7 może być cięty przez miR-671 i związane z nim białko Argonaute, nie może być cięty przez miR-7 i AGO2. Aktywność cięcia mikroRNA zależy od komplementarności poza 12. pozycją nukleotydową; żadne z miejsc wiążących CiRS-7 nie spełnia tego wymagania.
Eksperyment z danio pręgowanym , który nie ma locus CDR1 w swoim genomie, dostarcza dowodów na aktywność gąbki CiRS-7. Podczas rozwoju miR-7 ulega silnej ekspresji w mózgu danio pręgowanego. Aby wyciszyć ekspresję miR-7 u danio pręgowanego, Memczak i współpracownicy wykorzystali narzędzie o nazwie morfolino , które może tworzyć pary zasad i sekwestrować cząsteczki docelowe. Leczenie morfolino miało taki sam poważny wpływ na rozwój śródmózgowia, jak ektopowa ekspresja CiRS-7 w mózgach danio pręgowanego przy użyciu wstrzykniętych plazmidów . Wskazuje to na znaczącą interakcję między CiRS-7 i miR-7 in vivo.
Inną godną uwagi okrągłą gąbką miRNA jest SRY . SRY, który jest silnie wyrażany w mysich jądrach, działa jak miR-138 . W genomie SRY jest otoczony długimi odwróconymi powtórzeniami (IR) o długości ponad 15,5 kilozasad (kb). Gdy jeden lub oba IR zostaną usunięte, cyrkulacja nie występuje. To właśnie to odkrycie wprowadziło pomysł odwróconych powtórzeń umożliwiających cyrkulację.
Ponieważ okrągłe gąbki RNA charakteryzują się wysokimi poziomami ekspresji, stabilnością i dużą liczbą miejsc wiązania miRNA, prawdopodobnie będą bardziej efektywnymi gąbkami niż te, które są liniowe.
Inne możliwe funkcje circRNA
Chociaż ostatnio uwaga skupiła się na funkcjach „gąbki” circRNA, naukowcy rozważają również kilka innych możliwości funkcjonalnych. Na przykład niektóre obszary dorosłego hipokampa myszy wykazują ekspresję CiRS-7, ale nie miR-7, co sugeruje, że CiRS-7 może pełnić role niezależne od interakcji z miRNA.
Potencjalne role obejmują:
- Wiązanie się z białkami wiążącymi RNA (RBP) i RNA oprócz miRNA, tworząc kompleksy RNA-białko. Kompleksy te mogą regulować interakcje RBP i RNA, na przykład z kanonicznym liniowym transkryptem genu lub infekcją wirusową.
- Produkcja białka
- Chen i Sarnow 1995 wykazali, że syntetyczny circRNA, który zawierał IRES (wewnętrzne miejsce wejścia rybosomu) wytwarzał produkt białkowy in vitro , podczas gdy bez IRES nie. Chociaż testowany circRNA był czysto sztucznym konstruktem, Chen i Sarnow stwierdzili w swoim artykule, że byliby zainteresowani sprawdzeniem, czy kręgi naturalnie zawierają elementy IRES.
- Jeck i in. 2013: Przetestowano naturalne circRNA, które zawierały translację „kodon startowy”. Jednak żadna z tych cząsteczek nie wiązała się z rybosomami, co sugeruje, że wiele circRNA może nie ulegać translacji in vivo .
- Transport miRNA wewnątrz komórki. Fakt, że CiRS-7 może zostać pocięty przez miR-671, może wskazywać na istnienie systemu uwalniającego „ładunek” miRNA w odpowiednim czasie.
- Regulacja mRNA w komórce poprzez ograniczone parowanie zasad. Formalnie możliwe jest, że miR-7 moderuje aktywność regulacyjną CiRS-7, a nie odwrotnie!
Okrągłe intronowe długie niekodujące RNA (ciRNA)
Zwykle intronowe lassy (patrz wyżej) są odgałęzione i szybko degradowane. Jednak niepowodzenie usuwania rozgałęzień może prowadzić do tworzenia kolistych intronowych długich niekodujących RNA, znanych również jako ciRNA. Tworzenie ciRNA, zamiast być procesem losowym, wydaje się zależeć od obecności określonych elementów w pobliżu miejsca składania 5 'i miejsca rozgałęzienia (patrz wyżej).
CiRNA różnią się od circRNA tym, że znajdują się w jądrze, a nie w cytoplazmie . Ponadto cząsteczki te zawierają niewiele (jeśli w ogóle) miejsc wiążących miRNA. Wydaje się, że zamiast działać jak gąbki, ciRNA regulują ekspresję swoich genów macierzystych. Na przykład stosunkowo obfite ciRNA zwane ci-ankrd52 pozytywnie reguluje Pol II . Wiele ciRNA pozostaje w swoich „miejscach syntezy” w jądrze. Jednak ciRNA może pełnić inne role niż zwykła regulacja ich genów macierzystych, ponieważ ciRNA lokalizują się w dodatkowych miejscach w jądrze innych niż ich „miejsca syntezy”.
Okrągły RNA i choroba
Podobnie jak w przypadku większości zagadnień z biologii molekularnej , ważne jest, aby zastanowić się, w jaki sposób kolisty RNA może być użyty jako narzędzie pomagające ludzkości. Biorąc pod uwagę jego obfitość, konserwację ewolucyjną i potencjalną rolę regulacyjną, warto przyjrzeć się, w jaki sposób można wykorzystać koliste RNA do badania patogenezy i opracowywania interwencji terapeutycznych. Na przykład:
- Okrągły ANRIL (cANRIL) to kolista forma ANRIL, długiego niekodującego RNA (ncRNA). Ekspresja cANRIL jest skorelowana z ryzykiem miażdżycy tętnic , choroby, w której tętnice stają się twarde. Zaproponowano, że cANRIL może modyfikować INK4 /ARF, co z kolei zwiększa ryzyko miażdżycy. Dalsze badania nad ekspresją cANRIL mogą być potencjalnie wykorzystane do zapobiegania lub leczenia miażdżycy.
- miR-7 odgrywa ważną rolę regulacyjną w kilku nowotworach iw chorobie Parkinsona , która jest chorobą mózgu o nieznanym pochodzeniu. Być może aktywność gąbki CiRS-7 mogłaby pomóc w przeciwdziałaniu aktywności miR-7. Jeśli aktywność gąbek kolistych rzeczywiście może pomóc w zwalczaniu szkodliwej aktywności miRNA, naukowcy będą musieli znaleźć najlepszy sposób wprowadzenia ekspresji gąbek, być może za pośrednictwem transgenu, który jest syntetycznym genem przenoszonym między organizmami. Ważne jest również rozważenie, w jaki sposób transgeny mogą być wyrażane tylko w określonych tkankach lub wyrażane tylko po indukowaniu.
- Stwierdzono, że okrągłe RNA są regulowane przez niedotlenienie, zwłaszcza stwierdzono, że circRNA cZNF292 ma działanie proangiogenne w komórkach śródbłonka.
Okrągłe RNA odgrywają rolę w patogenezie choroby Alzheimera
Dube i wsp. wykazali po raz pierwszy, że koliste RNA w mózgu (circRNA) są częścią zdarzeń patogennych, które prowadzą do choroby Alzheimera , wysuwając hipotezę, że specyficzny circRNA będzie inaczej wyrażany w przypadkach AD w porównaniu z grupą kontrolną i że efekty te można wykryć wcześnie w chorobie. Zoptymalizowali i zweryfikowali nowatorski potok analiz kolistych RNA (circRNA). Przeprowadzili trzyetapowy projekt badania, wykorzystując dane sekwencji RNA mózgu Knight ADRC jako odkrycie (etap 1), wykorzystując dane z góry Synaj jako replikację (etap 2) i metaanalizę (etap 3), aby zidentyfikować najbardziej znaczące circRNA różnie wyrażane w chorobie Alzheimera. Korzystając z jego rurociągu, znaleźli 3547 circRNA, które przeszły rygorystyczną kontrolę jakości w kohorcie Knight ADRC, która obejmuje sekwencję RNA z 13 kontroli i 83 przypadków choroby Alzheimera, oraz 3924 circRNA przeszło rygorystyczną kontrolę jakości w zbiorze danych MSBB. Metaanaliza wyników odkrycia i replikacji ujawniła łącznie 148 circRNA, które były istotnie skorelowane z CDR po korekcie FDR. Ponadto 33 circRNA przeszło rygorystyczną, opartą na genach, wielokrotną korektę testu Bonferroniego 5 × 10-6, w tym circHOMER1 ( P = 2, 21 × 10 -18) i circCDR1-AS ( P = 2, 83 × 10 -8 ), między innymi . Przeprowadzili również dodatkowe analizy, aby wykazać, że ekspresja circRNA była niezależna od postaci liniowej, jak również od proporcji komórek, które mogą zakłócać analizy sekwencji RNA mózgu w badaniach nad chorobą Alzheimera. Przeprowadzili analizy koekspresji wszystkich circRNA wraz z formami liniowymi i stwierdzili, że circRNA, w tym te, które ulegały różnej ekspresji w chorobie Alzheimera w porównaniu z kontrolami, współeksprymowały ze znanymi przyczynowymi genami Alzheimera, takimi jak APP i PSEN1, co wskazuje, że niektóre circRNA są również częścią szlaku przyczynowego. Wykazali również, że ekspresja cirRNA w mózgu wyjaśnia więcej objawów klinicznych choroby Alzheimera niż liczba alleli APOε4, co sugeruje, że można ją wykorzystać jako potencjalny biomarker choroby Alzheimera. Jest to ważne badanie w tej dziedzinie, ponieważ po raz pierwszy circRNA jest oceniane ilościowo i walidowane (za pomocą PCR w czasie rzeczywistym) w próbkach ludzkiego mózgu w skali całego genomu oraz w dużych i dobrze scharakteryzowanych kohortach. Pokazuje również, że te formy RNA prawdopodobnie są zaangażowane w złożone cechy, w tym chorobę Alzheimera, co pomoże zrozumieć zdarzenia biologiczne, które prowadzą do choroby.
Wiroidy jako koliste RNA
Wiroidy to głównie patogeny roślin, które składają się z krótkich odcinków (kilkaset zasad nukleinowych) wysoce komplementarnych, kolistych, jednoniciowych i niekodujących RNA bez płaszcza białkowego. W porównaniu z innymi zakaźnymi patogenami roślin, wiroidy mają niezwykle małe rozmiary, w zakresie od 246 do 467 zasad nukleinowych; składają się zatem z mniej niż 10 000 atomów. Dla porównania, genom najmniejszych znanych wirusów zdolnych do samodzielnego wywołania infekcji ma długość około 2000 zasad nukleinowych.