Odwrócona wakcynologia
Wakcynologia odwrotna to udoskonalenie wakcynologii wykorzystującej bioinformatykę i praktyki farmakologii odwróconej , zapoczątkowane przez Rino Rappuoli i po raz pierwszy zastosowane przeciwko meningokokom grupy serologicznej B. Od tego czasu zastosowano go w kilku innych szczepionkach bakteryjnych. [ wymagany pełny cytat ]
Podejście obliczeniowe
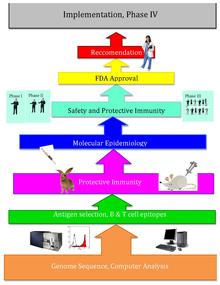
Podstawową ideą wakcynologii odwrotnej jest to, że cały genom patogenu można zbadać przesiewowo przy użyciu metod bioinformatycznych w celu znalezienia genów. Niektóre cechy, pod kątem których monitoruje się geny, mogą wskazywać na antygenowość i obejmują geny kodujące białka o lokalizacji zewnątrzkomórkowej , peptydy sygnałowe i epitopy komórek B. Geny te są filtrowane pod kątem pożądanych cech, które mogłyby stanowić dobre cele szczepionki, takich jak białka błony zewnętrznej . Po zidentyfikowaniu kandydatów są one produkowane syntetycznie i poddawane testom przesiewowym na zwierzęcych modelach zakażenia.
Historia
Po opublikowaniu przez Craiga Ventera genomu pierwszego wolno żyjącego organizmu w 1995 r., pod koniec XX wieku genomy innych mikroorganizmów stały się łatwiej dostępne. Odwrotna wakcynologia, projektowanie szczepionek z wykorzystaniem sekwencjonowanego genomu patogenu, wywodzi się z nowego bogactwa informacji genomicznych, a także z postępu technologicznego. Wakcynologia odwrotna jest znacznie skuteczniejsza niż wakcynologia tradycyjna, która wymaga hodowli dużych ilości specyficznych mikroorganizmów oraz szeroko zakrojonych testów laboratoryjnych na mokro. [ potrzebne źródło ]
W 2000 roku Rino Rappuoli i Instytut J. Craiga Ventera opracowali pierwszą szczepionkę wykorzystującą wakcynologię odwrotną przeciwko meningokokom grupy serologicznej B. Instytut J. Craiga Ventera i inne instytucje kontynuowały następnie prace nad szczepionkami przeciwko Streptococcus A, Streptococcus B, Staphylococcus aureus i Streptococcus pneumoniae.
Wakcynologia odwrotna z meningokokami B
Próby odwrotnej wakcynologii rozpoczęły się od meningokoków B (MenB). Meningokoki B były przyczyną ponad 50% meningokokowego zapalenia opon mózgowo-rdzeniowych, a naukowcom nie udało się stworzyć skutecznej szczepionki przeciwko temu patogenowi ze względu na unikalną strukturę bakterii. Otoczka polisacharydowa tej bakterii jest identyczna z powłoką ludzkiego autoantygenu, ale jej białka powierzchniowe znacznie się różnią; oraz brak informacji o białkach powierzchniowych spowodował, że opracowanie szczepionki było niezwykle trudne. W rezultacie Rino Rappuoli i inni naukowcy zwrócili się w stronę bioinformatyki, aby zaprojektować funkcjonalną szczepionkę.
Rappuoli i inni z Instytutu J. Craiga Ventera jako pierwsi zsekwencjonowali genom MenB. Następnie przeskanowali zsekwencjonowany genom pod kątem potencjalnych antygenów. Znaleźli ponad 600 możliwych antygenów, które zbadano metodą ekspresji w Escherichia coli . W prototypowych szczepionkach zastosowano najbardziej uniwersalne antygeny. Kilka z nich okazało się skutecznych w działaniu u myszy, jednakże same te białka nie oddziaływały skutecznie z ludzkim układem odpornościowym, ponieważ nie wywoływały dobrej odpowiedzi immunologicznej niezbędnej do zapewnienia ochrony. Później przez dodanie pęcherzyków błony zewnętrznej zawierających lipopolisacharydy z oczyszczania pęcherzyków w kulturach Gram-ujemnych. Dodatek tego adiuwanta (wcześniej zidentyfikowanego przy użyciu konwencjonalnych metod wakcynologii) wzmocnił odpowiedź immunologiczną do wymaganego poziomu. Później udowodniono, że szczepionka jest bezpieczna i skuteczna u dorosłych ludzi.
Późniejsze badania wakcynologii odwrotnej
Podczas opracowywania szczepionki MenB naukowcy przyjęli te same metody wakcynologii odwrotnej w przypadku innych patogenów bakteryjnych. Szczepionki Streptococcus A i Streptococcus B były dwiema z pierwszych stworzonych szczepionek odwrotnych. Ponieważ te szczepy bakteryjne indukują przeciwciała reagujące z ludzkimi antygenami, szczepionki przeciwko tym bakteriom nie musiały zawierać homologii z białkami kodowanymi w ludzkim genomie, aby nie powodować reakcji niepożądanych, co uzasadniało potrzebę stosowania szczepionek odwrotnych opartych na genomie.
Później zastosowano szczepionkę odwrotną do opracowania szczepionek przeciwko opornym na antybiotyki Staphylococcus aureus i Streptococcus pneumoniae
Plusy i minusy
Główną zaletą wakcynologii odwrotnej jest szybkie i skuteczne znajdowanie celów szczepionki. Tradycyjne metody mogą zająć dziesięciolecia, aby odkryć patogeny i antygeny, choroby i odporność. Jednakże metoda in silico może być bardzo szybka i pozwolić na identyfikację nowych szczepionek do testów w ciągu zaledwie kilku lat. Wadą jest to, że za pomocą tego procesu można celować wyłącznie w białka. Podczas gdy konwencjonalne podejścia wakcynologiczne mogą znaleźć inne cele biomolekularne, takie jak polisacharydy . [ potrzebne źródło ]
Dostępne oprogramowanie
Chociaż w ciągu ostatnich dziesięciu lat wykorzystywanie technologii bioinformatycznej do opracowywania szczepionek stało się typowe, laboratoria ogólne często nie dysponują zaawansowanym oprogramowaniem, które mogłoby to zrobić. Istnieje jednak coraz większa liczba programów zwiększających dostępność informacji o wakcynologii odwrotnej. NERVE to stosunkowo nowy program do przetwarzania danych. Chociaż należy go pobrać i nie zawiera wszystkich przewidywań epitopów, pomaga zaoszczędzić trochę czasu, łącząc etapy obliczeniowe wakcynologii odwrotnej w jeden program. Vaxign, jeszcze bardziej kompleksowy program, powstał w 2008 roku. Vaxign jest programem internetowym i całkowicie ogólnodostępnym.
Chociaż stwierdzono, że szczepionka Vaxign jest niezwykle dokładna i skuteczna, niektórzy naukowcy nadal korzystają z oprogramowania internetowego RANKPEP do przewidywania wiązań peptydowych. Zarówno Vaxign, jak i RANKPEP wykorzystują PSSM (macierze punktacji specyficzne dla pozycji) podczas analizy sekwencji białek lub dopasowań sekwencji.
Projekty bioinformatyki wspomaganej komputerowo stają się niezwykle popularne, ponieważ pomagają w prowadzeniu eksperymentów laboratoryjnych.
Inne osiągnięcia wynikają z wakcynologii odwrotnej i bioinformatyki
- Wakcynologia odwrotna spowodowała zwiększone skupienie się na biologii patogenów.
- Odwrotna wakcynologia doprowadziła do odkrycia pilusów u patogenów Gram-dodatnich, takich jak paciorkowce A, paciorkowce B i pneumokoki. Wcześniej uważano, że wszystkie bakterie Gram-dodatnie nie mają pilusów.
- Odwrotna wakcynologia doprowadziła również do odkrycia białka wiążącego czynnik G u meningokoków, które wiąże się z czynnikiem dopełniacza H u ludzi. Wiązanie z czynnikiem dopełniacza H umożliwia meningokokom wzrost w ludzkiej krwi, blokując jednocześnie alternatywne ścieżki. Model ten nie pasuje do wielu gatunków zwierząt, które nie mają tego samego czynnika dopełniacza H co ludzie, co wskazuje na różnicowanie meningokoków między różnymi gatunkami.
- ^ Pizza, M .; Rappuoli, R.; Scarlato, V.; Masignani, V.; Giuliani, M.; Aricò, B. (10 marca 2000). „Identyfikacja kandydatów na szczepionki przeciwko meningokokom grupy serologicznej B poprzez sekwencjonowanie całego genomu” . Nauka . 287 (5459): 1816–1820. Bibcode : 2000Sci...287.1816. . doi : 10.1126/science.287.5459.1816 . PMID 10710308 .
- Bibliografia _ Reverse Vaccinology Current Opinion in Microbiology 2000, 3:445–450
- ^ CH Woelk i in. „Udoskonalanie wakcynologii odwrotnej dzięki podejściu do uczenia maszynowego”. Szczepionka 29, nr. 45 (nd): 8156-8164. Science Citation Index, EBSCOhost (dostęp: 30 września 2012).
- ^ Michalik, Marcin; Djahanshiri, Bardia; Leo, Jack C.; Linke, Dirk (2016), Thomas, Sunil (red.), „Odwrotna wakcynologia: ścieżka od genomów i przewidywań epitopów do dostosowanych szczepionek rekombinowanych” , Vaccine Design: Methods and Protocols: Tom 1: Vaccines for Human Diseases , Nowy Jork, Nowy Jork: Springer Publishing ; Humana Press , tom. 1403, s. 87–106, doi : 10.1007/978-1-4939-3387-7_4 , ISBN 978-1-4939-3387-7 , PMID 27076126
- ^ a b c d e f g h Alessandro S, Rino R. Recenzja: Wakcynologia odwrotna: rozwój szczepionek w erze genomiki. Odporność [serial online]. nd;33:530-541. Dostępne w: ScienceDirect , Ipswich, MA. Dostęp: 30 września 2012 r.
- ^ Rappuoli, R. i A. Aderem. 2011. Wizja 2020 dotycząca szczepionek przeciwko HIV, gruźlicy i malarii. Natura 473: 463.
- ^ He Y, Xiang Z, Mobley H. Vaxign: pierwszy internetowy program do projektowania szczepionek dla wakcynologii odwrotnej i zastosowań do opracowywania szczepionek. Journal of Biomedicine & Biotechnology [serial online]. 2010; Dostępne w: CINAHL Plus z pełnym tekstem, Ipswich, MA. Dostęp: 30 września 2012 r.
- ^ Reche PA, Glutting JP i Reinherz EL. Przewidywanie peptydów wiążących MHC klasy I przy użyciu motywów profilu. Human Immunology 63, 701-709 (2002).
- ^ Sandro V, Jennifer LG, Francesco F i in. Recenzja: Biotechnologia wspomagana komputerowo: od immunoinformatyki po wakcynologię odwrotną. Trendy w biotechnologii [serial online]. nd;26:190-200. Dostępne w: ScienceDirect, Ipswich, MA. Dostęp: 30 września 2012 r.