Topienie w wysokiej rozdzielczości
Analiza topnienia w wysokiej rozdzielczości ( HRM ) jest potężną techniką w biologii molekularnej do wykrywania mutacji , polimorfizmów i różnic epigenetycznych w próbkach dwuniciowego DNA . Został odkryty i opracowany przez Idaho Technology i University of Utah. Ma zalety w stosunku do innych genotypowania , a mianowicie:
- Jest to opłacalne w porównaniu z innymi technologiami genotypowania , takimi jak sekwencjonowanie i typowanie TaqMan SNP. Dzięki temu idealnie nadaje się do projektów genotypowania na dużą skalę.
- Jest szybki i wydajny, dzięki czemu może szybko dokładnie genotypować wiele próbek.
- To jest proste. Przy dobrej jakości teście HRM, zaawansowane genotypowanie może być wykonywane przez osoby niebędące genetykami w każdym laboratorium z dostępem do maszyny do PCR w czasie rzeczywistym obsługującej HRM.
metoda
Analiza HRM jest przeprowadzana na próbkach dwuniciowego DNA. Zazwyczaj użytkownik stosuje reakcję łańcuchową polimerazy (PCR) przed analizą HRM w celu amplifikacji regionu DNA, w którym znajduje się mutacja będąca przedmiotem jego zainteresowania. W probówce z próbką znajduje się teraz wiele kopii interesującego regionu DNA. Ten amplifikowany region jest znany jako amplikon. Po procesie PCR rozpoczyna się analiza HRM. Proces ten polega po prostu na precyzyjnym ogrzaniu DNA amplikonu od około 50˚C do około 95˚C. W pewnym momencie tego procesu temperatura topnienia amplikonu zostaje osiągnięta i dwie nici DNA rozdzielają się lub „stopią”.
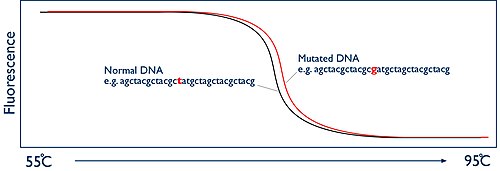
Kluczem do HRM jest monitorowanie tego rozdzielenia pasm w czasie rzeczywistym. Osiąga się to za pomocą barwnika fluorescencyjnego. Barwniki stosowane w HRM są znane jako barwniki interkalujące i mają unikalną właściwość. Wiążą się specyficznie z dwuniciowym DNA, a po związaniu jasno fluoryzują. W przypadku braku dwuniciowego DNA nie mają nic do wiązania i fluoryzują tylko na niskim poziomie. Na początku analizy HRM w próbce występuje wysoki poziom fluorescencji ze względu na miliardy kopii amplikonu. Ale gdy próbka jest podgrzewana i dwie nici DNA topią się, obecność dwuniciowego DNA zmniejsza się, a tym samym zmniejsza się fluorescencja. Maszyna HRM ma kamerę, która obserwuje ten proces, mierząc fluorescencję. Następnie maszyna po prostu wykreśla te dane jako wykres zwany krzywą topnienia, pokazujący poziom fluorescencji w funkcji temperatury:
Porównanie krzywych topnienia
Temperatura topnienia amplikonu, w której dwie nici DNA rozdzielają się, jest całkowicie przewidywalna. Jest to zależne od sekwencji zasad DNA. Jeśli porównujesz dwie próbki od dwóch różnych osób, powinny one dawać dokładnie ten sam kształt krzywej topnienia. Jeśli jednak jedna osoba ma mutację w regionie DNA, który zamplifikowałeś, zmieni to temperaturę, w której nici DNA topią się. Więc teraz dwie krzywe topnienia wyglądają inaczej. Różnica może być niewielka, być może ułamek stopnia, ale ponieważ maszyna HRM ma możliwość monitorowania tego procesu w „wysokiej rozdzielczości”, możliwe jest dokładne udokumentowanie tych zmian, a tym samym określenie, czy mutacja jest obecna, czy nie. .
Typ dziki, heterozygota czy homozygota?
Sprawy stają się nieco bardziej skomplikowane, ponieważ organizmy zawierają dwie ( lub więcej ) kopie każdego genu, znane jako dwa allele . Tak więc, jeśli próbka jest pobierana od pacjenta i amplifikowana za pomocą PCR, amplifikowane są obie kopie interesującego nas regionu DNA (alleli). Jeśli więc szukamy mutacji, mamy teraz trzy możliwości:
- Żaden allel nie zawiera mutacji
- Jeden lub inny allel zawiera mutację
- Oba allele zawierają mutację.
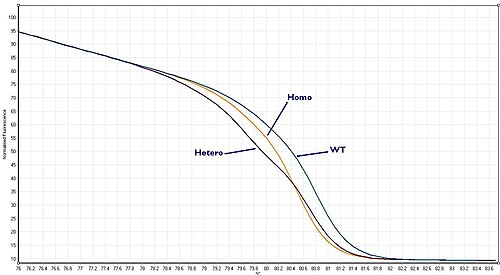
Te trzy scenariusze są znane odpowiednio jako „typ dziki”, „heterozygota” lub „homozygota”. Każdy daje krzywą topnienia, która jest nieco inna. Dzięki wysokiej jakości testowi HRM możliwe jest rozróżnienie wszystkich trzech scenariuszy.
Homozygotyczne warianty alleliczne można scharakteryzować przez przesunięcie temperatury na wynikowej krzywej topnienia uzyskanej w analizie HRM. Dla porównania, heterozygoty charakteryzują się zmianami kształtu krzywej topnienia. Wynika to z niedopasowania par zasad generowanego w wyniku zdestabilizowanego hybrydyzacji heterodupleksów między niciami typu dzikiego i wariantów. Różnice te można łatwo zobaczyć na wynikowej krzywej topnienia, a różnice w profilu topnienia między różnymi genotypami można wzmocnić wizualnie poprzez wygenerowanie krzywej różnicowej
Aplikacje
Typowanie SNP/wykrywanie mutacji punktowych
Konwencjonalne metody typowania SNP są zazwyczaj czasochłonne i kosztowne, wymagają multipleksowania kilku testów opartych na sondach lub użycia mikromacierzy DNA. HRM jest bardziej opłacalny i ogranicza konieczność projektowania wielu par starterów oraz konieczność zakupu drogich sond. Metoda HRM została z powodzeniem zastosowana do wykrycia pojedynczej zamiany G na A w genie Vssc (Voltage Sensitive Sodium Channel), która nadaje oporność na roztoczobójczy permetrynę u świerzbu świerzbowca. Ta mutacja powoduje zmianę kodowania białka (G1535D). Analiza roztoczy świerzbu pobranych z populacji podejrzanych o wrażliwość i tolerancję na permetrynę metodą HRM wykazała różne profile topnienia. Zaobserwowano, że amplikony z wrażliwych roztoczy mają wyższą temperaturę topnienia w porównaniu z tolerującymi roztoczami, zgodnie z oczekiwaniami na podstawie wyższej termostabilności pary zasad GC
W dziedzinie bardziej istotnej dla diagnostyki klinicznej wykazano, że HRM zasadniczo nadaje się do wykrywania mutacji w genach podatności na raka piersi BRCA1 i BRCA2. W tych genach zidentyfikowano ponad 400 mutacji.
Sekwencjonowanie genów jest złotym standardem identyfikacji mutacji. Sekwencjonowanie jest czasochłonne i pracochłonne i często jest poprzedzone technikami stosowanymi do identyfikacji heterodupleksowego DNA, które następnie dodatkowo wzmacniają te problemy. HRM oferuje szybszą i wygodniejszą metodę oceny obecności mutacji w zamkniętej probówce i daje wynik, który można dalej badać, jeśli jest to interesujące. W badaniu przeprowadzonym przez Scotta i in. w 2006 roku do oceny metodologii HRM wykorzystano 3 linie komórkowe z różnymi mutacjami BRCA. Stwierdzono, że profile topnienia otrzymanych produktów PCR można wykorzystać do rozróżnienia obecności lub braku mutacji w amplikonie. Podobnie w 2007 roku Krypuy i in. wykazali, że staranne zaprojektowanie testów HRM (w odniesieniu do umieszczenia startera) można z powodzeniem zastosować do wykrywania mutacji w genie TP53, który koduje białko supresorowe guza p53 w próbkach klinicznych raka piersi i jajnika. Oba te badania podkreśliły fakt, że zmiany w profilu topnienia mogą mieć postać przesunięcia temperatury topnienia lub oczywistej różnicy w kształcie krzywej topnienia. Oba te parametry są funkcją sekwencji amplikonu. Konsensus jest taki, że HRM jest efektywną kosztowo metodą, którą można zastosować jako wstępne badanie przesiewowe próbek podejrzanych o noszenie polimorfizmów lub mutacji. Zmniejszyłoby to liczbę próbek, które należy dalej badać przy użyciu bardziej konwencjonalnych metod.
Badanie zygotyczności
Obecnie istnieje wiele metod określania zygotyczności status genu w określonym locus. Metody te obejmują zastosowanie PCR ze specjalnie zaprojektowanymi sondami do wykrywania wariantów genów (najprostszym przypadkiem jest typowanie SNP). W przypadkach, gdy implikowane są dłuższe odcinki zmienności, może być wymagana analiza amplikonów po PCR. Można mierzyć zmiany w profilach restrykcyjnych, elektroforetycznych i chromatograficznych enzymów. Metody te są zwykle bardziej czasochłonne i zwiększają ryzyko kontaminacji amplikonu w laboratorium, ze względu na konieczność pracy z dużymi stężeniami amplikonów w laboratorium po PCR. Zastosowanie HRM skraca czas analizy i ryzyko kontaminacji. HRM jest bardziej opłacalnym rozwiązaniem, a element o wysokiej rozdzielczości pozwala nie tylko na określenie homo i heterozygotyczność , rozwiązuje również informacje o typie homo i heterozygotyczności, przy czym różne warianty genów powodują różne kształty krzywej topnienia. Badanie przeprowadzone przez Gundry'ego i in. 2003, wykazali, że znakowanie fluorescencyjne jednego startera (w parze) okazało się korzystniejsze niż stosowanie barwnika interkalującego, takiego jak SYBR green I . Jednak poczyniono postępy w opracowywaniu i stosowaniu ulepszonych barwników interkalujących, które zmniejszają problem hamowania PCR i obawy związane z nienasycającą interkalacją barwnika.
Epigenetyka
Metodologia HRM została również wykorzystana do zapewnienia wiarygodnej analizy stanu metylacji DNA. Ma to znaczenie, ponieważ zmiany statusu metylacji genów supresorowych nowotworów, genów regulujących apoptozę i naprawę DNA, są charakterystyczne dla nowotworów i mają również implikacje dla odpowiedzi na chemioterapię. Na przykład pacjenci z rakiem mogą być bardziej wrażliwi na leczenie środkami alkilującymi DNA, jeśli promotor genu naprawy DNA MGMT pacjenta jest metylowany. W badaniu, w którym przetestowano stan metylacji MGMT promotora na 19 próbkach jelita grubego, stwierdzono, że 8 próbek było metylowanych. W innym badaniu porównano moc predykcyjną MGMT u 83 pacjentów z glejakiem o wysokim stopniu złośliwości uzyskaną za pomocą MSP , pirosekwencjonowania i HRM. Stwierdzono, że metoda HRM jest co najmniej równoważna z pirosekwencjonowaniem w ilościowym określaniu poziomu metylacji.
Zmetylowany DNA można poddać obróbce za pomocą wodorosiarczynu, który przekształca niemetylowane cytozyny w uracyl . Dlatego produkty PCR otrzymane z matrycy, która pierwotnie była niemetylowana, będą miały niższą temperaturę topnienia niż te pochodzące z matrycy metylowanej. HRM oferuje również możliwość określenia proporcji metylacji w danej próbce, porównując ją z krzywą wzorcową, która jest generowana przez zmieszanie różnych proporcji zmetylowanego i niemetylowanego DNA. Może to dostarczyć informacji dotyczących stopnia metylacji, jaki może mieć guz, a tym samym wskazać charakter guza i jak daleko odbiega on od tego, co jest „normalne”.
HRM jest również praktycznie przydatny do stosowania w diagnostyce, ze względu na jego zdolność do przystosowania się do wysokowydajnych badań przesiewowych, a także minimalizuje możliwość rozprzestrzeniania się amplikonu i kontaminacji w laboratorium, dzięki formatowi zamkniętej probówki.
Barwniki interkalujące
Aby śledzić przejście dsDNA (dwuniciowy) do ssDNA (jednoniciowy), stosuje się barwniki interkalujące. Barwniki te wykazują zróżnicowaną emisję fluorescencji zależną od ich połączenia z dwuniciowym lub jednoniciowym DNA. SYBR Green I jest barwnikiem pierwszej generacji dla HRM. Fluorezyzuje po włączeniu do dsDNA, a nie ssDNA. Ponieważ może hamować PCR w wysokich stężeniach, jest stosowany w stężeniach poniżej nasycenia. Ostatnio niektórzy badacze odradzają stosowanie SYBR Green I w HRM, twierdząc, że konieczne są istotne modyfikacje protokołu. Dzieje się tak, ponieważ sugeruje się, że brak dokładności może wynikać z „przeskoków barwnika”, w których barwnik ze stopionego dupleksu może zostać ponownie włączony do regionów dsDNA, które jeszcze się nie stopiły. Nowe barwniki nasycające, takie jak LC Green i LC Green Plus, ResoLight, EvaGreen, Chromofy i SYTO 9 są dostępne na rynku i są z powodzeniem stosowane w HRM. Jednak niektóre grupy z powodzeniem stosowały SYBR Green I do HRM z instrumentami Corbett Rotorgene i zalecają stosowanie SYBR Green I do zastosowań HRM.
Projektowanie eksperymentów topnienia o wysokiej rozdzielczości
Testy topnienia o wysokiej rozdzielczości zazwyczaj obejmują amplifikację qPCR, po której następuje zebranie krzywej topnienia przy użyciu barwnika fluorescencyjnego. Ze względu na czułość analizy topnienia w wysokiej rozdzielczości konieczne jest staranne rozważenie warunków cykli PCR, jakości matrycy DNA i parametrów krzywej topnienia. Aby uzyskać dokładne i powtarzalne wyniki, należy zoptymalizować warunki cykli termicznych PCR, aby zapewnić amplifikację pożądanego regionu DNA z wysoką specyficznością i minimalnym odchyleniem między wariantami sekwencji. Krzywa topnienia jest zwykle wykonywana w szerokim zakresie temperatur w małych (~ 0,3 ° C) przyrostach, które są wystarczająco długie (~ 10 sekund), aby DNA osiągnęło równowagę na każdym etapie temperatury.
Oprócz typowych rozważań dotyczących projektowania starterów , projektowanie starterów do testów topnienia o wysokiej rozdzielczości obejmuje maksymalizację różnic termodynamicznych między produktami PCR należącymi do różnych genotypów. Mniejsze amplikony na ogół powodują większe wahania temperatury topnienia niż dłuższe amplikony, ale zmienności nie można przewidzieć naocznie. Z tego powodu bardzo ważne jest dokładne przewidywanie krzywej topnienia produktów PCR podczas projektowania starterów, które pozwolą rozróżnić warianty sekwencji. Oprogramowanie specjalistyczne, takie jak uMelt i DesignSignatures , są dostępne, aby pomóc zaprojektować startery, które zmaksymalizują zmienność krzywej topnienia, szczególnie dla testów topnienia o wysokiej rozdzielczości.