kinemaż

Kinematage (skrót od obrazu kinetycznego ) to interaktywna graficzna ilustracja naukowa. Często jest używany do wizualizacji cząsteczek , zwłaszcza białek , chociaż może również reprezentować inne typy danych trójwymiarowych (takie jak figury geometryczne, sieci społecznościowe lub czworościany składu zasad RNA ). System kinemage został zaprojektowany w celu optymalizacji łatwości użytkowania, interaktywnej wydajności oraz postrzegania i przekazywania szczegółowych informacji 3D. Informacje o kinemage są przechowywane w pliku tekstowym, czytelnym dla człowieka i maszyny, który opisuje hierarchię obiektów wyświetlanych i ich właściwości oraz zawiera opcjonalny tekst objaśniający. Format kinemage jest zdefiniowany chemiczny typ MIME „chemical/x-kinemage” z rozszerzeniem pliku „.kin”.
Wczesna historia
Kinemages zostały po raz pierwszy opracowane przez Davida Richardsona z Duke University School of Medicine dla czasopisma Protein Science , wydawanego przez Protein Society , którego premiera miała miejsce w styczniu 1992 r. Przez pierwsze 5 lat (1992–1996) każdy numer Protein Science zawierał dodatek na dyskietce zawierający interaktywna grafika komputerowa kinemage 3D do zilustrowania wielu artykułów, a także oprogramowanie Mage ( wieloplatformowe , bezpłatne, open-source ), aby je wyświetlić; materiał uzupełniający kinemage jest nadal dostępny na stronie internetowej czasopisma. Mage i RasMol były pierwszymi szeroko stosowanymi do grafiki makromolekularnej obsługującymi interaktywne wyświetlanie na komputerach osobistych . Kinemages są wykorzystywane do nauczania i jako dodatki do podręczników, do indywidualnej eksploracji i analizy struktur makrocząsteczkowych.

Zastosowania badawcze
Niedawno, wraz z dostępnością znacznie szerszej gamy innych narzędzi do grafiki molekularnej , użycie obrazów kinematycznych do prezentacji zostało wyprzedzone przez szeroką gamę zastosowań badawczych, wraz z nowymi funkcjami wyświetlania i rozwojem oprogramowania, które generuje dane wyjściowe w formacie kinemage z inne rodzaje obliczeń molekularnych. Analiza kontaktu wszystkich atomów dodaje i optymalizuje wyraźne atomy wodoru, a następnie wykorzystuje plamy powierzchni kropki do wyświetlenia wiązania wodorowego , van der Waalsa i zderzenia sterycznego oddziaływania między atomami. Wyniki można wykorzystać wizualnie (w obrazach kinematycznych) i ilościowo do szczegółowej analizy interakcji między powierzchniami cząsteczkowymi, najszerzej w celu walidacji i ulepszenia modeli molekularnych na podstawie danych z eksperymentalnej krystalografii rentgenowskiej . Zarówno Mage, jak i KiNG (patrz poniżej) zostały udoskonalone pod kątem wyświetlania danych kinematycznych w więcej niż 3 wymiarach (przechodzenie między widokami w różnych projekcjach 3-D, kolorowanie i wybieranie potencjalnych skupisk punktów danych oraz przełączanie do równoległych współrzędnych reprezentacji), używane na przykład do definiowania klastrów korzystnych konformacji szkieletu RNA w 7-wymiarowej przestrzeni dwuściennych kątów szkieletu między jedną a następną rybozą.
Korzystanie z sieci internetowej
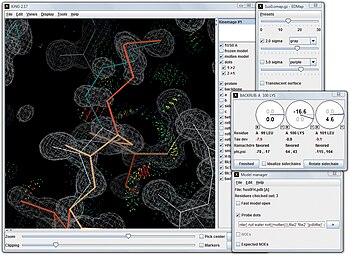
KiNG to przeglądarka kinemage typu open source, napisana w języku programowania Java przez Iana Davisa i Vincenta Chena, która może działać interaktywnie albo samodzielnie na komputerze użytkownika bez połączenia sieciowego, albo jako usługa internetowa na stronie internetowej . Interaktywny charakter kinemaży jest ich podstawowym celem i atrybutem. Aby docenić ich naturę, demonstracja KiNG w przeglądarce zawiera dwa przykłady, które można przesuwać w 3D, oraz instrukcje, jak osadzić kinemaż na stronie internetowej. Poniższy rysunek pokazuje, że KiNG jest używany do przebudowy łańcucha bocznego lizyny w strukturze krystalicznej o wysokiej rozdzielczości. KiNG jest jedną z przeglądarek znajdujących się na każdej stronie struktury w witrynie Protein Data Bank i wyświetla wyniki walidacji w 3D na stronie MolProbity. Kinemagazy można również wyświetlać w trybie immersyjnym rzeczywistości wirtualnej z oprogramowaniem KinImmerse typu open source. Całe oprogramowanie kinemage do wyświetlania i kontaktu w całości z atomami jest dostępne bezpłatnie na stronie internetowej kinemage .
Zobacz też
Linki zewnętrzne
- Oficjalna strona internetowa , oryginał Duke University, z przykładami i oprogramowaniem
- przykład kinemage w przeglądarce
- Bank danych białek RCSB
- MolProbity: walidacja struktury z kinematyką KiNG on-line