5S rybosomalnego RNA
| 5S rybosomalny RNA | |
|---|---|
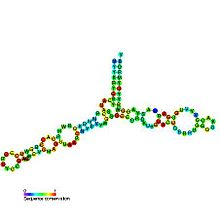 Przewidywana struktura drugorzędowa i konserwacja sekwencji rybosomalnego RNA 5S
| |
| Identyfikatory | |
| Symbol | 5S_rRNA |
| Rfam | RF00001 CL00113 |
| Inne dane | |
| typ RNA | gen ; rRNA |
| Domeny | eukariota ; bakterie ; Archeony |
| IŚĆ | GO:0005840 GO:0003735 |
| WIĘC | SO: 0000652 |
| Struktury PDB | PDBe |
Rybosomalny RNA 5S ( 5S rRNA ) jest cząsteczką rybosomalnego RNA o długości około 120 nukleotydów i masie 40 kDa . Jest strukturalnym i funkcjonalnym składnikiem dużej podjednostki rybosomu we wszystkich domenach życia ( bakterie , archeony i eukarionty ), z wyjątkiem mitochondrialnych rybosomów grzybów i zwierząt . Oznaczenie 5S odnosi się do sedymentacji cząsteczki prędkość w ultrawirówce, która jest mierzona w jednostkach Svedberga (S).

Biosynteza
U prokariotów gen 5S rRNA jest zwykle zlokalizowany w operonach rRNA poniżej małej i dużej podjednostki rRNA i podlega transkrypcji do policistronowego prekursora . Cechą charakterystyczną genomów jądrowych eukariotów jest występowanie wielu kopii genów 5S rRNA (5S rDNA) skupionych w tandemowych powtórzeniach, przy czym liczba kopii różni się w zależności od gatunku. Eukariotyczny 5S rRNA jest syntetyzowany przez polimerazę RNA III , podczas gdy inne eukariotyczne rRNA są odcinane od prekursora 45S transkrybowanego przez polimerazę RNA I. W oocytach Xenopus , wykazano, że palce 4–7 czynnika transkrypcyjnego TFIIIA dziewięciu palców cynkowych mogą wiązać się z centralnym regionem 5S RNA. Wiązanie między 5S rRNA i TFIIIA służy zarówno do tłumienia dalszej transkrypcji genu 5S RNA, jak i do stabilizacji transkryptu 5S RNA, dopóki nie będzie potrzebny do złożenia rybosomu.
Struktura
Struktura drugorzędowa 5S rRNA składa się z pięciu helis (oznaczonych cyframi rzymskimi IV ), czterech pętli (BE) i jednego zawiasu (A), które razem tworzą strukturę podobną do litery Y. Pętle C i D to końcowe spinki do włosów, a pętle B i E są wewnętrzne. Według badań filogenetycznych helisy I i III są prawdopodobnie przodkami. Helix III zawiera dwie wysoce konserwatywne adenozyny. Uważa się, że Helix V, ze swoją strukturą spinki do włosów, oddziałuje z TFIIIA.
Lokalizacja w rybosomie

Przy użyciu różnych technik molekularnych, w tym mikroskopii immunoelektronowej , mikroskopii krioelektronowej , chemicznego sieciowania międzycząsteczkowego i krystalografii rentgenowskiej , położenie 5S rRNA w dużej podjednostce rybosomu zostało określone z dużą precyzją. U bakterii i archeonów sama duża podjednostka rybosomu (LSU) składa się z dwóch ugrupowań RNA, 5S rRNA i innego większego RNA znanego jako 23S rRNA , wraz z licznymi powiązanymi białkami.
U eukariontów LSU zawiera rRNA 5S, 5,8S i 28S oraz jeszcze więcej białek. Struktura LSU w 3 wymiarach przedstawia jedną stosunkowo gładką powierzchnię i przeciwległą powierzchnię z trzema występami, zwłaszcza wypukłością L1, wypukłością środkową (CP) i łodygą L7/L12. Wypukłość L1 i łodyga L7/L12 są rozmieszczone bocznie wokół CP. 5S rRNA znajduje się w CP i uczestniczy w tworzeniu i strukturze tej projekcji. Inne główne składniki centralnej wypukłości obejmują 23S rRNA (lub alternatywnie 28S u eukariontów) i kilka białek, w tym L5, L18, L25 i L27.
Funkcje rybosomów
Dokładna funkcja 5S rRNA nie jest jeszcze jasna. U Escherichia coli delecje genu 5S rRNA zmniejszają szybkość syntezy białek i mają bardziej szkodliwy wpływ na sprawność komórek niż delecje porównywalnej liczby kopii innych ( 16S i 23S ) geny rRNA. Badania krystalograficzne wskazują, że białka wiążące 5S rRNA i inne białka centralnej wypukłości LSU odgrywają rolę w wiązaniu tRNA. Również topograficzna i fizyczna bliskość między 5S rRNA i 23S rRNA, które tworzą transferazę peptydylową i centrum asocjacyjne z GTPazą, sugeruje, że 5S rRNA działa jako mediator między dwoma centrami funkcjonalnymi rybosomu, tworząc wraz z wiązaniem 5S rRNA białka i inne składniki centralnej wypukłości, mostki między podjednostkami i miejsca wiązania tRNA.
Role w składaniu rybosomów
U eukariontów cytozolowy rybosom składa się z czterech rRNA i ponad 80 białek. Po transkrypcji końce 3' 5S rRNA są przetwarzane przez egzonukleazy Rex1p, Rex2p i Rex3p. Podjednostki 60S i 40S są eksportowane z jądra do cytoplazmy, gdzie łączą się, tworząc dojrzały i translacyjny -kompetentny rybosom 80S. Kiedy dokładnie 5S rRNA jest zintegrowany z rybosomem, pozostaje kontrowersyjne, ale ogólnie przyjmuje się, że 5S rRNA jest włączony do cząsteczki 90S, która jest prekursorem cząsteczki 60S, jako część małego, niezależnego od rybosomu kompleksu RNP utworzonego przez 5S rRNA i białko rybosomalne L5.
Interakcje z białkami
Poniżej wymieniono kilka ważnych białek, które oddziałują z 5S rRNA.
Białko
Oddziaływanie 5S rRNA z białkiem La zapobiega degradacji RNA przez egzonukleazy w komórce. Białko La znajduje się w jądrze wszystkich organizmów eukariotycznych i wiąże się z kilkoma typami RNA transkrybowanymi przez RNA pol III. Białko La oddziałuje z tymi RNA (w tym 5S rRNA) poprzez ich szlak oligourydynowy 3', wspomagając stabilność i fałdowanie RNA.
białko L5
W komórkach eukariotycznych rybosomalne białko L5 wiąże i stabilizuje 5S rRNA, tworząc przedrybosomalną cząsteczkę rybonukleoproteiny (RNP), która znajduje się zarówno w cytozolu, jak i jądrze. Niedobór L5 uniemożliwia transport 5S rRNA do jądra i powoduje zmniejszenie składania rybosomów.
Inne białka rybosomalne
U prokariotów 5S rRNA wiąże się z białkami rybosomalnymi L5, L18 i L25, podczas gdy u eukariontów 5S rRNA wiąże się tylko z rybosomalnym białkiem L5 . U T. brucei , czynnika sprawczego śpiączki , 5S rRNA oddziałuje z dwoma blisko spokrewnionymi białkami wiążącymi RNA, P34 i P37, których utrata skutkuje niższym globalnym poziomem 5S rRNA.
Obecność w rybosomach organelli
| identyfikatory | |
|---|---|
| 5S rRNA zakodowane w genomie mitochondrialnym | |
| Symbol | mtPerm-5S |
| Rfam | RF02547 CL00113 |
| Inne dane | |
| typ RNA | gen ; rRNA |
| Domeny | eukariota ; |
| IŚĆ | GO:0005840 GO:0003735 |
| WIĘC | SO: 0000652 |
| Struktury PDB | PDBe |
Mechanizmy translacyjne mitochondriów i plastydów (organelli pochodzenia bakteryjnego endosymbiotycznego) oraz ich bakteryjnych krewnych mają wiele wspólnych cech, ale wykazują również wyraźne różnice. Genomy organelli bez wyjątku kodują rRNA SSU i LSU, jednak rozmieszczenie genów 5S rRNA ( rrn5 ) jest najbardziej nierówne. Rrn5 jest łatwo identyfikowalny i powszechny w genomach większości plastydów. W przeciwieństwie do mitochondrialnego rrn5 początkowo wydawał się być ograniczony do roślin i niewielkiej liczby protistów. Dodatkowe, bardziej rozbieżne rRNA organelli 5S zidentyfikowano tylko za pomocą wyspecjalizowanych modeli kowariancji, które zawierają informacje o wyraźnym odchyleniu składu sekwencji i zmienności strukturalnej. Ta analiza wskazała dodatkowe geny 5S rRNA nie tylko w genomach mitochondrialnych większości protistów , ale także w genomach niektórych apikoplastów (niefotosyntetycznych plastydów patogennych pierwotniaków, takich jak Toxoplasma gondii i Eimeria tenella ).
Mitochondrialne rRNA 5S większości stramenopile obejmują największą różnorodność struktur drugorzędowych. Permutowane mitochondrialne 5S rRNA w brązowych algach reprezentują najbardziej niekonwencjonalny przypadek, w którym zamykająca helisa I, która w przeciwnym razie łączy ze sobą końce 5' i 3' cząsteczki, jest zastąpiona (zamkniętą) szpilką do włosów, co skutkuje otwartym trójdrożnym połączeniem.
Obecne dowody wskazują, że mitochondrialne DNA tylko kilku grup, zwłaszcza zwierząt , grzybów , pęcherzyków płucnych i euglenozoanów nie ma tego genu. Centralna wypukłość , w przeciwnym razie zajmowana przez 5S rRNA i związane z nim białka (patrz Figura 2 ), została przebudowana na różne sposoby. W grzybowych rybosomach mitochondrialnych 5S rRNA jest zastępowane sekwencjami ekspansji rRNA LSU. W kinetoplastydach (euglenozoans), centralna wypukłość jest wykonana w całości z ewolucyjnie nowych mitochondrialnych białek rybosomalnych. Wreszcie, zwierzęce rybosomy mitochondrialne dokooptowały specyficzne mitochondrialne tRNA (Val u kręgowców), aby zastąpić brakujący rRNA 5S.
Zobacz też
Linki zewnętrzne
- Strona dla rybosomalnego RNA 5S w Rfam
- 5SData zarchiwizowane 2010-04-27 w Wayback Machine
- 5S + rybosomal + RNA w US National Library of Medicine Medical Subject Headings (MeSH)
- 5S_rRNA w przeglądarce genomu UCSC .
- Halococcus morrhuae (archaebacterium) 5S rRNA
