Anduril (silnik przepływu pracy)
 | |
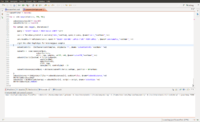 Przepływ pracy Anduril w Eclipse
| |
| Deweloperzy | Laboratorium Biologii Systemów Uniwersytetu w Helsinkach |
|---|---|
| Pierwsze wydanie | 1 lipca 2010 |
| Wersja stabilna | 2.0.0 ( 01.07.2016 ) / 1 lipca 2016
|
| Magazyn | |
| Napisane w | Jawa |
| System operacyjny | Linux , Microsoft Windows , Mac OS X |
| Typ | Silnik przepływu pracy |
| Licencja | GPL (v.1.x), BSD (v.2.x) |
| Strona internetowa | |
Anduril to oparta na komponentach struktura przepływu pracy typu open source do analizy danych naukowych, opracowana w Laboratorium Biologii Systemów Uniwersytetu Helsińskiego .
Anduril został zaprojektowany, aby umożliwić systematyczną, elastyczną i wydajną analizę danych, szczególnie w zakresie wysokowydajnych eksperymentów w badaniach biomedycznych. System przepływu pracy zapewnia obecnie komponenty do kilku rodzajów analiz, takich jak sekwencjonowanie , ekspresja genów , SNP , ChIP-on-chip , porównawcza hybrydyzacja genomowa i analiza mikromacierzy eksonów, a także cytometria i analiza obrazowania komórek .
Architektura i funkcje
Przepływ pracy to seria etapów przetwarzania połączonych ze sobą w taki sposób, że dane wyjściowe jednego kroku są używane jako dane wejściowe innego. Etapy przetwarzania realizują zadania analizy danych, takie jak import danych, testy statystyczne i generowanie raportów. W Anduril etapy przetwarzania są realizowane przy użyciu komponentów, które są wykonywalnym kodem wielokrotnego użytku, który można napisać w dowolnym języku programowania. Komponenty są połączone razem w przepływ pracy lub sieć komponentów, która jest wykonywana przez silnik przepływu pracy Anduril. Konfiguracja przepływu pracy odbywa się przy użyciu prostego, ale wydajnego języka skryptowego AndurilScript. Konfigurację i wykonywanie przepływu pracy można wykonać z Eclipse , popularnego uniwersalnego interfejsu GUI lub z wiersza poleceń.
Rdzeń silnika Anduril jest napisany w Javie, a komponenty są napisane w różnych językach programowania, w tym Java, R , MATLAB , Lua , Perl i Python . Komponenty mogą być również zależne od bibliotek innych firm, takich jak Bioconductor . Dostarczane są komponenty do obrazowania komórek i analizy mikromacierzy, ale użytkownicy mogą zaimplementować dodatkowe komponenty. Rdzeń Anduril został przetestowany na systemach Linux i Windows.
Anduril 1.0: język AndurilScript
Hello world w AndurilScript jest po prostu
std . echo ( "Witaj świecie!" )
Komentowanie jest zgodne ze składnią języka Java:
// Prosty komentarz /* Kolejny prosty komentarz */ /** Opis, który zostanie zawarty w opisie komponentu */
Komponenty są wywoływane przez przypisanie ich wywołań do nazwanych instancji komponentów. Nazwy nie mogą być ponownie używane w ramach jednego przepływu pracy. Istnieją specjalne komponenty dla plików wejściowych, które zawierają pliki zewnętrzne do skryptu. Obsługiwane typy atomowe to liczby całkowite, zmiennoprzecinkowe, logiczne i łańcuchowe, a wpisywanie odbywa się niejawnie.
in1 = INPUT ( ścieżka = "myFile.csv" ) stała 1 = 1 elementInstancja1 = Mój składnik ( port wejściowy1 = in1 , parametr_wejściowy1 = stała1 )
Przepływy pracy są konstruowane poprzez przypisanie wyjść instancji komponentów do wejść kolejnych komponentów.
składnikInstancja2 = InnyKomponent ( port wejściowy1 = elementInstancja1 . port wyjściowy1 )
Instancje komponentów można również opakować jako funkcje.
function MojaFunkcja ( InType1 in1 , ..., opcjonalnie InTypeM inM , ParType1 param1 , ..., ParTypeP paramP = defaultP ) -> ( OutType1 out1 , ..., OutTypeN outN ) { ... instrukcje ... zwracaj rekord ( out1 = x1 , ..., outN = xN ) }
Oprócz standardowych instrukcji if-else i switch-case, AndurilScript zawiera również pętle for.
// Iteruje po 1, 2, ..., 10 array = record () for i : std . zakres ( 1 , 10 ) { tablica [ i ] = Jakiś Składnik ( k = i ) }
Rozciągliwość
Anduril można rozbudowywać na wielu poziomach. Użytkownicy mogą dodawać nowe komponenty do istniejących pakietów komponentów. Jeśli jednak nowy komponent lub komponenty wykonują zadania niezwiązane z istniejącymi pakietami, użytkownicy mogą również tworzyć nowe pakiety.
Moksiskaan
Moksiskaan to platforma integracji danych dla badań nad rakiem i biologii molekularnej . Ramy zapewniają relacyjną bazę danych, która reprezentuje wykres jednostek biologicznych, takich jak geny, białka, leki, szlaki, choroby, procesy biologiczne, składniki komórkowe i funkcje molekularne. Ponadto istnieje szeroki zestaw narzędzi analitycznych i akcesyjnych zbudowanych na podstawie tych danych. Zdecydowana większość tych narzędzi jest zaimplementowana jako komponenty i funkcje Anduril.
Moksiskaan jest używany głównie do interpretacji list kandydujących genów uzyskanych z badań genomicznych. Jego narzędzia mogą być używane do generowania wykresów jednostek biologicznych związanych z genami wejściowymi. Dokładny przebieg tych wykresów może różnić się od przewidywań celu leku do szeregów czasowych kaskad sygnalizacyjnych. Niektóre z celów tych narzędzi są ściśle związane z IPA .
Zobacz też
- Systemy zarządzania przepływem pracy w bioinformatyce
- Wzór genu
- Keplera
- Tawerna Apache
- System zarządzania przepływem pracy
Dalsza lektura
- Naukowcy opracowują nową bazę danych, która zapewnia kompleksowy obraz genomu glejaka wielopostaciowego w raporcie badawczym Cancer Genome Atlas Research Briefs, marzec 2011, autorstwa Catherine Evans.
- Almeida, JS (2010). „Ekosystemy obliczeniowe dla genomiki medycznej opartej na danych” . Medycyna genomowa . 2 (9): 67. doi : 10.1186/gm188 . PMC 3092118 . PMID 20854645 .
- Sahu, B.; Laakso, M.; Ovaska, K.; Mirtti, T.; Lundin, J.; Rannikko, A.; Sankila, A.; Turunen, JP; Lundin, M.; Konsti, J.; Vesterinen, T.; Nordling, S.; Kallioniemi, O.; Hautaniemi S.; Janne, OA (2011). „Podwójna rola FoxA1 w wiązaniu receptora androgenowego z chromatyną, sygnalizacją androgenową i rakiem prostaty” . Dziennik EMBO . 30 (19): 3962–3976. doi : 10.1038/emboj.2011.328 . PMC 3209787 . PMID 21915096 .
- Pihlajamaa, P.; Zhang, F. -P .; Saarinen, L.; Mikkonen, L.; Hautaniemi S.; Janne, OA (2011). „Fitoestrogen Genisteina jest specyficznym tkankowo modulatorem receptora androgenowego” . Endokrynologia . 152 (11): 4395–4405. doi : 10.1210/en.2011-0221 . PMID 21878517 .
- Blom, H.; Rönnlund, D.; Scott, L.; Spicarova, Z.; Rantanen, V.; Widengren, J.; Aperia, A.; Brismar, H. (2011). „Analiza najbliższego sąsiada receptorów dopaminy D1 i Na + -K + -ATPaz w kolcach dendrytycznych wypreparowanych za pomocą mikroskopii STED”. Badania i technika mikroskopii . 75 (2): 220–228. doi : 10.1002/jemt.21046 . PMID 21809413 . S2CID 206067902 .
- Ehlers, PI; Kivimaki, AS; Turpeinen, AM; Korpela R.; Vapaatalo, H. (2011). „Wysokie działanie obniżające ciśnienie krwi i chroniące naczynia krwionośne produktów mlecznych w eksperymentalnym nadciśnieniu” . Brytyjski Dziennik Żywienia . 106 (9): 1353–1363. doi : 10.1017/S0007114511001723 . PMID 21736845 .
- Maliniemi, P.; Carlsson, E.; Kaukola, A.; Ovaska, K.; Niiranen, K.; Saksela, O.; Jeskanen, L.; Hautaniemi S.; Ranki, A. (2011). „Zmiany liczby kopii NAV3 i geny docelowe w raku podstawnokomórkowym i płaskonabłonkowym”. Dermatologia Eksperymentalna . 20 (11): 926–931. doi : 10.1111/j.1600-0625.2011.01358.x . PMID 21995814 . S2CID 26219786 .
- Chen, P.; Lepichowa, T.; Hu, Y.; Monni, O.; Hautaniemi, S. (2011). „Kompleksowa metoda przetwarzania danych z macierzy eksonów do analizy ilościowej alternatywnych wariantów składania” . Badania kwasów nukleinowych . 39 (18): e123. doi : 10.1093/nar/gkr513 . PMC 3185423 . PMID 21745820 .
-
Karinen S., Heikkinen T.; i in. (2011). „Przepływ pracy integracji danych w celu wyszukiwania genów napędzających choroby i wariantów genetycznych” . PLOS JEDEN . 6 (4): e18636. doi : 10.1371/journal.pone.0018636 . PMC 3075259 . PMID 21533266 .

- Heinonen M., Hemmes A.; i in. (2011). „Rola białka wiążącego RNA HuR w raku przewodowym piersi in situ” . Dziennik patologii . 224 (4): 529–539. doi : 10.1002/ścieżka.2889 . PMC 3504799 . PMID 21480233 .
- Louhimo R., Hautaniemi S. (2011). „CNAmet: pakiet R do integracji danych dotyczących liczby kopii, metylacji i ekspresji” . Bioinformatyka . 27 (6): 887–888. doi : 10.1093/bioinformatyka/btr019 . PMID 21228048 .
