C13orf42
| Identyfikatory | |||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| C13orf42 | |||||||||||||||||||||||||||||||||||||||||||||||
| , LINC00372, LINC00371, długie międzygenowe niekodujące białko RNA 371, chromosom 13 otwarta ramka odczytu 42 | |||||||||||||||||||||||||||||||||||||||||||||||
| Identyfikatory zewnętrzne | |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| Wikidane | |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
C13orf42 jest białkiem , które u ludzi jest kodowane przez otwartą ramkę odczytu 42 chromosomu 13 genu (C13orf42). Dane sekwencjonowania RNA wskazują na niską ekspresję genu C13orf42 w różnych tkankach. Przewiduje się, że białko C13orf42 będzie zlokalizowane w mitochondriach , jądrze i cytozolu . Przewidywania struktury trzeciorzędowej dla C13orf42 wskazują na wiele helis alfa .
Gen
Streszczenie
C13orf42 to gen kodujący białko zawierający 4 eksony. C13orf42 jest również znany pod pseudonimami LINC00371 i LINC00372. Sekwencjonowanie RNA pokazuje ekspresję genu na niskim poziomie w różnych tkankach.
Lokalizacja
C13orf42 znajduje się na nici ujemnej chromosomu 13 w pozycji 13q14.3 u ludzi. C13orf42 znajduje się od 51,08 Mb do 51,20 Mb na chromosomie 13 i obejmuje 118 kilozasad.
Sąsiedztwo
Genomowe sąsiedztwo C13orf42 składa się z kilku pseudogenów wraz z podjednostką B rybonukleazy H2 ( RNASEH2B ), niescharakteryzowanym LOC107984554 i rodziną o podobieństwie sekwencji 124 członków A (FAM124A).
eksony
Gen C13orf42 zawiera 4 egzony .
Wyrażenie
Sekwencjonowanie RNA C13orf42 wykazuje ekspresję w różnych tkankach, w tym w śledzionie, nerkach, sercu, mózgu, jądrach, skórze, przełyku, okrężnicy, jelicie cienkim, żołądku, płucach, łożysku, gruczole ślinowym, grasicy i tkance tłuszczowej. Sekwencjonowanie RNA ludzkiej tkanki płodowej pokazuje ekspresję C13orf42 począwszy od 20 tygodnia w jelicie, 16 tygodni w nerkach, 10 tygodni w płucach, a ekspresję w żołądku obserwuje się po 16 tygodniach, ale nie po 10, 18 lub 20 tygodniach. Zarejestrowana ekspresja RNA jest bardzo niska, a wszystkie wyniki są niższe niż 0,5 odczytów na kilozasadę transkryptu na milion zmapowanych odczytów (RPKM). mikromacierzy z NCBI geo (GDS425) pokazują ekspresję w dodatkowych tkankach, w tym w szpiku kostnym, wątrobie, mięśniach szkieletowych, rdzeniu kręgowym i trzustce.
Transkrypcja
Warianty
C13orf42 wytwarza cztery znane warianty transkryptu, wariant 1 , wariant 2 , wariant 3 i wariant X1 . Wariant transkrypcji 3 (numer dostępu: NM_001351589.3) jest najdłuższym wysokiej jakości mRNA o długości 3075 nukleotydów. Wariant transkryptu 3 zawiera 4 eksony i koduje białko o długości 325 aminokwasów.
Wszystkie warianty transkryptu 1, 2 i X1 nie mają pierwszego eksonu, ale są zgodne z eksonami 2, 3 i 4 wariantu transkryptu 3. Warianty 1 i 2 nie kodują białka, podczas gdy warianty 3 i X1 kodują białko. Wariant X1 ma długość 2717 nukleotydów i koduje białko o długości 189 aminokwasów, które jest dopasowane do ostatnich 187 aminokwasów dłuższego białka kodowanego przez wariant transkryptu 3 i różni się dwoma pierwszymi aminokwasami.
Białko
izoformy
Istnieją dwa znane białka kodowane przez izoformy C13orf42. Wariant transkrypcji 3 koduje najdłuższe białko o długości 325 aminokwasów. Wariant transkryptu X1 koduje białko o długości 189 aminokwasów. Białko to jest dopasowane do eksonów 2, 3 i 4 białka o 325 aminokwasach, ale brakuje mu eksonu 1.
Skład białek
C13orf42 ma przewidywany punkt izoelektryczny 9,3 i przewidywaną masę cząsteczkową 37,4 kDa. Ludzki C13orf42 jest bogatym w serynę i dodatnio naładowanym aminokwasy (lizynę i argininę). Ta kompozycja jest częściowo zachowana w ortologach.
Struktura trzeciorzędowa
, że trzeciorzędowa struktura C13orf42 o najwyższym poziomie ufności przewidywana przez I-Tasser ma wiele helis alfa . W poniższej strukturze zaznaczono reszty wskazane jako obecne w C13orf42 w większych ilościach (seryna, lizyna i arginina). Model wypełnienia przestrzeni i model ładunku pokazano również dla C13orf42.
Lokalizacja subkomórkowa
Przewiduje się, że ludzki C13orf42 będzie zlokalizowany w mitochondriach , jądrze , cytozolu i retikulum endoplazmatycznym z przewidywanym niskim odsetkiem ER (<5%). Ortologi pokazują podobną przewidywaną lokalizację subkomórkową, przy czym mitochondria, jądro i cytozol są najbardziej przewidywanymi lokalizacjami, jednak przewidywane wartości procentowe są różne.
Immunohistochemia
Przeciwciało C13orf42 B-4 (numer katalogowy: sc-376095) wykazuje barwienie cytoplazmatyczne i jądrowe w przewodach nasiennych i komórkach Lyediga tkanki jąder. Przeciwciało C13orf42 E-3 (numer katalogowy: sc-374567) wykazuje wybarwienie cytoplazmatyczne w przewodach nasiennych i komórkach Lyediga tkanki jądra oraz lokalizację cytoplazmatyczną i jąderkową w komórkach HeLa.
Modyfikacje potranslacyjne
Przewiduje się, że C13orf42 ma 10 wysoce konserwatywnych (w ponad 70% analizowanych ortologów z poniższej tabeli) miejsc fosforylacji. Miejsca fosforylacji obejmują jedno miejsce CK2 , jedno miejsce fosforylacji TYR , dwa miejsca fosforylacji cAMP i sześć miejsc fosforylacji PKC . Istnieją trzy przewidywane O-β-GlcNAc i dwa przewidywane miejsca yin-yang w C13orf42, które są w pełni konserwowane w ortologach. Miejsce yin-yang występuje, gdy O-β-GlcNAc i fosforylację dla tego samego miejsca. Nie przewiduje się, aby C13orf42 miał miejsca mirystylacji, ponieważ nie zawiera N-końcowej glicyny.
Domeny
C13orf42 nie ma zidentyfikowanych domen o wysokim poziomie pewności lub zachowania w ortologach.
Homologia i ewolucja
ortologi
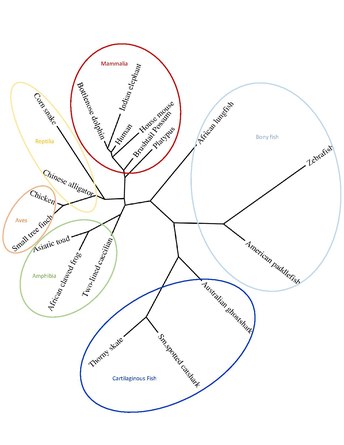
C13orf42 ma ortologi u ssaków , ptaków , gadów , płazów , ryb kostnoszkieletowych i ryb chrzęstnoszkieletowych , jak pokazano w poniższej tabeli ortologów. Nie znaleziono ortologów u ryb bezszczękowych, bezkręgowców, roślin, grzybów, wirusów ani bakterii. Wszystkie ssaki zawierają te same 4 eksony, co ludzkie białko C13orf42, a zwierzętom innym niż ssaki brakuje eksonu 4. Ortologi ssaków mają wysoki procent identyczności z ludzkim C13orf42, z których każdy ma ponad 62% identyczności. Najdalsze ortologi (ryby chrzęstne) mają identyczność sekwencji około 33%. Ludzki C13orf42 nie ma paralogów.
Tabela ortologów
| Rodzaj i gatunek | Nazwa zwyczajowa | Klasa taksonomiczna | Data rozbieżności (MYA) | Numer dostępowy | Długość (aminokwasy) | Procent identyczności z Homo sapiens | Procent podobieństwa do Homo sapiens |
|---|---|---|---|---|---|---|---|
| Homo sapiens | Człowiek | Naczelne ssaki | 0 | NP_001338518.1 | 325 | 100 | 100 |
| Mus musculus | Mysz | Dasyuromorfia | 87 | XP_030104110.1 | 318 | 76,9 | 84 |
| Tursiops truncatus | Delfin butlonosy | Walenie | 94 | XP_033699576.1 | 319 | 82,8 | 87,7 |
| Equus caballus | Koń | Perissodactyla | 94 | XP_023477317.1 | 326 | 82,6 | 90,2 |
| Mustela putorius | Tchórz europejski | Mięsożerca | 94 | XP_004775284.1 | 326 | 79,4 | 85 |
| Pipistrellus kuhlii | Pipistrelle Kuhla (nietoperz) | Rodzina nietoperzy | 94 | XP_036312978.1 | 325 | 79.1 | 86,8 |
| Ursus maritimus | Niedźwiedź polarny | niedźwiedziowate | 94 | XP_040478472.1 | 328 | 77,2 | 82,7 |
| Elephas maximus indicus | Słoń indyjski | trąbowate | 99 | XP_049709344.1 | 326 | 79,4 | 86,2 |
| Dasypus novemcinctus | Dziewięciopasmowy pancernik | Obrączka | 99 | XP_023446856.1 | 328 | 72,8 | 81,4 |
| Dromiciops gliroides | Monito del monte | mikrobioteria | 160 | XP_043849658.1 | 326 | 67,9 | 79,5 |
| Lisek Trichosurus | Opos pospolity | Diprotodoncja | 160 | XP_036599801.1 | 326 | 66,7 | 79,5 |
| Sarcophilus harrisii | Diabeł tasmański | Dasyuromorfia | 160 | XP_023355407.1 | 326 | 66,4 | 80.1 |
| Ornithorhynchus anatinus | Zabawa | stekowce | 180 | XP_028904285.1 | 330 | 62,3 | 73 |
| Aligator sinensis | Chiński aligator | Krokodyl | 319 | XP_025062978.1 | 266 | 48,6 | 61,7 |
| Dromaius novaehollandiae | Emu | kazuarowe | 319 | XP_025964173.1 | 268 | 48.2 | 59,8 |
| Gallus gallus | Kurczak | Galliformes | 319 | XP_004938779.1 | 268 | 47,6 | 61,3 |
| Camarhynchus parvulus | Mała zięba drzewna | Traupidae | 319 | XP_030802909.1 | 264 | 47.1 | 60,9 |
| Pelodiscus sinensis | Chiński żółw softshell | Testudyny | 319 | XP_014429996.1 | 267 | 47 | 60,6 |
| Phasianus colchicus | Bażant | Galliformes | 319 | XP_031471701.1 | 272 | 46,4 | 59,9 |
| Varanus komodoensis | waran z Komodo | łuskonośny | 319 | XP_044300138.1 | 269 | 45,9 | 60.1 |
| Bivittatus Pythona | pyton birmański | łuskonośny | 319 | XP_025026122.1 | 269 | 44.1 | 58 |
| Pantherophis guttatus | Wąż zbożowy | łuskonośny | 319 | XP_034287884.1 | 267 | 42,7 | 56,4 |
| Rhinatrema bivittatum | Dwurzędowy kątownik | Rhinatrematidae | 353 | XP_029459031.1 | 273 | 45,4 | 59,5 |
| Xenopus tropicalis | Zachodnia żaba szponiasta | Anura | 353 | XP_031752228.1 | 260 | 43,5 | 57,4 |
| Bufo gargarizany | Ropucha azjatycka | Anura | 353 | XP_044141363.1 | 262 | 40,7 | 56,8 |
| bufo bufo | Ropucha szara | Anura | 353 | XP_040278366.1 | 391 | 30,9 | 42 |
| Protopterus annectens | Ryby dwudyszne z Afryki Zachodniej | Lepidosireniformes | 408 | XP_043927617.1 | 269 | 37,8 | 53,5 |
| Łopatka Polyodon | Wiosłonos | Acipenseriformes | 431 | XP_041127510.1 | 275 | 33,8 | 52,4 |
| Danio Rerio | Danio pręgowany | karpiowate | 431 | XP_021329868.1 | 287 | 30.2 | 44,7 |
| Clupea harengus | Śledź atlantycki | Clupeiformes | 431 | XP_042559186.1 | 306 | 29 | 41,7 |
| Callorhinchus milii | Australijski rekin widmo | Chimeriformes | 464 | XP_042189981.1 | 267 | 35,5 | 53,3 |
| Amblyraja radiata | Ciernista łyżwa | Rajiformes | 464 | XP_032889397.1 | 267 | 33 | 47,5 |
| Scyliorhinus canicula | Rekin drobnoplamisty | Carcharhiniformes | 464 | XP_038658253.1 | 264 | 32,5 | 50,4 |
Filogeneza
Drzewo filogenetyczne pokazuje, że ludzki C13orf42 jest najbardziej spokrewniony ze swoimi ortologami ssaków i najdalej spokrewniony z ortologami ryb chrzęstnych.
Funkcjonować
Znaczenie kliniczne
Kanagal-Shamanna i in. al zidentyfikowali ATM z C13orf42 u pacjenta z przewlekłą białaczką limfocytową , która prowadzi do inaktywacji ATM.
Xiong i in. wskazali, że SNP rs7325564 jest istotnie powiązany z nasion i nosa u ludzi.