ChIA-PET
Analiza interakcji chromatyny za pomocą sekwencjonowania sparowanych znaczników końcowych ( ChIA-PET lub ChIA-PETS ) to technika, która obejmuje wzbogacanie oparte na immunoprecypitacji chromatyny (ChIP), zbliżoną ligację chromatyny, sparowane znaczniki końcowe i wysokoprzepustowe sekwencjonowanie w celu określenia de novo dalekosiężne interakcje chromatyny w całym genomie.
Geny mogą być regulowane przez regiony oddalone od promotora, takie jak elementy regulatorowe, izolatory i elementy graniczne oraz miejsca wiązania czynników transkrypcyjnych (TFBS) . Odkrycie wzajemnego oddziaływania między regulatorowymi a regionami kodującymi geny jest niezbędne do zrozumienia mechanizmów regulujących regulację genów w zdrowiu i chorobie (Maston i in., 2006). ChIA-PET można wykorzystać do identyfikacji unikalnych, funkcjonalnych chromatyny między dystalnymi i proksymalnymi miejscami wiązania regulacyjnych czynników transkrypcyjnych i promotorami genów, z którymi oddziałują.
ChIA-PET można również wykorzystać do wyjaśnienia mechanizmów kontroli genomu podczas procesów takich jak różnicowanie , proliferacja i rozwój komórek . Tworząc interakcji ChIA-PET dla białek regulatorowych i regionów promotorowych wiążących DNA , możemy lepiej identyfikować unikalne cele interwencji terapeutycznej (Fullwood i Yijun, 2009).
Metodologia
Metoda ChIA-PET łączy metody oparte na ChIP i metody oparte na przechwytywaniu konformacji chromosomów (3C), aby rozszerzyć możliwości obu podejść. Sekwencjonowanie ChIP (ChIP-Seq) jest popularną metodą stosowaną do identyfikacji TFBS, podczas gdy 3C zastosowano do identyfikacji interakcji chromatyny dalekiego zasięgu. Niezależnie od tego oba mają ograniczenia w identyfikowaniu interakcji dalekiego zasięgu de novo w całym genomie. Chociaż ChIP-Seq jest w stanie zidentyfikować TFBS w całym genomie, dostarcza jedynie liniowych informacji o miejscach wiązania białek wzdłuż chromosomów (ale nie o interakcjach między nimi) i może cierpieć z powodu wysokiego genomowego szumu tła (fałszywie dodatnie). Chociaż 3C jest w stanie analizować nieliniowe interakcje chromatyny dalekiego zasięgu, nie można go stosować w całym genomie i, podobnie jak ChIP-Seq, również cierpi z powodu wysokiego poziomu szumu tła. Ponieważ szum wzrasta w stosunku do odległości między oddziałującymi regionami (maks. 100 kb), do dokładnego scharakteryzowania oddziaływań chromatyny wymagane są pracochłonne i żmudne kontrole. opracowano alternatywne metody, takie jak Hi-C, do profilowania interakcji w całym genomie. Pomimo metod profilowania całego genomu zarówno dla interakcji TFBS, jak i dalekiego zasięgu, połączenie podejść z metodą ChIA-PET pozwala na identyfikację obszarów genomu, w których białko będące przedmiotem zainteresowania jest związane, jak również regionu genomu, z którym oddziałuje.
Metoda ChIA-PET z powodzeniem rozwiązuje problemy niespecyficznego szumu interakcji występującego w ChIP-Seq poprzez sonikację fragmentów ChIP w celu oddzielenia losowych przyłączy od określonych kompleksów interakcji. Następny krok, określany jako wzbogacenie, zmniejsza złożoność analizy całego genomu i dodaje specyficzności do interakcji chromatyny związanych z wcześniej określonymi TF (czynnikami transkrypcyjnymi). Zdolność podejść 3C do identyfikacji interakcji dalekiego zasięgu opiera się na teorii ligacji bliskości. Jeśli chodzi o interligację DNA, fragmenty, które są związane przez wspólne kompleksy białkowe, mają większą przewagę kinetyczną w warunkach rozcieńczenia niż te swobodnie dyfundujące w roztworze lub zakotwiczone w różnych kompleksach. ChIA-PET wykorzystuje tę koncepcję, wprowadzając sekwencje łącznikowe na wolne końce fragmentów DNA związanych z kompleksami białkowymi. W celu zbudowania łączności fragmentów związanych przez kompleksy regulatorowe, sekwencje łącznikowe poddaje się ligacji podczas jądrowej ligacji bliskości. Dlatego produkty ligacji połączonej z łącznikiem można analizować za pomocą ultrawysokoprzepustowego sekwencjonowania PET i mapować na genom referencyjny . Ponieważ ChIA-PET nie jest zależny od określonych miejsc do wykrywania, tak jak 3C i 4C, umożliwia bezstronne wykrywanie de-novo interakcji chromatyny w całym genomie. W porównaniu z Hi-C, zastosowanie metody pulldown przeciwciał ogranicza liczbę sekwencjonowanych fragmentów do interakcji chromatyny związanych z białkiem będącym przedmiotem zainteresowania, co również może ułatwić analizę danych.
Przepływ pracy
Część pracy w laboratorium mokrym:
- Formaldehyd jest używany do sieciowania kompleksów DNA-białko. Sonikacja służy do rozbicia chromatyny, a także do zmniejszenia niespecyficznych interakcji.
- Do wzbogacenia fragmentów chromatyny związanych z białkiem będącym przedmiotem zainteresowania stosuje się wybrane specyficzne przeciwciało. Materiał ChIP związany przez przeciwciało stosuje się do konstruowania ChIA-PET.
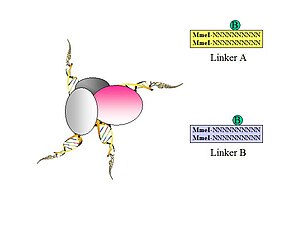
- Ryc. 1. Półłączniki biotynylowanych oligonukleotydów zawierające flankujące miejsca Mmel są stosowane do łączenia fragmentów DNA poddanych ligacji bliskościowej. Zaprojektowano dwa różne łączniki (A i B) ze specyficznymi kodami kreskowymi nukleotydów (CG lub AT) dla każdej z dwóch sekwencji łączników (pozwoli to na identyfikację chimerycznego produktu ligacji, jak opisano na Figurze 5).
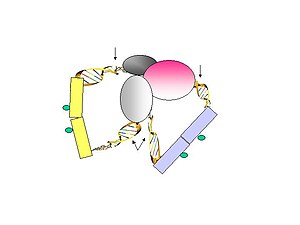
- Ryc. 2. Łączniki są ligowane z uwiązanymi fragmentami DNA.
- Figura 3. Fragmenty łącznika są ligowane na kulkach ChIP w warunkach rozcieńczenia. Oczyszczony DNA jest następnie trawiony przez Mmel, który tnie w pewnej odległości od miejsca rozpoznawania, aby uwolnić strukturę tag-linker-tag.
- Figura 4. Biotynylowane PET unieruchamia się następnie na kulkach magnetycznych sprzężonych ze streptawidyną.
- Rycina 5. Sekwencje PET ze składem kodu kreskowego łącznika AA (CG/CG) i BB (AT/AT) są uważane za możliwe wewnątrzkompleksowe produkty ligacji, podczas gdy sekwencje PET ze składem łącznika AB (CG/AT) są uważane za pochodzące z chimerycznych produktów ligacji między fragmentami DNA związanymi różnymi kompleksami chromatyny.
Część workflow dotycząca laboratorium suchego:
Ekstrakcja PET, mapowanie i analizy statystyczne
Znaczniki PET są ekstrahowane i mapowane na referencyjny ludzki genom in silico .
Identyfikacja pików wzbogaconych w ChIP (miejsca wiązania)
Samoligowane PET są używane do identyfikacji miejsc wzbogaconych w ChIP, ponieważ zapewniają najbardziej niezawodne mapowanie (20 + 20 bitów/s) do genomu referencyjnego.
Algorytm znajdowania szczytów wzbogacania ChIP
Wywołany pik jest uważany za miejsce wiązania, jeśli występuje wiele nakładających się samoligaturowanych PET. Współczynnik fałszywych odkryć (FDR) jest określany za pomocą symulacji statystycznych w celu oszacowania losowego tła nakładania się wirtualnego DNA pochodzącego z PET oraz szacowanego szumu tła.
Filtrowanie powtarzalnego DNA (wpływa na wiązanie niespecyficzne)
Usunięto regiony satelitarne i miejsca wiązania obecne w regionach o poważnych różnicach strukturalnych.
Liczba wzbogacenia ChIP
Liczby PET po samoligacji i międzyligacji (w obrębie okna + 250 pz) podano w każdym miejscu. Całkowita liczba samoligaturowanych i interligowanych PET w określonym miejscu jest nazywana liczbą wzbogacenia ChIP.
Ryc. 6. Klasyfikacja PET: Wyjątkowo dopasowane sekwencje PET można sklasyfikować według tego, czy pochodzą z jednego fragmentu DNA, czy z dwóch fragmentów DNA.
- Samoligaturujące PET
Jeżeli dwa znaczniki PET są zmapowane na tym samym chromosomie z rozpiętością genomową w zakresie fragmentów DNA ChIP (mniej niż 3 kb), z oczekiwaną orientacją samoligacji i na tej samej nici, uważa się, że pochodzą z samoligacja pojedynczego fragmentu DNA ChIP i uważana za PET z samoligacją.
- PET z interligacją
Jeśli PET nie spełnia tych kryteriów, to PET najprawdopodobniej powstał w wyniku ligacji między dwoma fragmentami DNA i jest określany jako PET międzyligacyjny. Dwa znaczniki PET z interligacji nie mają ustalonej orientacji znaczników, mogą nie znajdować się na tych samych niciach, mogą mieć dowolną rozpiętość genomową i mogą nie być mapowane na ten sam chromosom.
- Wewnątrzchromosomalne interligacyjne PET
Jeśli dwa znaczniki PET z interligacji są mapowane na tym samym chromosomie, ale z rozpiętością > 3 Kb w dowolnej orientacji, wówczas te PET są nazywane PET z interligacją wewnątrzchromosomalną.
- PET z interligacją międzychromosomalną
PET, które są mapowane na różne chromosomy, nazywane są PET z interligacją międzychromosomalną.
Ryc. 7. Proponowany mechanizm pokazujący, w jaki sposób dystalne elementy regulatorowe mogą inicjować dalekosiężne interakcje chromatyny z udziałem regionów promotorowych docelowych genów.
Interakcje tworzą struktury pętli DNA z wieloma TFBS w centrum kotwiczenia. Małe pętle mogą upakować geny w pobliżu centrum zakotwiczenia w ciasnym podprzedziale, co może zwiększyć lokalne stężenie białek regulatorowych w celu zwiększenia aktywacji transkrypcji. Mechanizm ten może również zwiększać wydajność transkrypcji, umożliwiając RNA pol II cyklizowanie ciasnych kolistych szablonów genów. Duże pętle interakcji z większym prawdopodobieństwem łączą ze sobą odległe geny na obu końcach pętli, znajdujące się w pobliżu miejsc zakotwiczenia w celu skoordynowanej regulacji, lub mogą rozdzielać geny w długie pętle, aby zapobiec ich aktywacji. Zaadaptowano z Fullwood et al. (2009).
Mocne i słabe strony
Zalety metody ChIA-PET
- ChIA-PET ma potencjał, aby być bezstronnym podejściem obejmującym cały genom i de-novo do analizy interakcji chromatyny dalekiego zasięgu (Fullwood i Yijun, 2009).
- Eksperyment ChIA-PET jest w stanie dostarczyć dwa globalne zestawy danych: miejsca wiązania czynnika białkowego (samoligujące PET); oraz interakcje między miejscami wiązania (interligowane PET).
- ChIA-PET obejmuje ChIP w celu zmniejszenia złożoności analizy całego genomu i dodaje specyficzności interakcjom chromatyny związanym z określonymi czynnikami będącymi przedmiotem zainteresowania.
- ChIA-PET jest kompatybilny z metodami sekwencjonowania nowej generacji opartymi na znacznikach, takimi jak pirosekwencjonowanie Roche 454, Illumina GA, ABI SOLiD i Helicos.
- ChIA-PET ma zastosowanie do wielu różnych czynników białkowych zaangażowanych w regulację transkrypcji lub strukturalną konformację chromatyny.
- Analizę ChIA-PET można zastosować do interakcji chromatyny zaangażowanych w określony proces jądrowy. Używając ogólnych TF, takich jak polimeraza RNA II, możliwe może być zidentyfikowanie wszystkich interakcji chromatyny zaangażowanych w regulację transkrypcji. Ponadto wykorzystanie czynników białkowych zaangażowanych w replikację DNA lub strukturę chromatyny pozwoliłoby na identyfikację wszystkich interakcji związanych z replikacją DNA i modyfikacją struktury chromatyny (Fullwood i in., 2009).
Słabości
- Powszechnie wiadomo, że kompleksy cis i trans-regulatorowe zawierają unikalne kombinacje białek w oparciu o warunki specyficzne dla komórki i tkanki (Dekker i in., 2006). Podczas gdy identyfikacja pojedynczego, funkcjonalnego TFBS jest znaczącym postępem, zastosowanie ChIA-PET do identyfikacji poszczególnych białek w kompleksie wymagałoby zgadywania i wielu eksperymentów w celu zidentyfikowania każdego wchodzącego w interakcje białka. Byłoby to kosztowne i czasochłonne przedsięwzięcie.
- ChIA-PET jest ograniczony jakością, czystością i specyficznością zastosowanych przeciwciał (Fullwood i in., 2009).
- ChIA-PET zależy od identyfikacji sekwencji, które można zmapować do sekwencji referencyjnej (ref.).
- ChIA-PET wymaga użycia algorytmów komputerowych wywołujących szczyty do organizowania i mapowania odczytów PET do genomu referencyjnego. Ze względu na różnice między platformami oprogramowania wyniki mogą się różnić w zależności od używanego programu.
- Chociaż powtarzające się regiony DNA mogą być związane z regulacją genów (Polak i Domany, 2006), należy je usunąć, ponieważ mogą wpływać na dane (Fullwood i in., 2009).
- Wzbogacanie z dwóch miejsc jednocześnie wprowadza uprzedzenia wobec otaczających regionów.
Historia
Fullwood i in. (2009) wykorzystali ChIA-PET do wykrywania i mapowania sieci interakcji chromatyny, w której pośredniczy receptor estrogenowy alfa (ER-alfa) w ludzkich komórkach nowotworowych. Uzyskana globalna mapa interakcjomu chromatyny ujawniła, że odległe miejsca wiązania ER-alfa były również zakotwiczone w promotorach genów poprzez interakcje chromatyny dalekiego zasięgu, co sugeruje, że ER-alfa działa poprzez rozległe pętle chromatyny w celu połączenia genów w celu skoordynowanej regulacji transkrypcji.
Analiza i oprogramowanie
| Oprogramowanie | Opis |
|---|---|
| C3PET | Pakiet oprogramowania do przetwarzania danych ChIA-PET. Wykorzystuje nieparametryczne podejście bayesowskie do przewidywania kompleksów białek oddziałujących z chromatyną. [1] [2] |
| Narzędzie ChIA-PET | Pakiet oprogramowania do przetwarzania danych ChIA-PET. [3] [4] |
| ChIA-PET2 | Pakiet oprogramowania do przetwarzania danych ChIA-PET. Obsługuje dane z różnych protokołów i zapewnia kontrolę jakości analizy danych. [5] [6] |
| ChIA-Sig | Pakiet oprogramowania do przetwarzania danych ChIA-PET przy użyciu modelu NCHG. Witryna internetowa ChIA-Sig [7] [8] |
| ELANDIA | Mapuje fragmenty DNA wzbogacone w ChIP do referencyjnego ludzkiego genomu. [9] |
| Mango | Pakiet oprogramowania do przetwarzania danych ChIA-PET. Wykonuje wszystkie wymagane etapy przetwarzania zestawów danych ChIA-PET i dostarcza statystycznych szacunków ufności dla interakcji. [10] [11] |
| Symulacja Monte Carlo | Służy do oszacowania współczynnika fałszywych odkryć. |
| Interakcje genomowe | Pakiet R do przetwarzania danych ChIA-PET lub Hi-C. [12] [13] |
| DAWAĆ | Biblioteka programistyczna do tworzenia niestandardowej przeglądarki genomu zgodnej z danymi ChIA-PET lub Hi-C. [14] [15] |
| Powtarzaj maskę | Maskowanie in silico powtarzających się elementów. [16] |
Alternatywy
Immunoprecypitacja chromatyny (ChIP):
Oryginalna metoda ChIP to technologia oparta na przeciwciałach, która selektywnie identyfikuje i wiąże białka w celu dostarczenia informacji dotyczących stanów chromatyny i transkrypcji genów . [17]
Mapowanie architektury genomu (GAM):
Ta technika eliminuje szereg wad związanych z technikami opartymi na 3C, zbierając trójwymiarowe odległości między dowolną liczbą loci genomowych .
Rozpoznawanie interakcji w podzielonej puli według rozszerzenia tagu (SPRITE)
SPRITE to technika mapowania interakcji wyższego rzędu w jądrze w całym genomie. Podejście to wykrywa interakcje zachodzące na większych odległościach przestrzennych i umożliwia wykrywanie w całym genomie wielu RNA i DNA zachodzących w tym samym czasie.
Kropla ChIA
ChIA-Drop to prosta metoda analizy multipleksowych interakcji chromatyny przy użyciu sekwencjonowania opartego na kroplach i połączonego z kodem kreskowym z dokładnością do pojedynczej cząsteczki. Poprzednie podejścia oparte na parach na poziomie populacji, takie jak Hi-C i ChIA-PET, różnią się od tej technologii.
- Barski i in., (2007). Profilowanie w wysokiej rozdzielczości metylacji histonów w ludzkim genomie. Komórka. (129); 823–37.
- Dekkera, (2002). Przechwytywanie konformacji chromosomu. Nauka. (295); 1306-1311.
- Dekkera (2006). Trzy „C” przechwytywania konformacji chromosomu: kontrole, kontrole, kontrole. Nat. Metody. (3); 17–21.
- Fullwood i in., (2009). Interaktom ludzkiej chromatyny związany z receptorem estrogenowym-α. Natura. (462); 58–64.
- Fullwood i Yijun (2009). Oparte na technologii ChIP metody identyfikacji dalekosiężnych oddziaływań chromatyny. J Cell Biochem. 107 ust. 1; 30–39.
- Johnson i in., (2007). Mapowanie całego genomu interakcji białko-DNA in vivo. Nauka. (316); 1497–502.
- Kuo i Allis, (1999). Sieciowanie in vivo i immunoprecypitacja do badania dynamicznych asocjacji białka: DNA w środowisku chromatyny. Metody. (19); 425–33.
- Li, G., Fullwood, MJ, Xu, H., Mulawadi, FH, Velkov, S., Vega, V., Ariyaratne, PN, Mohamed, YB, Ooi, HS, Tennakoon, C., Wei, CL, Ruan , Y. i Sung, narzędzie WK ChIA-PET do kompleksowej analizy interakcji chromatyny z sekwencjonowaniem znaczników ze sparowanymi końcami. Genom Biol, 11 (2). R22.
- Maston i in., (2006). Transkrypcyjne elementy regulacyjne w ludzkim genomie. rok Rev: Genomika. Hum Genet. (7); 29–59.
- Polak i Domany, (2006). Elementy Alu zawierają wiele miejsc wiążących czynniki transkrypcyjne i mogą odgrywać rolę w regulacji procesów rozwojowych. Genomika BMC. (7); 133.
- Wei i in., (2006). Globalna mapa miejsc wiązania czynnika transkrypcyjnego p53 w ludzkim genomie. Komórka. (124); 207–19.
Linki zewnętrzne
- Przeglądarka genomu ChIA-PET — ta przeglądarka służy do przeglądania danych z Fullwood et al. (2009) i zawiera niestandardową przeglądarkę interakcji całego genomu, która zapewnia makroskopowy obraz miejsc wiązania i interakcji wraz z całym krajobrazem genomu.