Hi-C (technika analizy genomu)

Hi-C (lub standardowy Hi-C ) to wysokowydajna technika genomowa i epigenomiczna , opisana po raz pierwszy w 2009 roku przez Liebermana-Aidena i in. uchwycić konformację chromatyny . Ogólnie rzecz biorąc, Hi-C jest uważany za pochodną szeregu wychwytywania konformacji chromosomów , w tym między innymi 3C (przechwytywanie konformacji chromosomów), 4C (przechwytywanie konformacji chromosomów na chipie/przechwytywanie konformacji chromosomów okrężnych) i 5C (kopia węglowa wychwytująca konformację chromosomu). Hi-C kompleksowo wykrywa interakcje chromatyny w całym genomie w jądro komórkowe poprzez połączenie podejścia 3C i sekwencjonowania nowej generacji (NGS) i zostało uznane za jakościowy skok w rozwoju technologii C (technologie oparte na przechwytywaniu konformacji chromosomów) i początek genomiki 3D.
Podobnie jak klasyczna technika 3C, Hi-C mierzy częstotliwość (jako średnią z populacji komórek), z jaką dwa fragmenty DNA łączą się fizycznie w przestrzeni 3D, łącząc strukturę chromosomu bezpośrednio z sekwencją genomową. Ogólna procedura Hi-C obejmuje najpierw sieciowanie materiału chromatynowego przy użyciu formaldehydu . Następnie chromatyna jest rozpuszczana i fragmentowana, a oddziałujące loci są ponownie łączone razem, aby stworzyć genomową bibliotekę chimerycznego DNA Cząsteczki. Względna obfitość tych chimer lub produktów ligacji jest skorelowana z prawdopodobieństwem, że odpowiednie fragmenty chromatyny oddziałują w przestrzeni 3D w całej populacji komórek. Podczas gdy 3C koncentruje się na analizie zestawu z góry określonych loci genomowych, aby zaoferować badania „jeden przeciwko niektórym” konformacji interesujących regionów chromosomów, Hi-C umożliwia profilowanie interakcji „wszystko przeciwko wszystkim” poprzez znakowanie wszystkich pofragmentowanych chromatyna z biotynylowanym nukleotydem przed ligacją. W rezultacie biotyną mogą być skuteczniej oczyszczane przez streptawidynę -powlekane kulki magnetyczne, a dane interakcji chromatyny można uzyskać przez bezpośrednie sekwencjonowanie biblioteki Hi-C.
Analizy danych Hi-C nie tylko ujawniają ogólną strukturę genomową chromosomów ssaków, ale także dają wgląd w biofizyczne właściwości chromatyny, a także bardziej szczegółowe kontakty dalekiego zasięgu między odległymi elementami genomu (np. między genami a elementami regulatorowymi ). W ostatnich latach Hi-C znalazło zastosowanie w wielu różnych dziedzinach biologicznych, w tym we wzroście i podziale komórek , regulacji transkrypcji , określaniu losu , rozwoju, chorobach i ewolucji genomu . Łącząc dane Hi-C z innymi zestawami danych, takimi jak mapy modyfikacji chromatyny w całym genomie i profile ekspresji genów, można również określić funkcjonalne role konformacji chromatyny w regulacji i stabilności genomu.
Historia
Na początku Hi-C była technologią o niskiej rozdzielczości i wysokim poziomie szumów, która była zdolna do opisywania regionów interakcji chromatyny tylko w przedziale wielkości 1 miliona par zasad (Mb). Skonstruowanie biblioteki Hi-C również wymagało kilku dni, a same zbiory danych miały niską wydajność i odtwarzalność. Niemniej jednak dane Hi-C dostarczyły nowych informacji na temat konformacji chromatyny oraz architektury jądrowej i genomowej, a perspektywy te zmotywowały naukowców do podjęcia wysiłków w celu zmodyfikowania tej techniki w ciągu ostatniej dekady.
W latach 2012-2015 miało miejsce kilka modyfikacji protokołu Hi-C, z trawieniem 4-ostrzowym lub dostosowaną głębszą głębokością sekwencjonowania w celu uzyskania wyższej rozdzielczości. Zastosowanie endonukleaz restrykcyjnych , które tną częściej lub DNazy i nukleaz Micrococcal również znacząco zwiększyło rozdzielczość metody. Niedawno (2017), Belaghzal i in. opisał protokół Hi-C 2.0, który był w stanie osiągnąć rozdzielczość w kilobazach (kb). Kluczową adaptacją do protokołu podstawowego było usunięcie SDS etap solubilizacji po trawieniu w celu zachowania struktury jądrowej i zapobieżenia przypadkowej ligacji między fragmentowaną chromatyną przez ligację w obrębie nienaruszonych jąder, co stanowiło podstawę Hi-C in situ. W 2021 roku Lafontaine i wsp. Opisali Hi-C 3.0, z wyższą rozdzielczością osiągniętą przez wzmocnienie sieciowania formaldehydem, a następnie glutaranem disukcynimidylu (DSG). Podczas gdy formaldehyd wychwytuje grupę aminową i iminową grupy zarówno białek, jak i DNA, estry NHS w DSG reagują z pierwszorzędowymi aminami na białkach i mogą wychwytywać interakcje amina-amina. Te aktualizacje protokołu podstawowego umożliwiły naukowcom przyjrzenie się bardziej szczegółowym strukturom konformacyjnym, takim jak przedział chromosomalny i domeny asocjacyjne topologicznie (TAD), a także cechom konformacyjnym o wysokiej rozdzielczości, takim jak pętle DNA.
Do tej pory pojawiło się już wiele pochodnych Hi-C, w tym in situ Hi-C, low Hi-C, SAFE Hi-C i Micro-C, z charakterystycznymi cechami związanymi z różnymi aspektami standardowego Hi-C, ale podstawowa zasada pozostała ta sama.
Tradycyjny Hi-C
Po raz pierwszy opublikowany przez Liebermana-Aidena i in. w 2009 r. zarys klasycznego przepływu pracy Hi-C jest następujący: komórki są sieciowane formaldehydem; chromatyna jest trawiona enzymem restrykcyjnym, który generuje wystający fragment 5' ; wystający fragment 5' wypełnia się biotynylowanymi zasadami, a otrzymany DNA o tępych końcach poddaje się ligacji. Produkty ligacji z biotyną na połączeniu są wybierane do użycia streptawidyny i dalej przetwarzane w celu przygotowania biblioteki gotowej do dalszych prób sekwencjonowania.
Interakcje parami, które Hi-C może uchwycić w genomie, są ogromne, dlatego ważne jest, aby przeanalizować odpowiednio dużą wielkość próbki, aby uchwycić unikalne interakcje, które można zaobserwować tylko u mniejszości ogólnej populacji. Aby uzyskać bibliotekę produktów ligacji o wysokiej złożoności, która zapewni wysoką rozdzielczość i głębię danych, wymagana jest próbka 20–25 milionów komórek jako dane wejściowe dla Hi-C. Pierwotne próbki ludzkie, które mogą być dostępne tylko w mniejszej liczbie komórek, można wykorzystać do przygotowania standardowej biblioteki Hi-C z zaledwie 1–5 milionami komórek. Jednak użycie tak niskiego wkładu komórek może wiązać się z niską złożonością biblioteki, co skutkuje wysokim odsetkiem podwójnych odczytów podczas przygotowywania biblioteki.
Standard Hi-C dostarcza danych o interakcjach parami w rozdzielczości od 1 do 10 Mb, wymaga dużej głębokości sekwencjonowania, a wykonanie protokołu zajmuje około 7 dni.
Sieciowanie formaldehydem
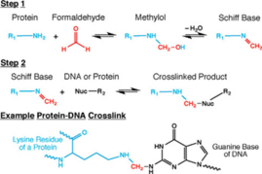
Błony komórkowe i jądrowe są wysoce przepuszczalne dla formaldehydu. Sieciowanie formaldehydowe jest często stosowane do wykrywania i oznaczania ilościowego interakcji DNA-białko i białko-białko. Interesująca w kontekście Hi-C i wszystkich metod opartych na 3C jest zdolność formaldehydu do wychwytywania interakcji chromosomów cis między dystalnymi segmentami chromatyny. Czyni to poprzez tworzenie wiązań kowalencyjnych między przestrzennie sąsiadującymi segmentami chromatyny. Formaldehyd może reagować z makrocząsteczkami w dwóch etapach: najpierw reaguje z nukleofilem na przykład na zasadzie DNA i tworzy addukt metylolowy, który jest następnie przekształcany w zasadę Schiffa . W drugim etapie zasada Schiffa, która może szybko się rozkładać, tworzy mostek metylenowy z inną grupą funkcyjną na innej cząsteczce. Może również tworzyć ten mostek metylenowy z małą cząsteczką w roztworze, takim jak glicyna , który jest stosowany w nadmiarze do gaszenia formaldehydu w Hi-C. Wygaszacze mogą zazwyczaj wywierać wpływ na formaldehyd spoza komórki. Kluczową cechą tej dwuetapowej reakcji sieciowania formaldehydem jest to, że wszystkie reakcje są odwracalne, co ma kluczowe znaczenie dla wychwytu chromatyny.
Sieciowanie jest kluczowym etapem procesu wychwytywania chromatyny, ponieważ funkcjonalnym odczytem techniki jest częstotliwość, z jaką dwa regiony genomowe są ze sobą sieciowane. Dlatego standaryzacja tego etapu jest ważna i w tym celu należy wziąć pod uwagę potencjalne źródła zmienności. Obecność surowicy, która zawiera wysokie stężenie białka, w pożywce hodowlanej może zmniejszyć skuteczne stężenie formaldehydu dostępnego do sieciowania chromatyny, poprzez jego sekwestrację w pożywce hodowlanej. Dlatego w przypadkach, gdy w hodowli stosuje się surowicę, należy ją usunąć na etapie sieciowania. Charakter komórek, tj. to, czy są one zawiesinowe czy przylegające, jest również istotnym czynnikiem branym pod uwagę przy etapie sieciowania. Komórki przylegające wiążą się z powierzchniami za pomocą mechanizmów molekularnych cytoszkielety . Wykazano, że istnieje związek między morfologią jądrową i komórkową utrzymywaną przez cytoszkielet, który, jeśli zostanie zmieniony, może negatywnie wpłynąć na globalną organizację jądrową. Komórki przylegające powinny zatem być usieciowane, gdy nadal są przyczepione do ich powierzchni hodowlanej.
Liza, trawienie restrykcyjne i biotynylacja
Komórki poddaje się lizie na lodzie z zimnym hipotonicznym buforem zawierającym chlorek sodu , Tris-HCl o pH 8,0 i niejonowy detergent IGEPAL CA-630 , uzupełniony inhibitorami proteazy . Inhibitory proteaz i inkubacja na lodzie pomagają zachować integralność usieciowanych kompleksów chromatyny z endogennymi proteazami. Etap lizy pomaga uwolnić materiał nukleinowy z komórek.
Po lizie komórkowej chromatyna jest rozpuszczana w rozcieńczonym SDS w celu usunięcia białek, które nie zostały usieciowane oraz otwarcia chromatyny i uczynienia jej bardziej dostępną dla późniejszego trawienia za pośrednictwem endonukleazy restrykcyjnej. Jeśli inkubacja z SDS przekroczy zalecane 10 minut, sieciowanie formaldehydem może zostać odwrócone, dlatego po inkubacji z SDS należy natychmiast przeprowadzić inkubację na lodzie. Niejonowy detergent o nazwie Triton X-100 jest używany do gaszenia SDS, aby zapobiec denaturacji enzymu w następnym etapie.
Dowolny enzym restrykcyjny, który generuje wystający fragment 5', taki jak Hindlll , może być użyty do trawienia obecnie dostępnej chromatyny przez noc. Ten wystający fragment 5' zapewnia matrycę wymaganą przez fragment Klenowa polimerazy DNA I do dodania biotynylowanego CTP lub ATP do strawionych końców chromatyny. Ten etap umożliwia wybór produktów ligacji Hi-C do przygotowania biblioteki.
Ligacja zbliżeniowa
Ligację rozcieńczeń przeprowadza się na fragmentach DNA, które wciąż są ze sobą usieciowane, aby sprzyjać wewnątrzcząsteczkowej ligacji fragmentów w obrębie tego samego kompleksu chromatyny zamiast ligacji między fragmentami w różnych kompleksach. Ponieważ ten etap ligacji zachodzi między fragmentami DNA o tępych końcach (ponieważ lepkie końce zostały wypełnione zasadami znakowanymi biotyną), reakcja może trwać do 4 godzin, aby zrekompensować jej nieodłączną nieefektywność. W wyniku ligacji bliskościowej końcowe miejsca HindIII są tracone i generowane jest miejsce NheI.
Usuwanie biotyny, ścinanie DNA, wybór rozmiaru i naprawa końcówek
Produkty ligacji znakowane biotyną można oczyszczać stosując ekstrakcję DNA fenolem-chloroformem . W celu usunięcia fragmentów z końcami znakowanymi biotyną, które nie zostały zligowane, stosuje się polimerazę DNA T4 o egzonukleazy 3' do 5' w celu usunięcia nukleotydów z końców takich fragmentów. Ten etap zapewnia, że żaden z tych niezligowanych fragmentów nie zostanie wybrany do przygotowania biblioteki. Reakcję zatrzymuje się za pomocą EDTA i ponownie oczyszcza DNA za pomocą ekstrakcji DNA fenolem-chloroformem.
Idealny rozmiar fragmentów DNA do biblioteki sekwencjonowania zależy od platformy sekwencjonowania, która będzie używana. DNA można najpierw pociąć na fragmenty o długości około 300–500 pz za pomocą sonikacji . Fragmenty tej wielkości nadają się do sekwencjonowania o dużej przepustowości. Po sonikacji fragmenty można wybrać pod względem wielkości za pomocą kulek AMPure XP firmy Beckman Coulter, aby uzyskać produkty ligacji o rozkładzie wielkości między 150 a 300 bp. Jest to optymalne okno wielkości fragmentu do tworzenia klastrów HiSeq.
Ścinanie DNA powoduje asymetryczne pęknięcia DNA i musi zostać naprawione przed ściągnięciem biotyny i ligacją adaptera sekwencjonowania. Osiąga się to za pomocą kombinacji enzymów, które wypełniają nawisy 5' i dodają grupy fosforanowe 5' i adenylan na końcach 3' fragmentów, aby umożliwić ligację adapterów sekwencjonowania.
Wyciąganie biotyny
Stosując nadmiar perełek streptawidyny, takich jak roztwór perełek streptawidyny My-One C1 z Dynabeads , można pobrać i wzbogacić biotynylowane produkty ligacji Hi-C. Ligację sparowanych adapterów końcowych Illumina przeprowadza się, podczas gdy fragmenty DNA są związane z kulkami streptawidyny. Adsorpcja na kulkach zwiększa efektywność ligacji tych tępo zakończonych fragmentów DNA z adapterami, ponieważ zmniejsza ich ruchliwość.
Przygotowanie i sekwencjonowanie biblioteki
Po zakończeniu ligacji adapterów przeprowadza się amplifikację PCR biblioteki. Etap PCR może wprowadzić dużą liczbę duplikatów w próbce produktu ligacji Hi-C o niskiej złożoności w wyniku nadmiernej amplifikacji. Powoduje to przechwycenie bardzo niewielu interakcji, a często dzieje się tak dlatego, że próbka wejściowa miała małą liczbę komórek. Ważne jest, aby miareczkować liczbę cykli wymaganych do uzyskania co najmniej 50 ng DNA z biblioteki Hi-C do sekwencjonowania. Im mniej cykli, tym lepiej, aby nie było artefaktów PCR (takich jak amplikony poza celem, nieswoistość itp.). Idealny zakres cykli PCR to 9–15 i bardziej idealne jest połączenie wielu reakcji PCR w celu uzyskania wystarczającej ilości DNA do sekwencjonowania, niż zwiększanie liczby cykli dla jednej reakcji PCR. Produkty PCR są ponownie oczyszczane przy użyciu perełek AMPure w celu usunięcia dimery starterów , a następnie oznaczane ilościowo przed sekwencjonowaniem. Regiony chromatyny, które oddziałują ze sobą, są następnie identyfikowane przez sekwencjonowanie sparowanych końców biotynylowanych produktów ligacji.
Każda platforma, która może pozwolić na sekwencjonowanie zligowanych fragmentów w poprzek połączenia NheI ( Roche 454) lub przez odczyty ze sparowanymi końcami lub sparowanymi parami ( platformy Illumina GA i HiSeq ) byłaby odpowiednia dla Hi-C. Przed wysokowydajnym sekwencjonowaniem jakość biblioteki należy zweryfikować za pomocą sekwencjonowania Sangera , w którym długi odczyt sekwencjonowania będzie odczytywany przez złącze biotynowe. Trzydzieści sześć lub 50 odczytów bp wystarcza do zidentyfikowania większości par oddziałujących z chromatyną przy użyciu sekwencjonowania sparowanych końców Illumina. Ponieważ średnia wielkość fragmentów w bibliotece wynosi 250 bp, stwierdzono, że odczyty sparowanych końców o długości 50 bp są optymalne do sekwencjonowania biblioteki Hi-C.
Kontrola jakości bibliotek Hi-C
Istnieje kilka punktów nacisku w całym przepływie pracy związanym z przygotowaniem próbki Hi-C, które są dobrze udokumentowane i opisane. DNA na różnych etapach można analizować na 0,8% żelach agarozowych, aby ocenić rozkład wielkości fragmentów. Jest to szczególnie ważne po ścinaniu etapów wyboru rozmiaru. Degradację DNA można również monitorować w postaci rozmazów pojawiających się pod produktami o niskiej masie cząsteczkowej na żelach. Degradacja może nastąpić z powodu niedodania wystarczającej ilości inhibitorów proteazy podczas lizy, aktywności endogennej nukleazy lub degradacji termicznej z powodu nieprawidłowego oblodzenia. Reakcje 3C PCR można przeprowadzić w celu przetestowania tworzenia się produktów ligacji w sąsiedztwie.
Warianty
Standardowy Hi-C ma wysoki koszt liczby komórek wejściowych, wymaga głębokiego sekwencjonowania, generuje dane o niskiej rozdzielczości i cierpi z powodu tworzenia zbędnych cząsteczek, które przyczyniają się do bibliotek o niskiej złożoności, gdy liczba komórek jest niska. Aby zwalczyć te problemy, aby móc zastosować tę technikę w kontekstach, w których liczba komórek jest czynnikiem ograniczającym, na przykład przy pierwotnej pracy z komórkami ludzkimi, od czasu pierwszej konceptualizacji Hi-C opracowano kilka wariantów Hi-C.
Cztery główne klasy, do których należą warianty Hi-C, to: ligacja rozcieńczająca, ligacja in situ, systemy jednokomórkowe i systemy poprawy o niskim poziomie szumów. Standard Hi-C jest typem ligacji rozcieńczającej, a inne ligacje rozcieńczające obejmują DNazę Hi-C i Capture Hi-C. W przeciwieństwie do standardu i Capture Hi-C, DNaza Hi-C wymaga jedynie 2–5 milionów komórek jako danych wejściowych, wykorzystuje DNazę I do fragmentacji chromatyny i wykorzystuje ligację bliskościową rozcieńczania w żelu. Wykazano, że zastosowanie DNazy znacznie poprawia wydajność i rozdzielczość Hi-C. Capture Hi-C to technika oznaczania obejmująca cały genom, mająca na celu zbadanie interakcji chromatyny w określonych loci za pomocą hybrydyzacji oparte na przechwytywaniu docelowych regionów genomowych. Po raz pierwszy został opracowany przez Mifsuda i in. promotora dalekiego zasięgu w komórkach ludzkich, generując bibliotekę przynęty z biotynylowanym RNA, która była ukierunkowana na 21 841 regionów promotora. Warianty te, oprócz innych (opisanych poniżej), stanowią modyfikacje fundamentalnej techniki standardowego Hi-C i usuwają jedno lub więcej ograniczeń oryginalnej metody.
In situ Hi-C
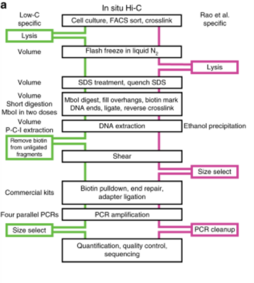
Po raz pierwszy opisany przez Rao i wsp. Hi-C in situ łączy standardowy Hi-C z testem ligacji jądrowej, tj. ligacją bliskościową przeprowadzaną w nienaruszonych jądrach. Protokół jest podobny do standardowego Hi-C pod względem podstawowego schematu przepływu pracy, ale różni się pod innymi względami. In situ Hi-C wymaga od 2 do 5 milionów komórek w porównaniu do idealnych 20 do 25 milionów wymaganych dla standardowego Hi-C, a ukończenie protokołu wymaga tylko 3 dni w porównaniu do 7 dni w przypadku standardowego Hi-C. Ponadto ligacja zbliżeniowa nie zachodzi w roztworze, jak w standardowym Hi-C, zmniejszając częstotliwość przypadkowych, biologicznie nieistotnych kontaktów i ligacji, na co wskazuje niższa częstotliwość kontaktów mitochondrialnego i jądrowego DNA w przechwyconym biotynylowanym DNA. Osiąga się to przez pozostawienie jąder w stanie nienaruszonym do etapu ligacji. Komórki nadal poddaje się lizie buforem zawierającym Tris-HCl o pH 8,0, chlorek sodu i detergent IGEPAL CA630 przed ligacją, ale zamiast homogenizacji lizatu komórkowego, jądra komórkowe osadza się po początkowej lizie w celu degradacji błony komórkowej. Po zakończeniu ligacji zbliżeniowej jądra komórkowe inkubuje się przez co najmniej 1,5 godziny w temperaturze 68 stopni Celsjusza w celu przepuszczania błony jądrowej i uwolnienia jej zawartości jądrowej.
Rozdzielczość, którą można osiągnąć za pomocą Hi-C in situ, może wynosić do 950 do 1000 bp w porównaniu z rozdzielczością 1 do 10 Mb standardowego Hi-C i rozdzielczością 100 kb DNazy Hi-C. Podczas gdy standardowy Hi-C wykorzystuje narzędzie tnące o 6 pz, takie jak HindIII, do etapu trawienia restrykcyjnego, Hi-C in situ wykorzystuje narzędzie tnące o 4 pz, takie jak MboI lub jego izoschizomer DpnII (który nie jest wrażliwy na metylację CpG ) w celu zwiększenia wydajności i rozdzielczości (ponieważ miejsca restrykcyjne MboI i DpnII częściej występują w genomie). Dane między powtórzeniami dla Hi-C in situ są spójne i wysoce powtarzalne, z bardzo mniejszym szumem tła i wykazują wyraźne interakcje chromatyny. Jest jednak możliwe, że niektóre z przechwyconych interakcji mogą nie być dokładnymi interakcjami międzycząsteczkowymi, ponieważ jądro jest gęsto upakowane białkiem i DNA, więc wykonanie ligacji zbliżeniowych w nienaruszonych jądrach może usunąć zakłócające interakcje, które mogą powstać tylko ze względu na naturę opakowania jądrowego i nie tyle unikalne interakcje chromosomalne z komórkowym wpływem funkcjonalnym. Wymaga to również niezwykle dużej głębokości sekwencjonowania, wynoszącej około 5 miliardów odczytów sparowanych końców na próbkę, aby osiągnąć rozdzielczość danych opisaną przez Rao i in. Istnieje kilka technik, które zaadaptowały koncepcję in situ Hi-C, w tym Sis Hi-C, OCEAN-C i przechwytywanie in situ Hi-C. Poniżej opisano dwie najbardziej znane techniki oparte na Hi-C in situ.
1. Niskie C
Low-C jest protokołem Hi-C in situ przystosowanym do stosowania w przypadku małej liczby komórek, co jest szczególnie przydatne w kontekstach, w których liczba komórek jest czynnikiem ograniczającym, na przykład w pierwotnej hodowli komórek ludzkich. Ta metoda wykorzystuje niewielkie zmiany, w tym stosowane objętości i stężenia oraz czas i kolejność niektórych etapów eksperymentalnych, aby umożliwić generowanie wysokiej jakości bibliotek Hi-C z liczby komórek tak niskiej jak 1000. Pomimo potencjału generowania użytecznych danych o wysokiej rozdzielczości z zaledwie 1000 komórek, Diaz i in. nadal zalecają użycie co najmniej 1 do 2 milionów komórek, jeśli jest to wykonalne, a jeśli nie, minimum 500 K komórek. Jakość biblioteki została najpierw oceniona na platformie Illumina MiSeq (odczyty 2x84 np sparowanych końcówek) i po przejściu kryteriów kontroli jakości (w tym niskich duplikatów PCR), biblioteka została zsekwencjonowana na Illumina NextSeq (2x80 bp sparowanych końcówek). Ogólnie rzecz biorąc, ta technika omija problem wymagający dużej liczby komórek wejściowych dla Hi-C i dużej głębokości sekwencjonowania wymaganej do uzyskania danych o wysokiej rozdzielczości, ale może osiągnąć rozdzielczość tylko do 5 kb i nie zawsze może być odtwarzalna ze względu na zmienny charakter wielkości użytych próbek i wygenerowanych z nich danych.
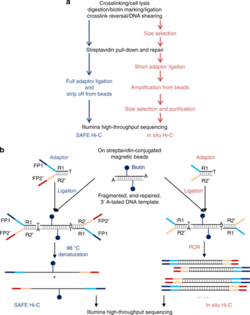
2. BEZPIECZNE Hi-C
SAFE Hi-C lub uproszczony, szybki i ekonomiczny Hi-C generuje wystarczającą ilość zligowanych fragmentów bez amplifikacji do wysokowydajnego sekwencjonowania. Opublikowane dane Hi-C in situ wskazują, że amplifikacja (na etapie PCR w celu przygotowania biblioteki) wprowadza zależne od odległości odchylenie amplifikacji, co skutkuje wyższym stosunkiem szumu do sygnału w stosunku do odległości genomowej. SAFE Hi-C z powodzeniem wykorzystano do wygenerowania wolnej od amplifikacji biblioteki ligacji Hi-C in situ z zaledwie 250 tysięcy komórek K562 . Fragmenty ligacji mają długość od 200 do 500 pz, średnio około 370 pz. Wszystkie biblioteki produktów ligacji sekwencjonowano przy użyciu platformy Illumina HiSeq (odczyty sparowanych końców 2x150 bp). Chociaż SAFE Hi-C może być używany do komórek wejściowych o wartości zaledwie 250 tysięcy, Niu i in. zaleca się użycie od 1 do 2 milionów komórek. Próbki wytwarzają wystarczającą ilość ligatów do sekwencjonowania na jednej czwartej ścieżki. Wykazano, że SAFE Hi-C zwiększa złożoność biblioteki ze względu na usuwanie duplikatów PCR, co obniża ogólny odsetek unikalnych sparowanych odczytów. Ogólnie rzecz biorąc, SAFE Hi-C zachowuje integralność interakcji chromosomalnych, jednocześnie zmniejszając potrzebę posiadania dużej głębokości sekwencjonowania i oszczędzając ogólne koszty i robociznę.
Mikro-C
Micro-C to wersja Hi-C, która obejmuje etap trawienia nukleazą mikrokokową (MNazą) w celu zbadania interakcji między parami nukleosomów, umożliwiając w ten sposób rozdzielczość subgenomowych struktur TAD w skali od 1 do 100 nukleosomów. Został po raz pierwszy opracowany do stosowania w drożdżach i wykazano, że zachowuje dane strukturalne uzyskane ze standardowego Hi-C, ale z większym stosunkiem sygnału do szumu. W przypadku stosowania z ludzkimi zarodkowymi komórkami macierzystymi i fibroblastami , na próbkę uzyskano od 2,6 do 4,5 miliarda jednoznacznie zmapowanych odczytów. Hsieh i in. przeanalizowali 2,64 miliarda odczytów z embrionalnych komórek macierzystych myszy i wykazali, że istnieje zwiększona moc wykrywania interakcji krótkiego zasięgu.
Jednoogniwowy Hi-C
Hi-C został również przystosowany do użytku z pojedynczymi komórkami, ale techniki te wymagają wysokiego poziomu wiedzy specjalistycznej do wykonania i są nękane takimi problemami, jak niska jakość danych, zasięg i rozdzielczość.
Analiza danych
Chimeryczne produkty ligacji DNA generowane przez Hi-C reprezentują parami oddziaływania chromatyny lub fizyczne kontakty 3D w jądrze i mogą być analizowane za pomocą różnych dalszych podejść. W skrócie, dane z głębokiego sekwencjonowania są wykorzystywane do tworzenia bezstronnych map interakcji chromatyny w całym genomie. Następnie można zastosować kilka różnych metod do analizy tych map w celu zidentyfikowania strukturalnych wzorców chromosomów i ich biologicznych interpretacji. Wiele z tych podejść do analizy danych ma również zastosowanie do sekwencjonowania 3C lub innych równoważnych danych.
Przeczytaj Mapowanie
Dane Hi-C wytworzone przez głębokie sekwencjonowanie mają postać tradycyjnego pliku FASTQ , a odczyty można dopasować do genomu będącego przedmiotem zainteresowania za pomocą oprogramowania do dopasowywania sekwencji (np. Bowtie , bwa itp.). Ponieważ produkty ligacji Hi-C mogą rozciągać się na setki megabaz i mogą łączyć loci na różnych chromosomach, dopasowanie odczytu Hi-C jest często chimeryczne w tym sensie, że różne części odczytu mogą być dopasowane do loci odległych od siebie, prawdopodobnie w różnych orientacjach. Wyrównywacze do długiego odczytu (np. minimapa2) często obsługują wyrównanie chimeryczne i mogą być bezpośrednio stosowane do długo czytanych danych Hi-C. Wyrównanie Hi-C przy krótkim odczycie jest trudniejsze.
Warto zauważyć, że Hi-C generuje połączenia ligacyjne o różnych rozmiarach, ale dokładna pozycja miejsca ligacji nie jest mierzona. Aby obejść ten problem, stosuje się mapowanie iteracyjne, aby uniknąć wyszukiwania miejsca połączenia przed podzieleniem odczytów na dwie części i mapowaniem ich osobno w celu zidentyfikowania par interakcji. Ideą mapowania iteracyjnego jest mapowanie jak najkrótszej sekwencji, aby zapewnić jednoznaczną identyfikację par interakcji przed dotarciem do miejsca skrzyżowania. W rezultacie odczyty o długości 25 pz, zaczynając od końca 5', są najpierw mapowane do genomu, a odczyty, które nie są jednoznacznie mapowane na pojedyncze loci, są wydłużane o dodatkowe 5 pz, a następnie ponownie mapowane. Ten proces jest powtarzany, aż wszystkie odczyty zostaną jednoznacznie odwzorowane lub dopóki odczyty nie zostaną rozszerzone na całość. Przechowywane są tylko sparowane odczyty końcowe z każdą stroną jednoznacznie odwzorowaną na pojedyncze loci genomowe. Wszystkie inne sparowane odczyty końcowe są odrzucane.
Kilka odmian technik mapowania odczytu jest zaimplementowanych w wielu potokach bioinformatycznych, takich jak ICE, HiC-Pro, HIPPIE, HiCUP i TADbit, w celu mapowania dwóch części sparowanego końca odczytywanego oddzielnie, w przypadku, gdy te dwie części pasują do różnych pozycji genomowych , odpowiadając w ten sposób na wyzwanie, w którym odczyty obejmują połączenia ligacyjne.
Przy zwiększonej długości odczytu nowsze potoki (np. Juicer i 4D-Nucleosome Data Portal) często dopasowują krótkie odczyty Hi-C za pomocą algorytmu dopasowywania zdolnego do chimerycznego dopasowania, takiego jak bwa-mem, chromap i dragmap . Ta procedura wywołuje wyrównanie raz i jest prostsza niż mapowanie iteracyjne.
Przypisywanie i filtrowanie fragmentów
Następnie każdemu z mapowanych odczytów przypisywana jest pojedyncza lokalizacja dopasowania genomowego zgodnie z jego zmapowaną pozycją 5' w genomie. Dla każdej pary odczytów miejsce jest przypisane tylko do jednego z fragmentów restrykcyjnych , zatem powinno znajdować się blisko miejsca restrykcyjnego i mniej niż maksymalna długość cząsteczki. Odczyty zmapowane w odległości większej niż maksymalna długość cząsteczki od najbliższych miejsc restrykcyjnych są wynikiem fizycznego pęknięcia chromatyny lub aktywności niekanonicznej nukleazy. Ponieważ odczyty te przekazują również informacje o interakcjach chromatyny, nie są one odrzucane, ale po przypisaniu lokalizacji genomowych musi nastąpić odpowiednie filtrowanie w celu usunięcia szumu technicznego w zbiorze danych.
W zależności od tego, czy odczytana para mieści się w tym samym, czy w różnych fragmentach restrykcyjnych, stosowane są różne kryteria filtrowania. Jeśli sparowane odczyty są mapowane na ten sam fragment restrykcyjny, prawdopodobnie reprezentują nieligowane zwisające końce lub zakreślone fragmenty, które nie zawierają informacji i dlatego są usuwane ze zbioru danych. Odczyty te mogą również reprezentować artefakty PCR, niestrawione fragmenty chromatyny lub po prostu odczyty o niskiej jakości wyrównania. Bez względu na ich pochodzenie, odczyty mapowane na ten sam fragment są uważane za „fałszywe sygnały” i są zazwyczaj odrzucane przed dalszym przetwarzaniem.
Pozostałe sparowane odczyty zmapowane na odrębne fragmenty restrykcyjne są również filtrowane w celu odrzucenia identycznych/nadmiarowych produktów PCR, co osiąga się przez usunięcie odczytów o dokładnie tej samej sekwencji lub pozycjach dopasowania 5'. Można również zastosować dodatkowe poziomy filtrowania, aby dopasować się do celu eksperymentu. Na przykład potencjalne niestrawione miejsca restrykcyjne można raczej odfiltrować niż pasywnie zidentyfikować, usuwając odczyty zmapowane na tej samej nici chromosomalnej z niewielką odległością (zdefiniowaną przez użytkownika, opartą na doświadczeniu) pomiędzy nimi.

Sortowanie i filtrowanie na poziomie pojemnika
W oparciu o ich współrzędne punktu środkowego, fragmenty restrykcyjne Hi-C są dzielone na ustalone przedziały genomowe, z rozmiarami przedziałów w zakresie od 40 kb do 1 Mb. Uzasadnieniem tego podejścia jest to, że zmniejszając złożoność danych i zmniejszając liczbę kandydujących interakcji obejmujących cały genom na pojemnik, pojemniki genomowe pozwalają na konstruowanie bardziej niezawodnych i mniej zaszumionych sygnałów, w postaci częstotliwości kontaktowych, w koszt rozdzielczości (chociaż długość fragmentu restrykcyjnego nadal pozostaje ostatecznym fizycznym ograniczeniem rozdzielczości Hi-C). Interakcje między pojemnikami są agregowane po prostu przez pobranie sumy, chociaż przez lata opracowano również bardziej ukierunkowane i informacyjne metody w celu dalszego wzmocnienia sygnału. Jedna taka metoda opisana przez Rao i in. ma na celu przesuwanie limitu wielkości pojemników do coraz mniejszych pojemników, ostatecznie obejmując > 80% pojemników po 1000 odczytów każdy, co znacznie zwiększyło rozdzielczość końcowych wyników analizy.
Filtrowanie na poziomie pojemników, podobnie jak filtrowanie na poziomie fragmentów, odbywa się również w celu usunięcia eksperymentalnych artefaktów z uzyskanych danych. Pojemniki z wysokim szumem i niskimi sygnałami są usuwane, ponieważ zazwyczaj reprezentują wysoce powtarzalną zawartość genomową wokół telomerów i centromerów . Odbywa się to poprzez porównanie sum poszczególnych przedziałów z sumą wszystkich przedziałów i usunięcie dolnego 1% przedziałów lub użycie wariancji jako miary szumu. Pojemniki o niskim pokryciu lub przedziały o trzy odchylenia standardowe poniżej środka rozkładu logarytmiczno-normalnego (który pasuje do całkowitej liczby kontaktów na przedział genomowy) są usuwane przy użyciu filtra MAD-max (maksymalne dozwolone odchylenie bezwzględne mediany). Po binningu dane Hi-C będą przechowywane w formacie symetrycznej macierzy.
Niedawno zaproponowano wiele podejść do wstępnego określenia optymalnego rozmiaru pojemnika dla różnych eksperymentów Hi-C. Li i in. w 2018 roku opisał deDoc, metodę, w której rozmiar pojemnika jest wybierany jako taki, przy którym entropia strukturalna macierzy Hi-C osiąga stabilne minimum. Z drugiej strony QuASAR oferuje nieco lepszą ocenę jakości i porównuje wyniki powtórzeń próbek (zakładając, że powtórzenia są rzeczywiście uwzględnione w celach eksperymentalnych), aby znaleźć maksymalną użyteczną rozdzielczość. W niektórych publikacjach próbowano również oceniać częstości interakcji na poziomie pojedynczego fragmentu, gdzie większe pokrycie można osiągnąć nawet przy mniejszej liczbie odczytów. HiCPlus, narzędzie opracowane przez Zhanga i in. w 2018 roku jest w stanie przypisać macierze Hi-C podobne do oryginalnych, używając tylko 1/16 oryginalnych odczytów.
Równoważenie/normalizacja
Równoważenie odnosi się do procesu korekcji stronniczości uzyskanych danych Hi-C i może być jawne lub niejawne. Jawne metody równoważenia wymagają wyraźnych definicji odchyleń, o których wiadomo, że są związane z odczytami Hi-C (lub ogólnie jakąkolwiek techniką sekwencjonowania o wysokiej przepustowości ), w tym możliwości mapowania odczytu, zawartości GC , a także długości poszczególnych fragmentów. Współczynnik korekcji jest najpierw obliczany dla każdego z rozważanych odchyleń, a następnie dla każdej ich kombinacji, a następnie stosowany do zliczeń odczytów na przedział genomowy.
Jednak niektóre odchylenia mogą pochodzić z nieznanego źródła, w którym to przypadku zamiast tego stosuje się podejście niejawnego równoważenia. Niejawne równoważenie opiera się na założeniu, że każde locus genomowe powinno mieć „równą widoczność”, co sugeruje, że sygnał interakcji w każdym locus genomowym w danych Hi-C powinien sumować się do tej samej całkowitej kwoty. Jedno podejście zwane korektą iteracyjną wykorzystuje algorytm równoważenia Sinkhorna-Knoppa i próbuje zrównoważyć macierz symetryczną przy użyciu wspomnianego założenia (poprzez wyrównanie sumy każdego wiersza i kolumny w macierzy). Algorytm iteracyjnie przechodzi między dwoma krokami: 1) podzielenie każdego wiersza przez jego średnią i 2) podzielenie każdej kolumny przez jego średnią, które gwarantują zbieżność na końcu i nie pozostawiają wyraźnie wysokich wierszy ani kolumn w macierzy interakcji. Istnieją również inne metody obliczeniowe służące do normalizacji odchyleń właściwych dla danych Hi-C, w tym normalizacja składowych sekwencyjnych (SCN), podejście równoważenia macierzy Knighta-Ruiza i normalizacja rozkładu wektorów własnych (ICE). Ostatecznie zarówno jawna, jak i ukryta metoda korekcji odchylenia dają porównywalne wyniki.
Analiza i interpretacja danych
Dzięki podzielonej na kategorie matrycy interakcji obejmującej cały genom, typowe wzorce interakcji obserwowane w genomach ssaków można zidentyfikować i zinterpretować biologicznie, podczas gdy rzadsze i rzadziej obserwowane wzorce, takie jak chromosomy koliste i skupienia centromerów, mogą wymagać dodatkowych, specjalnie dostosowanych metod do zidentyfikowania .
1. Współczynnik interakcji Cis/Trans
cis / trans to jeden z dwóch najsilniejszych wzorców interakcji obserwowanych na mapach Hi-C. Nie są specyficzne dla locus i dlatego są uważane za wzorzec na poziomie genomu. Zazwyczaj obserwuje się wyższą częstotliwość interakcji średnio dla par loci znajdujących się na tym samym chromosomie (w cis) niż par loci znajdujących się na różnych chromosomach (w trans). W macierzach interakcji Hi-C interakcje cis / trans pojawiają się jako kwadratowe bloki wyśrodkowane wzdłuż przekątnej, pasujące jednocześnie do poszczególnych chromosomów. Ponieważ ten wzorzec jest stosunkowo spójny dla różnych gatunków i typów komórek, można go wykorzystać do oceny jakości danych. Bardziej hałaśliwy eksperyment, z powodu losowej ligacji tła lub jakiegokolwiek nieznanego czynnika, spowoduje niższy stosunek interakcji cis do trans (ponieważ oczekuje się, że szum wpłynie na interakcje cis i trans w podobnym stopniu), a eksperymenty wysokiej jakości zazwyczaj mają stosunek interakcji cis/trans między 40 a 60 dla ludzkiego genomu.
2. Częstotliwość interakcji zależna od odległości
Ten wzorzec odnosi się do zależnego od odległości rozpadu częstotliwości interakcji na poziomie genomu i reprezentuje drugi z dwóch najsilniejszych wzorców interakcji Hi-C. W miarę zmniejszania się częstotliwości interakcji między cis-oddziałującymi loci (w wyniku dalszej odległości między nimi), można zaobserwować stopniowe zmniejszanie się częstotliwości interakcji oddalając się od przekątnej w macierzy interakcji.
Istnieją różne modele polimerów do statystycznego charakteryzowania właściwości par loci oddzielonych określoną odległością, ale dyskretne kategoryzowanie i dopasowywanie funkcji ciągłych to dwa powszechne sposoby analizy zależnych od odległości częstotliwości interakcji między punktami danych. Po pierwsze, częstotliwości interakcji można podzielić na kategorie na podstawie ich odległości genomowej, a następnie do danych dopasowuje się funkcję ciągłą, wykorzystując informacje o średniej z każdego przedziału. Wynikowa funkcja rozpadu jest wykreślana na wykresie logarytmicznym, tak że linia liniowa może być wykorzystana do przedstawienia rozkładów potęgowych przewidywanych przez modele polimerowe. Jednak często prosty model polimeru nie będzie wystarczający do pełnego przedstawienia zależnych od odległości częstotliwości interakcji, w którym to momencie mogą wystąpić bardziej skomplikowane funkcje rozpadu, co może wpłynąć na odtwarzalność danych ze względu na obecność specyficznych dla miejsca, a nie genomu -szerokie wzorce obserwowane w matrycy Hi-C (które nie są brane pod uwagę w modelach polimerowych).
3. Przedziały chromatyny
Najsilniejszym wzorcem specyficznym dla locus znalezionym na mapach Hi-C są przedziały chromatyny, które przybierają kształt kratki lub wzoru przypominającego szachownicę na macierzy interakcji, z naprzemiennymi blokami o wielkości od 1 do 10 Mb ( co czyni je łatwymi do ekstrakcji nawet w eksperymentach z bardzo małą próbką) w ludzkim genomie. Ten wzór można znaleźć zarówno przy wysokich, jak i niskich częstotliwościach. Ponieważ chromosomy składają się z dwóch typów regionów genomowych, które występują naprzemiennie wzdłuż długości poszczególnych chromosomów, częstotliwości interakcji między dwoma regionami tego samego typu i częstotliwości interakcji między dwoma regionami różnych typów mogą być całkiem różne.
Definicja aktywnego (A) i nieaktywnego (B) przedziału chromatyny jest oparta na analizie głównych składników , ustalonej po raz pierwszy przez Liebermana-Aidena i in. w 2009 r. Ich podejście obliczyło korelację macierzy Hi-C stosunku sygnału obserwowanego do oczekiwanego (uzyskanego z macierzy kontaktowej znormalizowanej na odległość) i wykorzystało znak pierwszego wektora własnego do oznaczenia dodatnich i ujemnych części wynikowego wykresu odpowiednio jako przedziały A i B. Wiele badań genomicznych wykazało, że przedziały chromatyny są skorelowane ze stanami chromatyny, takimi jak gęstość genów , dostępność DNA, zawartość GC, czas replikacji i znaki histonów . Dlatego przedziały typu A są bardziej szczegółowo zdefiniowane, aby reprezentować regiony euchromatyny o dużej gęstości genów , podczas gdy przedziały typu B reprezentują regiony heterochromatyczne o mniejszej aktywności genów. Ogólnie rzecz biorąc, przedziały chromatyny dają wgląd w ogólne zasady organizacji genomu będącego przedmiotem zainteresowania.
W ciągu ostatniej dekady opracowano coraz więcej narzędzi bioinformatycznych zdolnych do wywoływania przedziałów, w tym HOMER, HiTC R i CscoreTool. Chociaż każdy z nich ma swoje własne różnice i optymalizacje wykonane w stosunku do pierwotnego podejścia z 2009 roku, ich podstawowe protokoły nadal opierają się na analizie głównych składowych.
4. Domeny powiązane topologicznie (TAD)
TAD to struktury sub-Mb, które mogą zawierać cechy regulujące geny, takie jak lokalny promotor - wzmacniacz interakcje. Mówiąc bardziej ogólnie, TAD są uważane za wyłaniającą się właściwość podstawowych mechanizmów biologicznych, która definiuje TAD jako wytłaczanie pętli, kompartmentalizację lub dowolny dynamiczny wzór genomowy, a nie statyczną cechę strukturalną genomu. Zatem TAD reprezentują mikrośrodowiska regulacyjne i zwykle pojawiają się na mapie Hi-C jako bloki wysoce samooddziałujących regionów, w których częstotliwości interakcji w regionie są znacznie wyższe niż częstotliwości interakcji między dwoma sąsiednimi regionami. W macierzach interakcji Hi-C TAD są kwadratowymi blokami o podwyższonych częstotliwościach interakcji, wyśrodkowanymi wzdłuż przekątnej. Jest to jednak jedynie nadmiernie uproszczony opis, a zidentyfikowanie rzeczywistego wzorca wymaga znacznie więcej przetwarzania statystycznego i oszacowania.
Jedno podejście do identyfikacji TAD zostało opisane przez Dixona i wsp., gdzie najpierw obliczyli (w pewnym zakresie genomowym) różnicę między średnimi interakcjami w górę i średnimi interakcjami w dół każdego pojemnika w macierzy. Ta różnica została następnie przekształcona w statystykę chi-kwadrat opartą na Ukrytym Modelu Markowa, a każda gwałtowna zmiana tej wartości chi-kwadrat, zwanej wskaźnikiem kierunkowości, określi granice TAD. Alternatywnie, można po prostu wziąć stosunek między średnimi interakcjami w górę iw dół, aby zdefiniować granice TAD, podobnie jak Naumova i in.
Innym podejściem jest obliczenie średnich częstotliwości interakcji przechodzących przez każdy przedział, ponownie w pewnym z góry określonym zakresie genomowym. Wynikowa wartość jest określana jako ocena izolacji i może być traktowana jako średnia kwadratu przesuwającego się wzdłuż przekątnej macierzy (Crane i in.). Oczekuje się, że ta wartość będzie niższa na granicach TAD; w ten sposób można użyć standardowych technik statystycznych, aby znaleźć lokalne minima (granice) i zdefiniować regiony między kolejnymi granicami jako TAD.
Jednak, jak się dziś coraz częściej uznaje, TAD reprezentują hierarchiczną serię struktur, których nie można w pełni scharakteryzować za pomocą jednowymiarowych wyników uzyskanych poprzednimi metodami. Zwiększona rozdzielczość dostępna w nowszych zestawach danych może teraz wyraźnie odnosić się do TAD za pomocą podejść do analizy wieloskalowej. Jak po raz pierwszy wprowadził Armatus, można zidentyfikować domeny specyficzne dla rozdzielczości i obliczyć konsensusowy zestaw domen zachowanych w różnych rozdzielczościach, co przekształca problem wywołania TAD w optymalizację funkcji punktacji w oparciu o ich lokalne gęstości interakcji. Odmiany tego podejścia z różnymi funkcjami celu, takimi jak Lavaburst, MrTADFinder, 3DNetMod i Matryoshka, są również opracowywane w celu uzyskania lepszej wydajności obliczeniowej na zestawach danych o wyższej rozdzielczości.
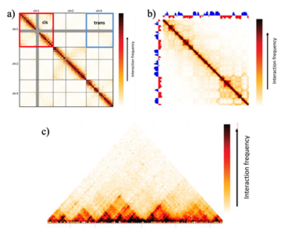
5. Interakcje punktowe
Z biologicznego punktu widzenia interakcje regulacyjne występują zwykle na znacznie mniejszą skalę niż TAD, a dwa elementy genomowe mogą aktywować/hamować ekspresję genu w odległości zaledwie 1 kb. Dlatego interakcje punktowe są ważne w interpretacji map Hi-C i oczekuje się, że pojawią się jako lokalne wzbogacenie prawdopodobieństwa kontaktu. Jednak obecne metodologie identyfikacji interakcji punktowych są z natury niejawne, ponieważ nie instruują, jak powinna wyglądać interakcja punktowa. Zamiast tego mutacje punktowe są identyfikowane jako wartości odstające z wyższymi częstotliwościami interakcji niż oczekiwano w macierzy Hi-C, biorąc pod uwagę, że model tła składa się tylko z najsilniejszych sygnałów, takich jak funkcje zaniku odległości. Model tła można oszacować i skonstruować przy użyciu zarówno lokalnych rozkładów sygnału, jak i podejść globalnych (tj. obejmujących cały chromosom/cały genom). Wiele z wyżej wymienionych pakietów bioinformatycznych zawiera algorytmy do identyfikacji interakcji punktowych. Krótko mówiąc, obliczana jest istotność poszczególnych interakcji parami, a znacząco wysokie wartości odstające są korygowane dla wielu testów, zanim zostaną uznane za prawdziwie informacyjne interakcje punktowe. Pomocne jest uzupełnienie zidentyfikowanych interakcji punktowych dodatkowymi dowodami, takimi jak analiza wyników wzbogacenia i powtórzeń biologicznych, aby wskazać, że te interakcje mają rzeczywiście znaczenie biologiczne.
Używa
Rozwój
1. Podział komórki
Hi-C może ujawnić zmiany konformacji chromatyny podczas podziału komórki. W interfazie chromatyny są na ogół luźne i żywe, dzięki czemu może zachodzić regulacja transkrypcji i inne działania regulacyjne. Wchodząc w mitozę i podział komórki, chromatyny ulegają zwartemu zwinięciu w gęste cylindryczne chromosomy. W ciągu ostatnich pięciu lat rozwój jednokomórkowej Hi-C umożliwił przedstawienie całego krajobrazu strukturalnego 3D chromatyn/chromosomów w całym cyklu komórkowym , a wiele badań wykazało, że te zidentyfikowane domeny genomowe pozostają niezmienione w interfazie i są usuwane przez mechanizmy wyciszania, gdy komórka wchodzi w mitozę. Kiedy podział mitotyczny jest zakończony i komórka ponownie wchodzi w interfazę, obserwuje się ponowne ustanowienie trójwymiarowych struktur chromatyny i przywrócenie regulacji transkrypcji.
2. Regulacja transkrypcji i determinacja losu
Podejrzewa się, że różnicowaniu embrionalnych komórek macierzystych (ESC) i indukowanych pluripotencjalnych komórek macierzystych (iPSC) w różne dojrzałe linie komórkowe towarzyszą globalne zmiany w strukturach chromosomów, a co za tym idzie, dynamika interakcji umożliwiająca regulację aktywacji/wyciszenia transkrypcji. Do zbadania tego pytania badawczego można użyć standardowego Hi-C.
W 2015 roku Dixon i in. zastosowali standard Hi-C, aby uchwycić globalną dynamikę 3D w ludzkich ESC podczas ich różnicowania w komórki z piątką . Ze względu na zdolność Hi-C do przedstawiania dynamicznych interakcji w TAD związanych z różnicowaniem, naukowcy odkryli wzrost liczby miejsc DHS, zdolności wiązania CTCF , aktywnych modyfikacji histonów i ekspresji genów docelowych w tych TAD będących przedmiotem zainteresowania i znaleźli znaczące udział głównych czynników pluripotencji , takich jak OCT4 , NANOG i SOX2 w sieci interakcji podczas komórek somatycznych . Od tego czasu Hi-C została uznana za jedną ze standardowych metod badania transkrypcyjnych czynności regulacyjnych i potwierdziła, że architektura chromosomu jest ściśle związana z losem komórki.
3. Wzrost i rozwój
Wzrost i rozwój somatyczny ssaków rozpoczyna się od zapłodnienia plemnika i oocytu , po którym następuje stadium zygoty , stadium 2-, 4- i 8-komórkowe, stadium blastocysty i wreszcie stadium zarodka . Hi-C umożliwił zbadanie kompleksowej architektury genomowej podczas wzrostu i rozwoju, ponieważ zarówno sis-Hi-C, jak i in situ Hi-C donoszą, że TAD i genomowe przedziały A i B nie są oczywiście obecne i wydają się być mniej dobrze - zbudowane w komórkach jajowych Te cechy strukturalne chromatyny dopiero stopniowo ustalają się od słabszych częstotliwości do czystszych i częstszych punktów danych po zapłodnieniu, w miarę postępu etapów rozwojowych.
Ewolucja genomu
Ponieważ dane dotyczące struktur genomu 3D stają się coraz bardziej rozpowszechnione w ostatnich latach, Hi-C zaczyna być używany jako środek do śledzenia ewolucyjnych cech/zmian strukturalnych. Genomowe polimorfizmy pojedynczego nukleotydu (SNP) i TAD są zazwyczaj konserwowane u różnych gatunków, wraz z czynnikiem CTCF w ewolucji domeny chromatyny. Jednak inne czynniki zostały ujawnione przez techniki Hi-C, aby doświadczyć ewolucji strukturalnej w architekturze 3D. Obejmują one użycia kodonów (CUFS), współregulację genów paralogowych i współewoluujące przestrzennie moduły ortologiczne (SCOM). W przypadku ewolucji domen na dużą skalę, translokacje chromosomalne , regiony synteniczne, jak również regiony rearanżacji genomu były względnie konserwatywne. Odkrycia te sugerują, że technologie Hi-C są w stanie zapewnić alternatywny punkt widzenia na eukariotyczne drzewo życia.
Rak
W kilku badaniach wykorzystano Hi-C do opisania i zbadania architektury chromatyny w różnych nowotworach i ich wpływu na patogenezę choroby. Kloetgen i in. użyty in situ Hi-C do badania ostrej białaczki limfoblastycznej z komórek T (T-ALL) i odkrył fuzję TAD, która usunęła miejsce izolacji CTCF, umożliwiając promotorowi onkogenu MYC bezpośrednią interakcję z dystalnym superwzmacniaczem . Fang i in. pokazali również, w jaki sposób następuje specyficzny wzrost lub utrata izolacji chromatyny dla T-ALL, co zmienia siłę architektury TAD genomu, przy użyciu in situ Hi-C. Low-C zastosowano do mapowania struktury chromatyny pierwotnych komórek B rozlanego chłoniaka z dużych komórek B pacjenta i został wykorzystany do znalezienia dużej zmienności strukturalnej chromosomów między pacjentem a zdrowymi komórkami B. Ogólnie rzecz biorąc, zastosowanie Hi-C i jego wariantów w badaniach nad rakiem zapewnia unikalny wgląd w molekularne podstawy czynników napędzających nieprawidłowości komórek. Może pomóc w wyjaśnieniu zjawisk biologicznych (wysoka ekspresja MYC w T-ALL) i pomóc w opracowaniu leku ukierunkowanego na mechanizmy unikalne dla komórek rakowych.
- ; ^ a b c d e f g hi j k l m n o Lieberman -Aiden, E van Berkum, Holandia; Williams, L; Imakajew, M; Ragoczy, T; Mówiąc, A; Amit, ja; Lajoie, BR; Sabo, PJ; Dorschner, MO; Sandstrom, R; Bernstein, B .; Bender, MA; Groudine, M ; Gnirke, A; Stamatoyannopoulos, J ; Mirny, Luizjana; Lander, Hiszpania ; Dekker, J (9 października 2009). „Kompleksowe mapowanie interakcji dalekiego zasięgu ujawnia zasady fałdowania ludzkiego genomu” . nauka . 326 (5950): 289–93. Bibcode : 2009Sci...326..289L . doi : 10.1126/science.1181369 . PMC 2858594 . PMID 19815776 .
- ^ a b c d Lin, Da; Hong, Ping; Zhang, Siheng; Xu, Weize; Jamal, Mahomet; Yan, Keji; Lei, Yingying; Li, Liang; Ruan, Yijun; Fu, Zhen F.; Li, Guoliang; Cao, Gang (maj 2018). „Hi-C tylko do trawienia i ligacji jest wydajną i opłacalną metodą wychwytywania konformacji chromosomów” . Genetyka przyrody . 50 (5): 754–763. doi : 10.1038/s41588-018-0111-2 . ISSN 1546-1718 . PMID 29700467 . S2CID 13740808 .
- ; ^ a b c d e f g hi j k l m n o p q r s t u v w x y z Kong, Siyuan Zhang, Yubo (1 lutego 2019). „Rozszyfrowanie Hi-C: od genomu 3D do funkcji” . Biologia komórki i toksykologia . 35 (1): 15–32. doi : 10.1007/s10565-018-09456-2 . ISSN 1573-6822 . PMID 30610495 . S2CID 57427743 .
- ^ a b c d e f g h i j k l m n o p q r s t u v w x y z aa ab ac ad ae af ag ah ai aj ak al am an ao ap aq ar jak w au av aw topór ay az ba bb bc bd be bf bg bh bi bj bk bl bm bn bo bp bq br bs bt bu bv bw bx autor: Belton, Jon-Matthew; McCord, Rachel Patton; Gibcus, Johan; Naumowa, Natalia; Zhan, Ye; Dekker, Praca (listopad 2012). „Hi-C: kompleksowa technika wychwytywania konformacji genomów” . Metody . 58 (3): 268–276. doi : 10.1016/j.ymet.2012.05.001 . ISSN 1046-2023 . PMC 3874846 . PMID 22652625 .
- ^ a b Eagen, Kyle P. (czerwiec 2018). „Zasady architektury chromosomów ujawnione przez Hi-C” . Trendy w naukach biochemicznych . 43 (6): 469–478. doi : 10.1016/j.tibs.2018.03.006 . ISSN 0968-0004 . PMC 6028237 . PMID 29685368 .
- ^ ab Kim , Kyukwang; Kim, Mooyoung; Kim, Jubin; Lee, Dongsung; Jung, Inkyung (1 stycznia 2022). „Hi-C jako dalmierz molekularny do badania rearanżacji genomowych” . Seminaria z biologii komórki i rozwoju . 121 : 161–170. doi : 10.1016/j.semcdb.2021.04.024 . ISSN 1084-9521 . PMID 33992531 . S2CID 234746398 .
- ^ a b c d Jin, Fulai; Li, Yan; Dixon, Jesse R.; Selvaraj, Siddarth; Tak, Zhen; Lee, Ah Young; Jen, Chia-An; Schmitt, Anthony D.; Espinoza, Celso; Ren, Bing (14 listopada 2013). „Wysokiej rozdzielczości mapa trójwymiarowego interakcjomu chromatyny w komórkach ludzkich” . Natura . 503 (7475): 290–294. Bibcode : 2013Natur.503..290J . doi : 10.1038/natura12644 . ISSN 0028-0836 . PMC 3838900 . PMID 24141950 .
- ^ a b c d e f Dixon, JR; Selwaraj, S; Yue, F; Kim, A; Li, Y; Shen, Y; Hu, M; Liu, JS; Ren, B (11 kwietnia 2012). „Domeny topologiczne w genomach ssaków zidentyfikowane na podstawie analizy oddziaływań chromatyny” . Natura . 485 (7398): 376–80. Bibcode : 2012Natur.485..376D . doi : 10.1038/natura11082 . PMC 3356448 . PMID 22495300 .
- Bibliografia _ Yaffe, Eitan; Kenigsberg, Efraim; Bantignies, Frédéric; Leblanc, Benjamin; Hoichman, Michał; Parrinello, Hugues; Tanay, Amos; Cavalli, Giacomo (3 lutego 2012). „Trójwymiarowe fałdowanie i zasady organizacji funkcjonalnej genomu Drosophila” . komórka . 148 (3): 458–472. doi : 10.1016/j.cell.2012.01.010 . ISSN 0092-8674 . PMID 22265598 . S2CID 17364610 .
- ^ a b c d Dixon, Jesse R.; Jung, Inkyung; Selvaraj, Siddarth; Shen, Yin; Antosiewicz-Bourget, Jessica E.; Lee, Ah Young; Tak, Zhen; Kim, Audrey; Radżagopal, Nisza; Xie, Wei; Diao, Yarui; Liang, Jing; Zhao, Huimin; Łobanenkow, Wiktor W.; Ecker, Joseph R.; Thomson, James A.; Ren, Bing (luty 2015). „Reorganizacja architektury chromatyny podczas różnicowania komórek macierzystych” . Natura . 518 (7539): 331–336. Bibcode : 2015Natur.518..331D . doi : 10.1038/natura14222 . ISSN 1476-4687 . PMC 4515363 . PMID 25693564 .
- ^ a b c d Belaghzal, Houda; Dekker, Hiob; Gibcus, Johan H. (1 lipca 2017). „Hi-C 2.0: Zoptymalizowana procedura Hi-C do mapowania konformacji chromosomów w całym genomie w wysokiej rozdzielczości” . Metody . 123 : 56–65. doi : 10.1016/j.ymet.2017.04.004 . ISSN 1046-2023 . PMC 5522765 . PMID 28435001 .
- ^ abc Lafontaine , Denis L.; Yang, Liyan; Dekker, Hiob; Gibcus, Johan H. (lipiec 2021). „Hi-C 3.0: ulepszony protokół do przechwytywania konformacji chromosomów całego genomu” . Aktualne protokoły . 1 (7): e198. doi : 10.1002/cpz1.198 . ISSN 2691-1299 . PMC 8362010 . PMID 34286910 .
- ^ a b c d e f g hi j k l m n o Rao , Suhas SP; Huntley, Miriam H.; Durand, Newa C.; Stamenova, Elena K.; Boczkow, Iwan D.; Robinson, James T.; Sanborn, Adrian L.; Machol, Ido; Omer, Arina D.; Lander, Eric S.; Aiden, Erez Lieberman (18 grudnia 2014). „Trójwymiarowa mapa ludzkiego genomu w rozdzielczości Kilobase ujawnia zasady zapętlania chromatyny” . komórka . 159 (7): 1665-1680. doi : 10.1016/j.cell.2014.11.021 . ISSN 0092-8674 . PMC 5635824 . PMID 25497547 .
- ^ a b c d e f g hi Hoffman , Elizabeth A .; Frey, Brian L.; Smith, Lloyd M.; Auble, David T. (30 października 2015). „Sieciowanie formaldehydowe: narzędzie do badania kompleksów chromatyny” . Journal of Biological Chemistry . 290 (44): 26404–26411. doi : 10.1074/jbc.R115.651679 . ISSN 0021-9258 . PMC 4646298 . PMID 26354429 .
- ^ a b c d e f g h i j k l m n o p q r s t u v w x y z aa ab ac ad ae af ag ah ai aj ak al am van Berkum, Nynke L.; Lieberman-Aiden, Erez; Williams, Louise; Imakajew, Maksym; Gnirke, Andreas; Mirny, Leonid A.; Dekker, Hiob; Lander, Eric S. (6 maja 2010). „Hi-C: metoda badania trójwymiarowej architektury genomów” . Journal of Visualized Experiments (39): 1869. doi : 10.3791/1869 . ISSN 1940-087X . PMC 3149993 . PMID 20461051 .
- ; ^ a b c d e f g hi j k l m n o p q r Niu, Longjian Shen, Wei; Huang, Yingzhang; On, Na; Zhang, Yuedong; Słońce, Jialei; Wan, Jing; Jiang, Daxin; Yang, Manyun; Tse, Yu Chung; Li, Li; Hou, Chunhui (19 lipca 2019). „Przygotowanie biblioteki bez amplifikacji za pomocą SAFE Hi-C wykorzystuje produkty ligacji do głębokiego sekwencjonowania w celu ulepszenia tradycyjnej analizy Hi-C” . Biologia komunikacji . 2 (1): 267. doi : 10.1038/s42003-019-0519-y . ISSN 2399-3642 . PMC 6642088 . PMID 31341966 .
- Bibliografia _ Chan, GL; Haseltine, Waszyngton (10 maja 1985). „Egzonukleaza polimerazy DNA T4 (3' – 5'), enzym do wykrywania i oznaczania ilościowego stabilnych uszkodzeń DNA: przykład światła ultrafioletowego” . Badania kwasów nukleinowych . 13 (9): 3285–3304. doi : 10.1093/nar/13.9.3285 . ISSN 0305-1048 . PMC 341235 . PMID 2987881 .
- ^ abc Wenxiu ; Ma, Tak, Ferhat; Lee, Choli; Gulsoy, Gunhan; Deng, Xinxian; Gotuj, Savannah; Hesson, Jennifer; Cavanaugh, Christopher; Ware, Carol B.; Krumm, Anton; Shendure, Jay; Blau, Carl Anthony; Disteche, Christine M.; Szlachetny, William S.; Duan, Zhijun (styczeń 2015). „Mapy interakcji chromatyny w dokładnej skali ujawniają cis-regulacyjny krajobraz ludzkich genów lincRNA” . Metody natury . 12 (1): 71–78. doi : 10.1038/nmeth.3205 . ISSN 1548-7105 . PMC 4281301 . PMID 25437436 .
- ^ abc Mifsud , Borbala; Tavares-Cadete, Filipe; Młody, Alicja N.; Cukier, Robert; Schoenfelder, Stefan; Ferreira, Lauren; Wingett, Steven W.; Andrews, Szymon; Szary, William; Ewels, Philip A.; Herman, Bram; Happe, Scott; Higgs, Andy; LeProust, Emily; Następuje, George A.; Fraser, Piotr; Luscombe, Mikołaj M.; Osborne, Cameron S. (czerwiec 2015). „Mapowanie kontaktów promotora dalekiego zasięgu w komórkach ludzkich z przechwytywaniem Hi-C w wysokiej rozdzielczości” . Genetyka przyrody . 47 (6): 598–606. doi : 10.1038/ng.3286 . ISSN 1546-1718 . PMID 25938943 . S2CID 6036717 .
- ^ Cullen, Katherine E.; Kladde, Michael P.; Seyfred, Mark A. (9 lipca 1993). „Interakcja między regionami regulującymi transkrypcję chromatyny prolaktyny” . nauka . 261 (5118): 203–206. Bibcode : 1993Sci...261..203C . doi : 10.1126/science.8327891 . PMID 8327891 .
- ^ Zhou, Yufan; Cheng, Xiaolong; Yang, Yini; Li, Tian; Li, Jingwei; Huang, Tim H.-M.; Wang, Junbai; Lin, Shili; Jin, Victor X. (12 sierpnia 2020). „Modelowanie i analiza danych Hi-C przez HiSIF identyfikuje charakterystyczne pętle promotor-dystalne” . Medycyna genomowa . 12 (1): 69. doi : 10.1186/s13073-020-00769-8 . ISSN 1756-994X . PMC 7425017 . PMID 32787954 .
- ^ a b c d e f g h ja Díaz, Noelia; Kruse, Kai; Erdmann, Tabea; Staiger, Annette M.; Ott, niemiecki; Lenz, Georg; Vaquerizas, Juan M. (29 listopada 2018). „Analiza konformacji chromatyny pierwotnej tkanki pacjenta przy użyciu niskonakładowej metody Hi-C” . Komunikacja natury . 9 (1): 4938. Bibcode : 2018NatCo...9.4938D . doi : 10.1038/s41467-018-06961-0 . ISSN 2041-1723 . PMC 6265268 . PMID 30498195 .
- ^ a b c d de Souza, Natalie (wrzesień 2015). „Mapy Micro-C struktury genomu” . Metody natury . 12 (9): 812. doi : 10.1038/nmeth.3575 . ISSN 1548-7105 . PMID 26554092 . S2CID 5765554 .
- ^ a b c d Burgess, Darren J. (czerwiec 2020). „Struktura chromosomu w mikroskali” . Natura Recenzje Genetyka . 21 (6): 337. doi : 10.1038/s41576-020-0243-y . ISSN 1471-0064 . PMID 32346116 . S2CID 216560645 .
- ^ ab Hsieh, Tsung -Han S.; Cattoglio, Klaudia; Słobodyaniuk, Elena; Hansen, Anders S.; Rando, Oliver J.; Tjian, Robert; Darzacq, Xavier (7 maja 2020). „Rozwiązywanie trójwymiarowego krajobrazu fałdowania chromatyny ssaków związanej z transkrypcją” . Komórka molekularna . 78 (3): 539–553.e8. doi : 10.1016/j.molcel.2020.03.002 . ISSN 1097-2765 . PMC 7703524 . PMID 32213323 .
- ^ a b c d e f g h i j k l m n o p q r s t u v w x y z aa ab ac ad ae af ag ah ai aj ak al am an ao ap aq ar jak w au av aw topór ay az ba bb bc bd be bf Lajoie, Bryan R.; Dekker, Hiob; Kaplan, Noam (styczeń 2015). „Przewodnik autostopowicza po analizie Hi-C: praktyczne wskazówki” . Metody . 72 : 65–75. doi : 10.1016/j.ymet.2014.10.031 . ISSN 1046-2023 . PMC 4347522 . PMID 25448293 .
- ^ a b c d e f g h ja j k l m n o p q r s t u v w x y z aa ab ac ad ae af ag ah ai Pal, Koustav; Forcato, Mattia; Ferrari, Francesco (20 grudnia 2018). „Analiza Hi-C: od generowania danych do integracji” . Recenzje biofizyczne . 11 (1): 67–78. doi : 10.1007/s12551-018-0489-1 . ISSN 1867-2450 . PMC 6381366 . PMID 30570701 .
- ^ a b c Forcato, Mattia; Bicciato, Silvio (2021). Analiza obliczeniowa danych Hi-C . Przechwytywanie konformacji chromosomu: metody i protokoły . Metody w biologii molekularnej . Tom. 2157. Sp. z o.o. s. 103–125. doi : 10.1007/978-1-0716-0664-3_7 . ISBN 978-1-0716-0663-6 . PMID 32820401 . S2CID 221219811 .
- ^ a b c d e f g h ja Gong, Haiyan; Yang, Yi; Zhang, Sichen; Li, Minghong; Zhang, Xiaotong (1 stycznia 2021). „Zastosowanie analizy danych Hi-C i innych omicznych w badaniach nad ludzkim rakiem i różnicowaniem komórek” . Dziennik biotechnologii obliczeniowej i strukturalnej . 19 : 2070–2083. doi : 10.1016/j.csbj.2021.04.016 . ISSN 2001-0370 . PMC 8086027 . PMID 33995903 .
- ^ Langmead, Ben (grudzień 2010). „Dopasowywanie odczytów krótkich sekwencji z Bowtie” . Aktualne protokoły w bioinformatyce . 32 (1): Jednostka 11.7. doi : 10.1002/0471250953.bi1107s32 . hdl : 2027.42/137758 . ISSN 1934-3396 . PMC 3010897 . PMID 21154709 .
- Bibliografia _ Durbin, Richard (15 lipca 2009). „Szybkie i dokładne wyrównanie krótkiego odczytu z transformacją Burrowsa-Wheelera” . Bioinformatyka . 25 (14): 1754-1760. doi : 10.1093/bioinformatyka/btp324 . PMC 2705234 . PMID 19451168 .
- Bibliografia _ „Minimapa2: dopasowanie parami sekwencji nukleotydowych” . Bioinformatyka . 34 (18): 3094–3100. doi : 10.1093/bioinformatyka/bty191 . PMC 6137996 . PMID 29750242 .
- ^ a b Imakaev, Maxim; Fudenberg, Geoffrey; McCord, Rachel Patton; Naumowa, Natalia; Gołoborodko, Anton; Lajoie, Bryan R.; Dekker, Hiob; Mirny, Leonid A. (październik 2012). „Iteracyjna korekta danych Hi-C ujawnia cechy organizacji chromosomów” . Metody natury . 9 (10): 999–1003. doi : 10.1038/nmeth.2148 . ISSN 1548-7105 . PMC 3816492 . PMID 22941365 .
- ^ a b Sługa, Nicolas; Varoquaux, Nelle; Lajoie, Bryan R.; Viara, Eric; Chen, Chong-Jian; Vert, Jean-Philippe; Słyszałem, Edyto; Dekker, Hiob; Barillot, Emmanuel (1 grudnia 2015). „HiC-Pro: zoptymalizowany i elastyczny potok do przetwarzania danych Hi-C” . Biologia genomu . 16 (1): 259. doi : 10.1186/s13059-015-0831-x . ISSN 1474-760X . PMC 4665391 . PMID 26619908 .
- ^ Hwang, Yih-Chii; Lin, Chiao-Feng; Valladares, Otto; Malamon, Jan; Kuksa, Paweł P.; Zheng, Qi; Gregory, Brian D.; Wang, Li-San (15 kwietnia 2015). „HIPPIE: wysokoprzepustowy rurociąg identyfikacyjny dla elementów wzmacniających oddziałujących z promotorem” . Bioinformatyka . 31 (8): 1290–1292. doi : 10.1093/bioinformatyka/btu801 . ISSN 1367-4803 . PMC 4393516 . PMID 25480377 .
- Bibliografia _ Węgorze, Filip; Furlan-Magaril, Mayra; Nagano, Takashi; Schoenfelder, Stefan; Fraser, Piotr; Andrews, Simon (20 listopada 2015). „HiCUP: potok do mapowania i przetwarzania danych Hi-C” . F1000Badania . 4 : 1310. doi : 10.12688/f1000research.7334.1 . ISSN 2046-1402 . PMC 4706059 . PMID 26835000 .
- ^ Serra, François; Baù, Davide; Goodstadt, Mike; Castillo, Dawid; Filion, Guillaume J.; Marti-Renom, Marc A. (19 lipca 2017). „Automatyczna analiza i modelowanie 3D danych Hi-C przy użyciu TADbit ujawnia cechy strukturalne kolorów chromatyny muchy” . Biologia obliczeniowa PLOS . 13 (7): e1005665. Bibcode : 2017PLSCB..13E5665S . doi : 10.1371/journal.pcbi.1005665 . ISSN 1553-7358 . PMC 5540598 . PMID 28723903 .
- ^ Durand, Neva C .; Shamim, Muhammad S.; Machol, Ido; Rao, Suhas SP; Huntley, Miriam H.; Lander, Eric S.; Aiden, Erez Lieberman (27 lipca 2016). „Juicer zapewnia system jednym kliknięciem do analizowania eksperymentów Hi-C w rozdzielczości pętli” . Systemy komórkowe . 3 (1): 95–98. doi : 10.1016/j.cels.2016.07.002 . ISSN 2405-4712 . PMC 5846465 . PMID 27467249 .
- ^ Reiff, Sarah B.; Schroeder, Andrew J.; Kırlı, Koray; Cosolo, Andrea; Bakker, Klara; Lee, Soohyun; Wit, Aleksander D.; Bałaszow, Aleksander K.; Vitzthum, Carl; Ronchetti, William; Pitman, Kent M.; Johnson, Jeremy; Ehmsen, Shannon R.; Kerpedjiev, Piotr; Abdennur, Nezar; Imakajew, Maksym; Öztürk, Serkan Utku; Çamoğlu, Uğur; Mirny, Leonid A.; Gehlenborg, Nils; Alver, Burak H.; Park, Peter J. (2022). „Portal danych nukleomowych 4D jako źródło wyszukiwania i wizualizacji wybranych danych nukleomicznych” . Komunikacja natury . 13 (1): 2365. doi : 10.1038/s41467-022-29697-4 . PMC 9061818 . PMID 35501320 .
- Bibliografia _ „Dopasowywanie odczytów sekwencji, sekwencji klonowania i kontigów składania za pomocą BWA-MEM”. arXiv : 1303,3997 [ q-bio.GN ].
- Bibliografia _ Pieśń, L.; Wang, X.; Cheng, H.; Wang, C.; Meyer, Kalifornia; Liu, T.; Tang, M.; Aluru, S.; Yue, F.; Liu, XS; Li, H. (2021). „Szybkie wyrównanie i wstępne przetwarzanie profili chromatyny za pomocą Chromap” . Komunikacja natury . 12 (1): 6566. doi : 10.1038/s41467-021-26865-w . PMC 8589834 . PMID 34772935 .
- ^ Forcato, Mattia; Nicoletti, Chiara; Pal, Koustav; Livi, Carmen Maria; Ferrari, Francesco; Bicciato, Silvio (lipiec 2017). „Porównanie metod obliczeniowych do analizy danych Hi-C” . Metody natury . 14 (7): 679–685. doi : 10.1038/nmeth.4325 . ISSN 1548-7105 . PMC 5493985 . PMID 28604721 .
- ^ Nora, Elphège P.; Gołoborodko, Anton; Valton, Anne-Laure; Gibcus, Johan H.; Uebersohn, Alec; Abdennur, Nezar; Dekker, Hiob; Mirny, Leonid A.; Bruneau, Benoit G. (18 maja 2017). „Ukierunkowana degradacja CTCF oddziela lokalną izolację domen chromosomów od kompartmentalizacji genomu” . komórka . 169 (5): 930–944.e22. doi : 10.1016/j.cell.2017.05.004 . ISSN 0092-8674 . PMC 5538188 . PMID 28525758 .
- Bibliografia _ Yin, Xianchen; Xu, Bingxiang; Wang, Danyang; Han, Jimin; Wei, Yi; Deng, Yun; Xiong, Ying; Zhang, Zhihua (15 sierpnia 2018). „Dekodowanie domen wiążących topologicznie z danymi Hi-C o bardzo niskiej rozdzielczości za pomocą entropii strukturalnej wykresu” . Komunikacja natury . 9 (1): 3265. Bibcode : 2018NatCo...9.3265L . doi : 10.1038/s41467-018-05691-7 . ISSN 2041-1723 . PMC 6093941 . PMID 30111883 .
-
^
Sauria, Michael EG; Taylor, James (14 listopada 2017). „QuASAR: ocena jakości odtwarzalności układu przestrzennego w danych Hi-C” : 204438. doi : 10.1101/204438 . S2CID 90376810 .
{{ cite journal }}: Cite journal wymaga|journal=( pomoc ) - ^ Ramirez, Fidel; Lingg, Tomasz; Toscano, Sarah; Lam, Kin Chung; Georgiew, Plamen; Chung, Ho-Ryun; Lajoie, Bryan; de Wit, Elzo; Zhan, Ye; de Laat, Wouter; Dekker, Hiob; Manke, Thomas; Akhtar, Asifa (1 października 2015). „Miejsca o wysokim powinowactwie tworzą sieć interakcji, aby ułatwić rozprzestrzenianie się kompleksu MSL na chromosomie X u Drosophila” . Komórka molekularna . 60 (1): 146–162. doi : 10.1016/j.molcel.2015.08.024 . ISSN 1097-2765 . PMC 4806858 . PMID 26431028 .
- ^ ab ; Zhang, Yan An, Lin; Xu, Jie; Zhang, Bo; Zheng, W. Jim; Hu, Ming; Tang, Jijun; Yue, Feng (21 lutego 2018). „Zwiększenie rozdzielczości danych Hi-C za pomocą głębokiej konwolucyjnej sieci neuronowej HiCPlus” . Komunikacja natury . 9 (1): 750. Bibcode : 2018NatCo...9..750Z . doi : 10.1038/s41467-018-03113-2 . ISSN 2041-1723 . PMC 5821732 . PMID 29467363 .
- ^ a b c Knopp, Paweł; Sinkhorn, Richard (styczeń 1967). „O macierzach nieujemnych i macierzach podwójnie stochastycznych” . Pacific Journal of Mathematics . 21 (2): 343–348. doi : 10.2140/pjm.1967.21.343 . ISSN 0030-8730 .
- Bibliografia _ Marie-Nelly, Hervé; Marbuty, Martial; Koszul, Romain; Mozziconacci, Julien (30 sierpnia 2012). „Normalizacja mapy kontaktów chromosomów” . Genomika BMC . 13 : 436. doi : 10.1186/1471-2164-13-436 . ISSN 1471-2164 . PMC 3534615 . PMID 22935139 .
- ^ Rycerz Filip; Ruiz, Daniel (26 października 2012). „Szybki algorytm równoważenia macierzy” . IMA Journal of numerical Analysis . 33 (3): 1029–1047. doi : 10.1093/imanum/drs019 .
- ^ abc Samuele ; Raccosta, Librizzi, Fabio; Jagger, Alistair M.; Noto, Rozyna; Martorana, Vincenzo; Lomas, David A.; Irving, James A.; Manno, Mauro (styczeń 2021). „Koncepcje skalowania w fizyce polimerów Serpin” . Materiały . 14 (10): 2577. Bibcode : 2021Mate...14.2577R . doi : 10.3390/ma14102577 . ISSN 1996-1944 . PMC 8156723 . PMID 34063488 .
- ^ abc Fudenberg , Geoffrey; Mirny, Leonid A (1 kwietnia 2012). „Struktura chromatyny wyższego rzędu: fizyka pomostowa i biologia” . Aktualna opinia w genetyce i rozwoju . 22 (2): 115–124. doi : 10.1016/j.gde.2012.01.006 . hdl : 1721.1/103044 . ISSN 0959-437X . PMC 3697851 . PMID 22360992 .
- Bibliografia _ Benner, Krzysztof; Spann, Natanael; Bertolino, Eric; Lin, Yin C.; Laslo, Piotr; Cheng, Jason X.; Murre, Cornelis; Singh, Harinder; Szkło, Christopher K. (28 maja 2010). „Proste kombinacje czynników transkrypcyjnych określających linię pierwotną elementów cis-regulatorowych wymaganych do tożsamości makrofagów i komórek B” . Komórka molekularna . 38 (4): 576–589. doi : 10.1016/j.molcel.2010.05.004 . ISSN 1097-2765 . PMC 2898526 . PMID 20513432 .
- ^ Zheng, Xiaobin; Zheng, Yixian (1 maja 2018). „CscoreTool: szybka analiza przedziału Hi-C w wysokiej rozdzielczości” . Bioinformatyka . 34 (9): 1568-1570. doi : 10.1093/bioinformatyka/btx802 . ISSN 1367-4803 . PMC 5925784 . PMID 29244056 .
- ^ de Wit, Elzo (7 lutego 2020). „TAD, jak nazywa je dzwoniący” . Journal of Molecular Biology . 432 (3): 638–642. doi : 10.1016/j.jmb.2019.09.026 . ISSN 0022-2836 . PMID 31654669 . S2CID 204918507 .
- ^ Naumowa, Natalia; Imakajew, Maksym; Fudenberg, Geoffrey; Zhan, Ye; Lajoie, Bryan R.; Mirny, Leonid A.; Dekker, Hiob (22 listopada 2013). „Organizacja chromosomu mitotycznego” . nauka . 342 (6161): 948–953. Bibcode : 2013Sci...342..948N . doi : 10.1126/science.1236083 . ISSN 0036-8075 . PMC 4040465 . PMID 24200812 .
- ^ abc Żuraw , Emily; Bian, Qian; McCord, Rachel Patton; Lajoie, Bryan R.; Wheeler, Bayly S.; Ralston, Edward J.; Uzawa, Satoru; Dekker, Hiob; Meyer, Barbara J. (lipiec 2015). „Przebudowa sterowana kondensacją topologii chromosomu X podczas kompensacji dawki” . Natura . 523 (7559): 240–244. Bibcode : 2015Natur.523..240C . doi : 10.1038/natura14450 . ISSN 1476-4687 . PMC 4498965 . PMID 26030525 .
- ^ a b c Filippova, Darya; Patro, Rob; Duggal, Geet; Kingsford, Carl (3 maja 2014). „Identyfikacja alternatywnych domen topologicznych w chromatynie” . Algorytmy dla biologii molekularnej . 9 (1): 14. doi : 10.1186/1748-7188-9-14 . ISSN 1748-7188 . PMC 4019371 . PMID 24868242 .
- Bibliografia _ Abdennur, Nezar; Gołoborodko, Anton; Pekowska, Aleksandra; Fudenberg, Geoffrey; Loe-Mie, Yann; Fonseca, Nuno A; Hubera, Wolfganga; Haering, chrześcijanin; Mirny, Leonid; Szpic, Francois (2 listopada 2017). „Dwa niezależne tryby organizacji chromatyny ujawnione przez usunięcie kohezyny” . Natura . 551 (7678): 51–56. Bibcode : 2017Natur.551...51S . doi : 10.1038/natura24281 . ISSN 0028-0836 . PMC 5687303 . PMID 29094699 .
- ^ Yan, Koon-Kiu; Lou, Shaoke; Gerstein, Mark (24 lipca 2017). „MrTADFinder: podejście oparte na modułowości sieci do identyfikacji topologicznie powiązanych domen w wielu rozdzielczościach” . Biologia obliczeniowa PLOS . 13 (7): e1005647. Bibcode : 2017PLSCB..13E5647Y . doi : 10.1371/journal.pcbi.1005647 . ISSN 1553-734X . PMC 5546724 . PMID 28742097 .
- ^ Norton, Heidi K.; Emerson, Daniel J.; Huang, Harvey; Kim, Jesi; Tytus, Katelyn R.; Gu, Shi; Bassett, Danielle S.; Phillips-Cremins, Jennifer E. (luty 2018). „Wykrywanie hierarchicznego fałdowania genomu z modułowością sieci” . Metody natury . 15 (2): 119–122. doi : 10.1038/nmeth.4560 . ISSN 1548-7105 . PMC 6029251 . PMID 29334377 .
- Bibliografia _ Patro, Rob (wrzesień 2019). „Przewidywanie struktury bogatej chromatyny na podstawie danych Hi-C” . Transakcje IEEE / ACM dotyczące biologii obliczeniowej i bioinformatyki . 16 (5): 1448–1458. doi : 10.1109/TCBB.2018.2851200 . ISSN 1557-9964 . PMID 29994683 . S2CID 54563346 .
- ^ ab Ou , Horng D.; Phan, Sebastien; Deerinck, Thomas J.; Thor, Andrea; Ellisman, Mark H.; O'Shea, Clodagh C. (28 lipca 2017). „ChromEMT: Wizualizacja struktury chromatyny 3D i zagęszczania komórek dziewięciofazowych i mitotycznych” . nauka . 357 (6349): eaag0025. doi : 10.1126/science.aag0025 . ISSN 0036-8075 . PMC 5646685 . PMID 28751582 .
- ^ ab Nagano , Takashi; Lubling, Janów; Várnai, Csilla; Dudley, Karmel; Leung, skrzydło; Baran, Yael; Mendelson Cohen, Netta; Wingett, Steven; Fraser, Piotr; Tanay, Amos (lipiec 2017). „Dynamika cyklu komórkowego organizacji chromosomów w rozdzielczości pojedynczej komórki” . Natura . 547 (7661): 61–67. Bibcode : 2017Natur.547...61N . doi : 10.1038/natura23001 . ISSN 1476-4687 . PMC 5567812 . PMID 28682332 .
- Bibliografia _ Mateo, Leslie J.; Su, Jun-Han; Sinnott-Armstrong, Nicholas A.; Parker, Mirae; Kinrot, Seon; Yamaya, Kei; Boettiger, Alistair N.; Zhuang, Xiaowei (26 października 2018). „Śledzenie chromatyny w super rozdzielczości ujawnia domeny i interakcje kooperacyjne w pojedynczych komórkach” . nauka . 362 (6413): eau1783. Bibcode : 2018Sci...362.1783B . doi : 10.1126/science.aau1783 . ISSN 0036-8075 . PMC 6535145 . PMID 30361340 .
- ^ ab Yu , Miao; Ren, Bing (6 października 2017). „Trójwymiarowa organizacja genomów ssaków” . Roczny przegląd biologii komórki i rozwoju . 33 (1): 265–289. doi : 10.1146/annurev-cellbio-100616-060531 . ISSN 1081-0706 . PMC 5837811 . PMID 28783961 .
- ^ Niakan, Kathy K.; Han, Jinnuo; Pedersen, Roger A.; Szymon, Karol; Pera, Renee A. Reijo (1 marca 2012). „Rozwój ludzkiego zarodka przed implantacją” . Rozwój . 139 (5): 829–841. doi : 10.1242/dev.060426 . ISSN 0950-1991 . PMC 3274351 . PMID 22318624 .
- ^ abc Zhenhai ; Du, Zheng, Hui; Huang, Bo; Mamo, Rui; Wu, Jingyi; Zhang, Xianglin; On, Jing; Xiang, Yunlong; Wang, Qiujun; Li, Yuanyuan; Mamo, Jing; Zhang, Xu; Zhang, Ke; Wang, Yang; Zhang, Michael Q.; Gao, Juntao; Dixon, Jesse R.; Wang, Xiaowo; Zeng, Jianyang; Xie, Wei (lipiec 2017). „Przeprogramowanie alleliczne architektury chromatyny 3D podczas wczesnego rozwoju ssaków” . Natura . 547 (7662): 232–235. Bibcode : 2017Natur.547..232D . doi : 10.1038/natura23263 . ISSN 1476-4687 . PMID 28703188 . S2CID 4410486 .
- ^ a b c Ke, Yuwen; Xu, Yanan; Chen, Xuepeng; Feng, Songjie; Liu, Zhenbo; Słońce, Yaoyu; Yao, Xuelong; Li, Fangzhen; Zhu, Wei; Gao, Lei; Chen, Haojie; Du, Zhenhai; Xie, Wei; Xu, Xiaocui; Huang, Xingxu; Liu, Jiang (13 lipca 2017). „Struktury chromatyny 3D dojrzałych gamet i przeprogramowanie strukturalne podczas embriogenezy ssaków” . komórka . 170 (2): 367–381.e20. doi : 10.1016/j.cell.2017.06.029 . ISSN 0092-8674 . PMID 28709003 . S2CID 23974814 .
- ^ Diament, Alon; Tuller, Tamir (1 czerwca 2019). „Modelowanie trójwymiarowej organizacji genomu w ewolucji i patogenezie” . Seminaria z biologii komórki i rozwoju . 90 : 78–93. doi : 10.1016/j.semcdb.2018.07.008 . ISSN 1084-9521 . PMID 30030143 . S2CID 51704135 .
- ^ ab Vietri , Matteo; Barrington, Christopher; Henderson, Stephen; Ernst, Krystyna; Odom, Duncan; Tanay, Amos; Hadjur, Suzana (26 lutego 2015). „Porównawcza Hi-C ujawnia, że CTCF leży u podstaw ewolucji architektury domeny chromosomalnej” . Raporty komórkowe . 10 (8): 1297–1309. doi : 10.1016/j.celrep.2015.02.004 . ISSN 2211-1247 . PMC 4542312 . PMID 25732821 .
- ^ Diament, Alon; Pinter, Ron Y.; Tuller, Tamir (16 grudnia 2014). „Trójwymiarowa organizacja genomu eukariotycznego jest silnie skorelowana z ekspresją i funkcją użycia kodonów” . Komunikacja natury . 5 (1): 5876. Bibcode : 2014NatCo...5.5876D . doi : 10.1038/ncomms6876 . ISSN 2041-1723 . PMID 25510862 .
- ^ Ibn-Salem, Jonasz; Muro, Enrique M.; Andrade-Navarro, Miguel A. (9 stycznia 2017). „Współregulacja genów paralogowych w trójwymiarowej architekturze chromatyny” . Badania kwasów nukleinowych . 45 (1): 81–91. doi : 10.1093/nar/gkw813 . ISSN 0305-1048 . PMC 5224500 . PMID 27634932 .
- ^ Diament, Alon; Tuller, Tamir (5 maja 2017). „Śledzenie ewolucji organizacji genów 3D pokazuje jej związek z dywergencją fenotypową” . Badania kwasów nukleinowych . 45 (8): 4330–4343. doi : 10.1093/nar/gkx205 . ISSN 0305-1048 . PMC 5416853 . PMID 28369658 .
- ^ Bonev, Boyan; Cavalli, Giacomo (listopad 2016). „Organizacja i funkcja genomu 3D” . Nature Recenzje Genetyka . 17 (11): 661–678. doi : 10.1038/nrg.2016.112 . ISSN 1471-0064 . PMID 27739532 . S2CID 31259189 .
- ^ Komory, Emily V .; Bickmore, Wendy A.; Semple, Colin A. (4 kwietnia 2013). „Rozbieżność struktury chromatyny wyższego rzędu ssaków jest powiązana z lokacjami rozwojowymi” . Biologia obliczeniowa PLOS . 9 (4): e1003017. Bibcode : 2013PLSCB...9E3017C . doi : 10.1371/journal.pcbi.1003017 . ISSN 1553-7358 . PMC 3617018 . PMID 23592965 .
- ^ abc Kloetgen , Andreas; Thandapani, Palaniradża; Ntziachristos, Panagiotis; Ghebrechristos, Yohana; Nomikou, Sofia; Łazaris, Charalampos; Chen, Xufeng; Hu, Hai; Bakogianni, Sofia; Wang, Jingjing; Fu, Yi; Boccalatte, Francesco; Zhong, Hua; Paietta, Elżbieta; Trimarchi, Tomasz; Zhu, Yixing; Van Vlierberghe, Pieter; Inghirami, Giorgio G.; Lionnet, Tymoteusz; Aifantis, Iannis; Tsirigos, Aristotelis (kwiecień 2020). „Trójwymiarowe krajobrazy chromatyny w ostrej białaczce limfoblastycznej z limfocytów T” . Genetyka przyrody . 52 (4): 388–400. doi : 10.1038/s41588-020-0602-9 . ISSN 1546-1718 . PMC 7138649 . PMID 32203470 .
- ^ a b c Fang, Celestia; Wang, Zhenjia; Han, Cuijuan; Safgren, Stephanie L.; Helmin, Kathryn A.; Adelman, Emmalee R.; Serafin, Walentyna; Basso Giuseppe; Eagen, Kyle P.; Gaspar-Maia, Alexandre; Figueroa, Maria E.; Piosenkarz, Benjamin D.; Ratan, Aakrosh; Ntziachristos, Panagiotis; Zang, Chongzhi (15 września 2020). „Swoiste dla raka wiązanie CTCF ułatwia onkogenną dysregulację transkrypcji” . Biologia genomu . 21 (1): 247. doi : 10.1186/s13059-020-02152-7 . ISSN 1474-760X . PMC 7493976 . PMID 32933554 .