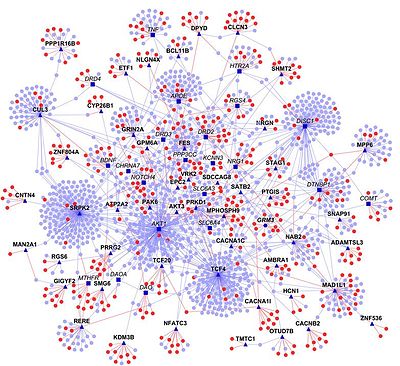
Interaktom
W biologii molekularnej interaktom to cały zestaw interakcji molekularnych w określonej komórce . Termin ten w szczególności odnosi się do fizycznych interakcji między cząsteczkami (takich jak interakcje między białkami, znane również jako interakcje białko-białko , PPI lub między małymi cząsteczkami a białkami), ale może również opisywać zestawy pośrednich interakcji między genami ( interakcje genetyczne ).

Słowo „interaktom” zostało pierwotnie ukute w 1999 roku przez grupę francuskich naukowców pod przewodnictwem Bernarda Jacqa. Z matematycznego punktu widzenia interaktomy są zwykle wyświetlane jako wykresy . Chociaż interaktomy można opisać jako sieci biologiczne , nie należy ich mylić z innymi sieciami, takimi jak sieci neuronowe czy sieci pokarmowe .
Sieci oddziaływań molekularnych
Interakcje molekularne mogą zachodzić między cząsteczkami należącymi do różnych rodzin biochemicznych (białka, kwasy nukleinowe, lipidy, węglowodany itp.), a także w obrębie danej rodziny. Ilekroć takie cząsteczki są połączone oddziaływaniami fizycznymi, tworzą sieci oddziaływań molekularnych, które są ogólnie klasyfikowane według natury zaangażowanych związków. Najczęściej interakcja odnosi się do sieci interakcji białko-białko (PPI) (PIN) lub jej podzbiorów. Na przykład interaktom białkowy Sirt-1 i interaktom drugiego rzędu z rodziny Sirt to sieć obejmująca Sirt-1 i jego bezpośrednio oddziałujące białka, gdzie jako interaktom drugiego rzędu ilustruje interakcje sąsiadów drugiego rzędu (sąsiedzi sąsiadów). Innym szeroko badanym typem interaktomu jest interakcja białkowo-DNA, zwana także a sieć regulatorowa genów , sieć utworzona przez czynniki transkrypcyjne, białka regulatorowe chromatyny i ich geny docelowe. Nawet sieci metaboliczne można uznać za sieci oddziaływań molekularnych: metabolity, czyli związki chemiczne w komórce, są przekształcane w siebie przez enzymy , które muszą fizycznie związać swoje substraty.
W rzeczywistości wszystkie typy interatomów są ze sobą połączone. Na przykład interakcja białkowa zawiera wiele enzymów, które z kolei tworzą sieci biochemiczne. Podobnie sieci regulacyjne genów pokrywają się zasadniczo z sieciami interakcji białek i sieciami sygnalizacyjnymi.
Rozmiar
Sugerowano, że rozmiar interakcjomu organizmu koreluje lepiej niż rozmiar genomu z biologiczną złożonością organizmu. Chociaż mapy interakcji białko-białko, zawierające kilka tysięcy interakcji binarnych, są obecnie dostępne dla kilku gatunków, żadna z nich nie jest obecnie kompletna, a rozmiar interakcji jest nadal przedmiotem dyskusji.
Drożdże
, że interaktom drożdży, tj. wszystkie interakcje białko-białko między białkami Saccharomyces cerevisiae , obejmuje od 10 000 do 30 000 interakcji. Rozsądne oszacowanie może być rzędu 20 000 interakcji. Większe szacunki często obejmują interakcje pośrednie lub przewidywane, często z oczyszczania powinowactwa / spektrometrii mas (AP/MS).
Sieci interakcji genetycznych
Geny oddziałują na siebie w tym sensie, że wpływają na swoje funkcje. Na przykład mutacja może być nieszkodliwa, ale w połączeniu z inną mutacją może okazać się śmiertelna. Mówi się, że takie geny „oddziałują genetycznie”. Połączone w ten sposób geny tworzą genetyczne sieci interakcji . Niektóre z celów tych sieci to: opracowanie funkcjonalnej mapy procesów zachodzących w komórce, identyfikacja celu leku za pomocą chemoproteomiki oraz przewidywanie funkcji niescharakteryzowanych genów.
W 2010 roku najbardziej „kompletny” interakcja genów wyprodukowany do tej pory został skompilowany na podstawie około 5,4 miliona porównań dwóch genów w celu opisania „profili interakcji dla ~ 75% wszystkich genów w pączkujących drożdżach ", z około 170 000 interakcji genów. Geny pogrupowano na podstawie podobnych funkcji, aby zbudować funkcjonalną mapę procesów zachodzących w komórce. Dzięki tej metodzie badanie było w stanie przewidzieć znane funkcje genów lepiej niż jakikolwiek inny zestaw danych w skali genomu jako jak również dodawanie informacji funkcjonalnych dla genów, które nie zostały wcześniej opisane. Z tego modelu można obserwować interakcje genetyczne w wielu skalach, co pomoże w badaniu koncepcji, takich jak konserwacja genów. Niektóre z obserwacji poczynionych w ramach tego badania są takie, że istnieje było dwa razy więcej negatywnych niż pozytywnych interakcji , negatywne interakcje były bardziej pouczające niż pozytywne, a geny z większą liczbą połączeń częściej powodowały śmiertelność po zakłóceniu.
Interaktomika
Interaktomika to dyscyplina z pogranicza bioinformatyki i biologii , która zajmuje się badaniem zarówno interakcji, jak i konsekwencji tych interakcji między białkami i innymi cząsteczkami w komórce . Interactomics ma zatem na celu porównanie takich sieci interakcji (tj. interaktomów) między gatunkami iw ich obrębie, aby dowiedzieć się, w jaki sposób cechy takich sieci są zachowywane lub zróżnicowane.
Interactomics jest przykładem biologii systemowej „z góry na dół”, która obejmuje ogólny widok biosystemu lub organizmu. Gromadzone są duże zestawy danych dotyczących całego genomu i danych proteomicznych, po czym wnioskuje się o korelacjach między różnymi cząsteczkami. Na podstawie danych formułowane są nowe hipotezy dotyczące sprzężeń zwrotnych między tymi cząsteczkami. Hipotezy te można następnie zweryfikować za pomocą nowych eksperymentów.
Eksperymentalne metody mapowania interaktomów
Badanie interaktomów nazywa się interakcją. Podstawową jednostką sieci białkowej jest interakcja białko-białko (PPI). Chociaż istnieje wiele metod badania PPI, stosunkowo niewiele z nich zastosowano na dużą skalę do mapowania całych interakcji.
Drożdżowy system dwóch hybryd (Y2H) nadaje się do badania interakcji binarnych między dwoma białkami jednocześnie. Oczyszczanie metodą powinowactwa, a następnie spektrometria mas jest odpowiednia do identyfikacji kompleksu białkowego. Obie metody mogą być używane w sposób o dużej przepustowości (HTP). Dwa hybrydowe ekrany drożdży umożliwiają fałszywie dodatnie interakcje między białkami, które nigdy nie ulegają ekspresji w tym samym czasie i miejscu; Spektrometria mas z wychwytem powinowactwa nie ma tej wady i jest obecnie złotym standardem. Dane z dwóch hybryd drożdży lepiej wskazują na niespecyficzne tendencje do interakcji lepkich, podczas gdy spektrometria mas z wychwytem powinowactwa lepiej wskazuje na funkcjonalne interakcje białko-białko in vivo.
Metody obliczeniowe do badania interakcji
Po utworzeniu interakcjomu istnieje wiele sposobów analizy jego właściwości. Istnieją jednak dwa ważne cele takich analiz. W pierwszej kolejności naukowcy starają się wyjaśnić właściwości systemowe interaktomów, np. topologię ich interakcji. Po drugie, badania mogą koncentrować się na poszczególnych białkach i ich roli w sieci. Analizy takie przeprowadzane są głównie bioinformatycznymi i obejmują między innymi:
Walidacja
Najpierw należy ocenić zasięg i jakość interakcjomu. Interaktomy nigdy nie są kompletne, biorąc pod uwagę ograniczenia metod eksperymentalnych. Na przykład oszacowano, że typowe Y2H wykrywają tylko około 25% wszystkich interakcji w interakcjom. Zasięg interaktomu można ocenić, porównując go z wzorcami dobrze znanych interakcji, które zostały znalezione i potwierdzone w niezależnych testach. Inne metody odfiltrowują fałszywe alarmy, obliczając podobieństwo znanych adnotacji zaangażowanych białek lub definiując prawdopodobieństwo interakcji przy użyciu subkomórkowej lokalizacji tych białek.
Przewidywanie PPI
Wykorzystując dane eksperymentalne jako punkt wyjścia, przeniesienie homologii jest jednym ze sposobów przewidywania interakcji. W tym przypadku PPI z jednego organizmu są wykorzystywane do przewidywania interakcji między białkami homologicznymi w innym organizmie („ interologi ”). Podejście to ma jednak pewne ograniczenia, przede wszystkim dlatego, że dane źródłowe mogą nie być wiarygodne (np. zawierać wyniki fałszywie dodatnie i fałszywie ujemne). Ponadto białka i ich interakcje zmieniają się podczas ewolucji, a zatem mogły zostać utracone lub zdobyte. Niemniej jednak przewidziano liczne interaktomy, np. u Bacillus licheniformis .
Niektóre algorytmy wykorzystują dowody eksperymentalne dotyczące kompleksów strukturalnych, szczegóły atomowe interfejsów wiążących i tworzą szczegółowe modele atomowe kompleksów białko-białko, a także inne interakcje białko-cząsteczka. Inne algorytmy wykorzystują tylko informacje o sekwencji, tworząc w ten sposób bezstronne, kompletne sieci interakcji z wieloma błędami.
Niektóre metody wykorzystują uczenie maszynowe do rozróżnienia, w jaki sposób pary białek oddziałujących na siebie różnią się od par białek niewchodzących w interakcje pod względem cech parami, takich jak kolokalizacja komórkowa, koekspresja genów, jak blisko DNA znajdują się geny kodujące te dwa białka itd. NA. Stwierdzono, że Random Forest jest najskuteczniejszą metodą uczenia maszynowego do przewidywania interakcji białek. Takie metody zastosowano do odkrywania interakcji białkowych na ludzkim interakcjom, w szczególności interaktomu białek błonowych i interakcjomu białek związanych ze schizofrenią.
Eksploracja tekstu PPI
Podjęto pewne wysiłki, aby systematycznie wyodrębnić sieci interakcji bezpośrednio z literatury naukowej. Takie podejścia różnią się pod względem złożoności, od prostych statystyk współwystępowania podmiotów, które są wymieniane razem w tym samym kontekście (np. zdanie), po wyrafinowane przetwarzanie języka naturalnego i metody uczenia maszynowego do wykrywania relacji interakcji.
Przewidywanie funkcji białek
Sieci interakcji białek zostały wykorzystane do przewidywania funkcji białek o nieznanych funkcjach. Zwykle opiera się to na założeniu, że niescharakteryzowane białka mają podobne funkcje, jak ich białka oddziałujące ( wina przez asocjację ). Na przykład stwierdzono, że YbeB, białko o nieznanej funkcji, oddziałuje z białkami rybosomalnymi, a później wykazano, że bierze udział w translacji bakteryjnej i eukariotycznej (ale nie archeonów) . Chociaż takie prognozy mogą opierać się na pojedynczych interakcjach, zwykle znajduje się kilka interakcji. W ten sposób cała sieć interakcji może być wykorzystana do przewidywania funkcji białek, biorąc pod uwagę, że pewne funkcje są zwykle wzbogacane wśród interaktorów. Termin hypothome został użyty do określenia interakcjomu, w którym co najmniej jeden z genów lub białek jest hipotetycznym białkiem .
Perturbacje i choroby
Topologia interakcjomu umożliwia przewidywanie, w jaki sposób sieć reaguje na zakłócenia (np . usuwanie) węzłów (białek) lub krawędzi (interakcje). Takie zaburzenia mogą być spowodowane mutacjami genów, a co za tym idzie ich białek, a reakcja sieciowa może objawiać się chorobą . Analiza sieciowa może zidentyfikować cele leków i biomarkery chorób.
Struktura i topologia sieci
Sieci interakcji można analizować za pomocą narzędzi teorii grafów . Właściwości sieci obejmują stopni , współczynniki grupowania , centralność pomiędzy i wiele innych. Rozkład właściwości między białkami interakcjomu ujawnił, że sieci interaktomów często mają topologię bez skali , w której moduły funkcjonalne w sieci wskazują wyspecjalizowane podsieci. Takie moduły mogą być funkcjonalne, jak w ścieżce sygnalizacyjnej lub strukturalny, jak w kompleksie białkowym. W rzeczywistości identyfikacja kompleksów białkowych w interaktomie jest trudnym zadaniem, biorąc pod uwagę, że sama sieć nie ujawnia bezpośrednio obecności stabilnego kompleksu.
Studiował interaktomy
Interaktomy wirusowe
Wirusowe interakcje białkowe składają się z interakcji między białkami wirusowymi lub fagowymi. Byli jednymi z pierwszych projektów interaktomowych, ponieważ ich genomy są małe, a wszystkie białka można analizować przy ograniczonych zasobach. Wirusowe interaktomy są połączone z interakcjami swojego gospodarza, tworząc sieci interakcji wirus-gospodarz. Niektóre opublikowane interakcje wirusów obejmują
Fag
- Bakteriofag lambda Escherichia coli
- Escherichia coli T7
- Streptococcus pneumoniae Dp-1
- Streptococcus pneumoniae Cp-1
Interaktomy lambda i VZV są nie tylko istotne dla biologii tych wirusów, ale także z powodów technicznych: były pierwszymi interakcjami, które zostały zmapowane za pomocą wielu wektorów Y2H , co dowodzi ulepszonej strategii pełniejszego badania interakcji, niż wykazały poprzednie próby.
Wirusy ludzkie (ssacze).
- Ludzki wirus ospy wietrznej i półpaśca (VZV)
- Wirus Chandipura
- Wirus Epsteina-Barra (EBV)
- Wirus zapalenia wątroby typu C (HPC), interakcje człowiek-HCV
- Wirus zapalenia wątroby typu E (HEV)
- Wirus opryszczki pospolitej 1 (HSV-1)
- Herpeswirus związany z mięsakiem Kaposiego (KSHV)
- Mysi wirus cytomegalii (mCMV)
Interaktomy bakteryjne
Stosunkowo niewiele bakterii zostało kompleksowo zbadanych pod kątem ich interakcji białko-białko. Jednak żaden z tych interakcji nie jest kompletny w tym sensie, że uchwycił wszystkie interakcje. W rzeczywistości oszacowano, że żadne z nich nie obejmuje więcej niż 20% lub 30% wszystkich interakcji, głównie dlatego, że w większości z tych badań zastosowano tylko jedną metodę, z których wszystkie odkrywają tylko podzbiór interakcji. Wśród opublikowanych interakcji bakteryjnych (w tym częściowych) znajdują się m.in
| Gatunek | białka ogółem | interakcje | typ | odniesienie |
| Helicobacter pylori | 1553 | ~ 3004 | R2H | |
| Campylobacter jejuni | 1623 | 11687 | R2H | |
| Treponema blada | 1040 | 3649 | R2H | |
| Escherichia coli | 4288 | (5993) | AP/MS | |
| Escherichia coli | 4288 | 2234 | R2H | |
| Mesorhizobium loti | 6752 | 3121 | R2H | |
| Prątek gruźlicy | 3959 | >8000 | B2H | |
| Mycoplasma genitalium | 482 | AP/MS | ||
| Synechocystis sp. PCC6803 | 3264 | 3236 | R2H | |
| Gronkowiec złocisty (MRSA) | 2656 | 13219 | AP/MS |
Interaktomy E. coli i Mycoplasma zostały przeanalizowane przy użyciu oczyszczania powinowactwa kompleksów białkowych na dużą skalę i spektrometrii mas (AP / MS), dlatego nie jest łatwo wywnioskować bezpośrednie interakcje. Inni wykorzystali obszerne dwuhybrydowe drożdży (Y2H). Interakcja Mycobacterium tuberculosis została przeanalizowana przy użyciu bakteryjnego ekranu dwuhybrydowego (B2H).
Należy zauważyć, że za pomocą metod obliczeniowych przewidziano wiele dodatkowych interakcji (patrz sekcja powyżej).
Interaktory eukariotyczne
Podjęto kilka prób mapowania interakcji eukariotycznych metodami HTP. Chociaż żadne biologiczne interaktomy nie zostały w pełni scharakteryzowane, przebadano ponad 90% białek w Saccharomyces cerevisiae i scharakteryzowano ich interakcje, co czyni go najlepiej scharakteryzowanym interaktomem. Gatunki, których interaktomy zostały szczegółowo zbadane, obejmują
Niedawno interakcja patogen-gospodarz wirusa zapalenia wątroby typu C/człowieka (2008), wirusa Epsteina Barra/człowieka (2008), wirusa grypy/człowieka (2009) została wyznaczona za pomocą HTP w celu zidentyfikowania podstawowych składników molekularnych patogenów i układu odpornościowego gospodarza .
Przewidywane interaktomy
Jak opisano powyżej, można przewidzieć PPI, a tym samym całe interaktomy. Chociaż wiarygodność tych przewidywań jest dyskusyjna, dostarczają one hipotez, które można przetestować eksperymentalnie. Interaktomy przewidziano dla wielu gatunków, np
- Człowiek ( Homo sapiens )
- Ryż ( Oryza sativa )
- Xanthomonas oryzae
- Arabidopsis thaliana
- Pomidor zwyczajny ( Solanum lycopersicum )
- Gorczyca polna ( Brassica rapa )
- Kukurydza, kukurydza ( Zea mays )
- Topola ( Populus trichocarpa )
- SARS-CoV-2
Właściwości sieciowe
Sieci interakcji białek można analizować za pomocą tego samego narzędzia, co inne sieci. W rzeczywistości mają wiele wspólnych właściwości z sieciami biologicznymi lub społecznymi . Niektóre z głównych cech są następujące.

Dystrybucja stopni
Rozkład stopni opisuje liczbę białek, które mają określoną liczbę połączeń. Większość sieci interakcji białek wykazuje bez skali ( prawo potęgowe ), gdzie rozkład łączności P(k) ~ k −γ , gdzie k jest stopniem. Zależność tę można również postrzegać jako linię prostą na wykresie logarytmicznym , ponieważ powyższe równanie jest równe log(P(k)) ~ —y•log(k). Jedną z cech takich rozkładów jest to, że istnieje wiele białek z kilkoma interakcjami i niewiele białek z wieloma interakcjami, przy czym te ostatnie nazywane są „hubami”.
Piasty
Silnie połączone węzły (białka) nazywane są koncentratorami. Han i in. ukuli termin „ centrum imprezowe ” dla centrów, których ekspresja jest skorelowana z partnerami interakcji. Piasty imprezowe łączą również białka w modułach funkcjonalnych, takich jak kompleksy białkowe. W przeciwieństwie do „ centrów randkowych „ nie wykazują takiej korelacji i wydają się łączyć różne moduły funkcjonalne. Koncentratory stron znajdują się głównie w zestawach danych AP/MS, podczas gdy koncentratory danych znajdują się głównie w binarnych interaktywnych mapach sieciowych. Należy zauważyć, że ważność koncentratora daty/koncentratora imprezy rozróżnienie było kwestionowane. Piasty stron składają się na ogół z białek wielointerfejsowych, podczas gdy piasty dat są częściej białkami interfejsów pojedynczej interakcji. Zgodnie z rolą koncentratorów dat w łączeniu różnych procesów, u drożdży liczba binarnych interakcji danego białka wynosi skorelowane z liczbą fenotypów obserwowanych dla odpowiedniego zmutowanego genu w różnych warunkach fizjologicznych.
Moduły
Węzły zaangażowane w ten sam proces biochemiczny są ze sobą silnie powiązane.
Ewolucja
Ewolucję złożoności interaktomu opisano w badaniu opublikowanym w Nature . W tym badaniu po raz pierwszy zauważono, że granicom między prokariotami , jednokomórkowymi eukariotami i wielokomórkowymi eukariotami towarzyszą redukcje efektywnej wielkości populacji o rzędy wielkości, z jednoczesnym wzmocnieniem skutków losowego dryfu genetycznego . Wynikający z tego spadek wydajności selekcji wydaje się być wystarczający, aby wpływać na szeroki zakres atrybutów na poziomie genomowym w sposób nieadaptacyjny. Badanie Nature pokazuje, że zmienność siły losowego dryfu genetycznego może również wpływać na różnorodność filogenetyczną na poziomie subkomórkowym i komórkowym. W związku z tym wielkość populacji należałoby uznać za potencjalny wyznacznik mechanistycznych ścieżek leżących u podstaw długoterminowej ewolucji fenotypowej. W badaniu wykazano ponadto, że istnieje filogenetycznie szeroka odwrotna zależność między siłą dryfu a integralnością strukturalną podjednostek białkowych. Zatem nagromadzenie łagodnie szkodliwych mutacji w populacjach o małej wielkości indukuje wtórną selekcję dla interakcje białko-białko , które stabilizują kluczowe funkcje genów, łagodząc degradację strukturalną promowaną przez nieefektywną selekcję. W ten sposób złożone architektury białek i interakcje niezbędne do genezy różnorodności fenotypowej mogą początkowo pojawić się dzięki mechanizmom nieadaptacyjnym.
Krytyka, wyzwania i odpowiedzi
Kiemer i Cesareni wyrażają następujące obawy co do stanu pola (około 2007 r.), zwłaszcza w przypadku interaktomii porównawczej: Procedury eksperymentalne związane z polem są podatne na błędy, co prowadzi do „hałaśliwych wyników”. Prowadzi to do tego, że 30% wszystkich zgłoszonych interakcji to artefakty. W rzeczywistości dwie grupy stosujące te same techniki na tym samym organizmie stwierdziły mniej niż 30% wspólnych interakcji. Jednak niektórzy autorzy argumentowali, że taka niepowtarzalność wynika z niezwykłej wrażliwości różnych metod na małe zróżnicowanie eksperymentalne. Na przykład identyczne warunki w testach Y2H skutkują bardzo różnymi interakcjami, gdy stosuje się różne wektory Y2H.
Techniki mogą być stronnicze, tj. technika określa, które interakcje zostaną znalezione. W rzeczywistości każda metoda ma wbudowane uprzedzenia, zwłaszcza metody białkowe. Ponieważ każde białko jest inne, żadna metoda nie jest w stanie uchwycić właściwości każdego białka. Na przykład większość metod analitycznych, które dobrze sprawdzają się w przypadku białek rozpuszczalnych, słabo radzi sobie z białkami błonowymi. Dotyczy to również technologii Y2H i AP/MS.
Interactomy nie są prawie kompletne, być może z wyjątkiem S. cerevisiae. Nie jest to tak naprawdę krytyka, ponieważ każdy obszar naukowy jest początkowo „niekompletny”, dopóki metodologie nie zostaną ulepszone. Interactomics w 2015 roku to miejsce, w którym sekwencjonowanie genomu miało miejsce pod koniec lat 90., biorąc pod uwagę, że dostępnych jest tylko kilka zestawów danych dotyczących interakcji (patrz tabela powyżej).
Chociaż genomy są stabilne, interaktomy mogą się różnić w zależności od tkanek, typów komórek i etapów rozwoju. Ponownie, nie jest to krytyka, ale raczej opis wyzwań w tej dziedzinie.
Trudno jest dopasować ewolucyjnie spokrewnione białka u odlegle spokrewnionych gatunków. Podczas gdy homologiczne sekwencje DNA można stosunkowo łatwo znaleźć, znacznie trudniej jest przewidzieć interologie homologiczne („interologi”), ponieważ homologi dwóch oddziałujących białek nie muszą oddziaływać. Na przykład nawet w proteomie dwa białka mogą wchodzić w interakcje, ale ich paralogi nie.
Każdy interakcja białko-białko może reprezentować tylko częściową próbkę potencjalnych interakcji, nawet jeśli rzekomo ostateczna wersja zostanie opublikowana w czasopiśmie naukowym. Dodatkowe czynniki mogą odgrywać role w interakcjach białek, które nie zostały jeszcze włączone do interakcji. Siła wiązania różnych interaktorów białkowych, czynniki mikrośrodowiskowe, wrażliwość na różne procedury i stan fizjologiczny komórki wpływają na interakcje białko-białko, ale zwykle nie są uwzględniane w badaniach interakcji.
Zobacz też
- Bioinformatyka , Omika , Proteomika , Genomika
- Sieci biologiczne
- BioPlex
- konektom
- Słowniczek teorii grafów
- Interaktom ludzki
- Sieć interakcji
- Lista tematów omicznych w biologii
- Biologia matematyczna
- Sieć metaboliczna
- Modelowanie sieci metabolicznych
- Szlak metaboliczny
- Medycyna sieciowa
- Oddziaływania białko-białko
- Biologia systemów
- Chemoproteomika
Dalsza lektura
- Park J, Lappe M, Teichmann SA (marzec 2001). „Mapowanie interakcji rodziny białek: repertuary interakcji rodziny białek wewnątrzcząsteczkowych i międzycząsteczkowych w PDB i drożdżach”. J Mol Biol . 307 (3): 929–38. doi : 10.1006/jmbi.2001.4526 . PMID 11273711 .
Linki zewnętrzne
Serwery sieciowe Interactome
- Protinfo PPC przewiduje atomową strukturę 3D kompleksów białkowo-białkowych. Kittichotirat W, Guerquin M, Bumgarner R, Samudrala R (2009). „Protinfo PPC: serwer WWW do przewidywania kompleksów białkowych na poziomie atomowym” . Badania kwasów nukleinowych . 37 (problem z serwerem sieci Web): W519–W525. doi : 10.1093/nar/gkp306 . PMC 2703994 . PMID 19420059 .
- IBIS (serwer) raportuje, przewiduje i integruje wiele typów zachowanych interakcji dla białek.
Narzędzia do wizualizacji Interactome
- Wizualizacja danych opartych na sieci Web GPS-Prot dla interakcji białek
- PINV - Wizualizator sieci interakcji białek
Bazy danych Interactome
- Baza danych BioGRID
- mentha interaktywna przeglądarka Calderone; i in. (2013). „mentha: źródło do przeglądania zintegrowanych sieci interakcji białek” . Metody natury . 10 (8): 690–691. doi : 10.1038/nmeth.2561 . PMID 23900247 . S2CID 9733108 .
- IntAct: baza danych interakcji molekularnych
- Interactome.org — dedykowana witryna interaktywna.