faga T7

|
|
| Wirus Escherichia T7 | |
|---|---|
| Klasyfikacja wirusów | |
| (nierankingowe): | Wirus |
| królestwo : | Duplodnaviria |
| Królestwo: | Heunggongvirae |
| Gromada: | Urowiricota |
| Klasa: | Caudoviricetes |
| Zamówienie: | Caudovirales |
| Rodzina: | Autographiviridae |
| Rodzaj: | Wirus teseptima |
| Gatunek: |
Wirus Escherichia T7
|
Bakteriofag T7 (lub fag T7 ) to bakteriofag , wirus infekujący bakterie. Zakaża większość szczepów Escherichia coli i polega na rozmnażaniu się tych żywicieli. Bakteriofag T7 ma lityczny cykl życiowy , co oznacza, że niszczy zainfekowaną komórkę. Posiada również kilka właściwości, które czynią go idealnym fagiem do eksperymentów: jego oczyszczanie i koncentracja dały spójne wartości w analizach chemicznych; można go uczynić niezakaźnym przez wystawienie na działanie światła UV; i może być stosowany w prezentacji fagowej do klonowania Białka wiążące RNA .
Odkrycie
W badaniu przeprowadzonym w 1945 roku przez Demereca i Fano, T7 został użyty do opisania jednego z siedmiu typów fagów (T1 do T7), które rosną litycznie na Escherichia coli; chociaż wszystkie siedem fagów ponumerowano arbitralnie, później odkryto, że fagi o numerach nieparzystych lub fagi T-nieparzyste mają wspólne cechy morfologiczne i biochemiczne, które odróżniają je od fagów T-parzystych. Zanim fag został fizycznie nazwany T7, był używany we wcześniejszych eksperymentach. Niemiecko-amerykański biofizyk Max Delbrück pracował z tym samym wirusem pod koniec lat trzydziestych XX wieku, nazywając go fagiem δ, oraz francusko-kanadyjski mikrobiolog Félix d'Herelle prawdopodobnie badał swojego bliskiego krewnego w latach dwudziestych XX wieku.
Zastępy niebieskie
T7 rośnie na szorstkich szczepach Escherichia coli (tj. tych, które nie mają pełnej długości polisacharydu O-antygenu na swojej powierzchni) i niektórych innych bakteriach jelitowych , ale bliscy krewni również infekują gładkie, a nawet otoczkowe szczepy. E. coli jest bardziej oporna na T7 niż na inne podobne fagi. [ potrzebne źródło ]
Struktura wirionu
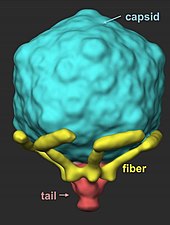
Wirus ma złożoną symetrię strukturalną, z kapsydem faga, który jest dwudziestościenny (dwadzieścia ścian) o wewnętrznej średnicy 55 nm i ogonem o średnicy 19 nm i długości 28,5 nm przymocowanym do kapsydu. Wyrzucanie białek z kapsydu podczas infekcji powoduje, że wirus zmienia strukturę, gdy wchodzi do komórki.
Genom
Genom faga T7 był jednym z pierwszych całkowicie zsekwencjonowanych genomów i został opublikowany w 1983 r. Głowa cząstki faga zawiera około 40 kbp genomu dsDNA , który koduje 55 białek . Genom zawiera liczne nakładające się geny , które częściowo usunięto poprzez „refaktoryzację” genomu w celu wytworzenia T7.1.
Koło życia
Cykl życiowy T7 wynosi 17 minut w temperaturze 37˚C, tj. czas od zakażenia do lizy komórki gospodarza, kiedy uwalniane są nowe fagi. Ze względu na krótki okres utajenia większość badań fizjologicznych przeprowadza się w temperaturze 30˚C, gdzie zakażone komórki ulegają lizie po 30 minutach. Jednak szczepy T7 o wysokiej sprawności zostały wyizolowane z okresem utajenia wynoszącym zaledwie ~11 minut w 37˚C rosnących w optymalnych warunkach w wynikach na bogatych pożywkach. Ten zaadaptowany fag może skutecznie powiększyć swoją populację o ponad 10 13 w ciągu jednej godziny wzrostu.
Infekcja bakterii gospodarza

Fag T7 rozpoznaje pewne receptory na powierzchni komórek E. coli i wiąże się z powierzchnią komórki za pomocą wirusowych włókien ogonka. W niektórych szczepach T7 włókna ogonka są zastąpione kolcami ogona, które rozkładają antygeny O lub K na powierzchni komórki poprzez aktywność enzymatyczną . [ potrzebne źródło ]
W procesie adsorpcji i penetracji lizozymy tworzą otwór w warstwie peptydoglikanu ściany komórkowej bakterii, umożliwiając przeniesienie wirusowego DNA do bakterii. Krótki, gruby ogon faga podobnego do T7 jest zbyt krótki, aby objąć otoczkę komórki, a aby wyrzucić genom faga do komórki na początku infekcji, białka wirionu muszą najpierw utworzyć kanał z czubka ogona do cytoplazmy komórki. Fag uwalnia również pięć białek potrzebnych do rozpoczęcia replikacji genomu wirusa i rozszczepienia genomu gospodarza. Bakteriofag T7 został wyewoluowany w celu obejścia kilku mechanizmów obronnych bakterii gospodarza, w tym ściany komórkowej peptydoglikanu i systemie CRISPR . Gdy fag T7 wprowadzi genom wirusa, proces replikacji DNA genomu gospodarza zostaje zatrzymany i rozpoczyna się replikacja genomu wirusa. [ potrzebne źródło ]
W optymalnych warunkach fag T7 może zakończyć proces lityczny w ciągu 25 minut, co prowadzi do śmierci komórki gospodarza E. coli . W czasie lizy wirus może wytworzyć ponad 100 potomstwa.
składniki
Gp5 (kodowany przez gen gp5 ) jest polimerazą DNA faga T7 . Polimeraza T7 wykorzystuje endogenną tioredoksynę E. coli , białko REDOX, jako ślizgowy zacisk DNA podczas replikacji DNA faga (chociaż zwykle tioredoksyna pełni inną funkcję). Zacisk przesuwny utrzymuje polimerazę na DNA, co zwiększa szybkość syntezy.
Replikacja i naprawa DNA
Fag T7 ma najprostszy znany replisom DNA , składający się z helikazy i prymazy , które znajdują się w pojedynczym łańcuchu polipeptydowym , który tworzy heksamer w obecności DNA i ATP lub dTTP . Polimeraza DNA T7 , wspomagana przez tioredoksynę E. coli , przeprowadza syntezę zarówno wiodącej, jak i opóźnionej nici DNA .
U faga T7 pęknięcia dwuniciowe DNA są prawdopodobnie naprawiane przez wstawienie fragmentu DNA dawcy w szczelinę w miejscu pęknięcia. Ta naprawa pęknięć dwuniciowych jest ułatwiona przez genu 2.5, które promuje hybrydyzację homologicznych komplementarnych nici DNA .
Replikacyjne półprodukty
Replikujący się wewnątrzkomórkowy DNA faga T7, po rozciągnięciu po lizie komórki, jest zwykle dłuższy niż chromosom dojrzałego faga (11 do 15 µM) i może występować w postaci wysoce połączonych liniowych nici do 66 razy dłuższych niż dojrzały fag chromosom. Replikujący się DNA można również zobaczyć w postaci zwiniętych struktur pierścieniowych, które wydają się odpowiadać wielokrotnie zapętlonym konfiguracjom DNA, w których superhelikalne skręty, niezbędne do zagęszczenia DNA, zostały złagodzone przez nacięcie nici podczas lizy komórki. [ potrzebne źródło ]
Zastosowania w biologii molekularnej
Sekwencja promotora T7 jest szeroko stosowana w biologii molekularnej ze względu na jej niezwykle wysokie powinowactwo do polimerazy RNA T7 , a tym samym wysoki poziom ekspresji.
T7 był używany jako model w biologii syntetycznej . Chan i in. (2005) „ zrefaktoryzowali ” genom T7, zastępując około 12 kbp jego genomu zmodyfikowanym DNA. Zmodyfikowany DNA został zaprojektowany tak, aby był łatwiejszy w obróbce na wiele sposobów: poszczególne elementy funkcjonalne zostały oddzielone miejscami endonukleazy restrykcyjnej w celu prostej modyfikacji, a nakładające się domeny kodujące białka zostały rozdzielone i, w razie potrzeby, zmodyfikowane przez ciche mutacje pojedynczej pary zasad . T7 został przetestowany na ludzkim kostniakomięsaku do leczenia komórek nowotworowych. [ potrzebne źródło ]
Linki zewnętrzne
- T7 + Phage w US National Library of Medicine Medical Subject Headings (MeSH)
- [1] Strefa Wirusowa
- Nowe Szczegóły dotyczące interakcji T7-host . Magazyn mikrobów
- Proteom referencyjny T7 w Uniprot


![Schematic view of the phage T7 genome. Boxes are genes, numbers are gene numbers. Colors indicate functional groups as shown. White boxes are genes of unknown function or without annotation. Modified after [16]](http://upload.wikimedia.org/wikipedia/commons/thumb/d/db/T7_phage_genome.png/500px-T7_phage_genome.png)


