Wyświetlacz faga
Prezentacja fagowa to technika laboratoryjna służąca do badania interakcji białko-białko , białko - peptyd i białko- DNA , która wykorzystuje bakteriofagi ( wirusy infekujące bakterie ) do łączenia białek z kodującą je informacją genetyczną . W tej technice gen kodujący białko będące przedmiotem zainteresowania jest wstawiany do białka otoczki faga gen, powodując, że fag „pokazuje” białko na zewnątrz, jednocześnie zawierając gen białka w środku, co skutkuje połączeniem między genotypem a fenotypem . Te prezentujące fagi można następnie przeszukiwać pod kątem innych białek, peptydów lub sekwencji DNA, w celu wykrycia interakcji między prezentowanym białkiem a tymi innymi cząsteczkami. W ten sposób duże biblioteki białek można przeszukiwać i amplifikować w procesie zwanym selekcją in vitro , który jest analogiczny do selekcji naturalnej .
Najczęstszymi bakteriofagami stosowanymi w prezentacji fagowej są fagi nitkowate M13 i fd , chociaż stosowano również fagi T4 , T7 i λ .
Historia
Prezentacja fagowa została po raz pierwszy opisana przez George'a P. Smitha w 1985 r., Kiedy zademonstrował prezentację peptydów na fagu nitkowatym (długie, cienkie wirusy infekujące bakterie) poprzez fuzję białka kapsydu wirusa z jednym peptydem ze zbioru sekwencji peptydowych. To pokazało różne peptydy na zewnętrznych powierzchniach kolekcji klonów wirusowych, gdzie etap przesiewania procesu wyizolował peptydy o najwyższym powinowactwie wiązania. W 1988 roku Stephen Parmley i George Smith opisali biopanning dla selekcji powinowactwa i wykazał, że rekurencyjne rundy selekcji mogą wzbogacić klony obecne na poziomie 1 na miliard lub mniej. W 1990 roku Jamie Scott i George Smith opisali tworzenie dużych losowych bibliotek peptydów prezentowanych na fagu nitkowatym. Technologia prezentacji fagowej była dalej rozwijana i ulepszana przez grupy z Laboratorium Biologii Molekularnej z Gregiem Winterem i Johnem McCaffertym , The Scripps Research Institute z Richardem Lernerem i Carlosem Barbasem oraz Niemieckie Centrum Badań nad Rakiem z Frankiem Breitlingiem i Stefanem Dübelem do prezentacji białek, takich jak przeciwciała, do inżynierii białek terapeutycznych . Smith i Winter otrzymali połowę nagrody Nobla 2018 w dziedzinie chemii za ich wkład w rozwój prezentacji fagowej. Patent George'a Pieczenika, który twierdzi, że ma pierwszeństwo z 1985 r., Opisuje również generowanie bibliotek peptydów.
Zasada
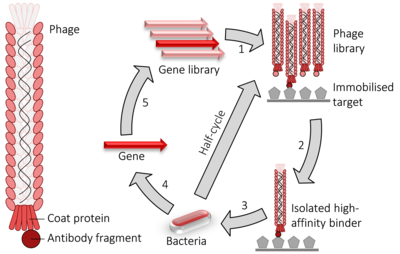
Podobnie jak system dwuhybrydowy , prezentacja na fagach jest wykorzystywana do wysokowydajnych badań przesiewowych interakcji białek. W przypadku na fagu nitkowatym M13 , DNA kodujący białko lub peptyd będący przedmiotem zainteresowania jest ligowany z genem pIII lub pVIII, kodującym odpowiednio mniejsze lub większe białko otoczki . Czasami stosuje się wiele miejsc klonowania, aby upewnić się, że fragmenty zostaną wstawione we wszystkich trzech możliwych ramkach odczytu , tak aby fragment cDNA został przetłumaczony we właściwej oprawie. Hybryda genu faga i wstawki DNA jest następnie wstawiana (proces znany jako „ transdukcja ”) do komórek bakteryjnych E. coli , takich jak TG1, SS320, ER2738 lub XL1-Blue E. coli . Jeśli wektor „ fagemidowy ” (uproszczony wektor konstrukcyjny do prezentacji), cząsteczki faga nie zostaną uwolnione z komórek E. coli , dopóki nie zostaną zainfekowane fagiem pomocniczym , co umożliwia upakowanie DNA faga i złożenie dojrzałych wirionów z odpowiednim fragmentem białka jako częścią ich zewnętrznej otoczki na drugorzędnym (pIII) lub głównym (pVIII) białku otoczki. Poprzez unieruchomienie odpowiedniego DNA lub docelowego białka na powierzchni studzienki płytki do mikromiareczkowania , fag, który prezentuje białko, które wiąże się z jednym z tych celów na swojej powierzchni, pozostanie, podczas gdy inne zostaną usunięte przez wypłukanie. Te, które pozostają, mogą być eluowane , użyte do wytworzenia większej ilości faga (poprzez infekcję bakteryjną fagiem pomocniczym) i do wytworzenia mieszaniny fagowej, która jest wzbogacona odpowiednim (tj. wiążącym) fagiem. Powtarzające się powtarzanie tych etapów jest określane jako „płukanie” w odniesieniu do wzbogacania próbki złota poprzez usuwanie niepożądanych materiałów. Faga eluowanego w ostatnim etapie można użyć do zakażenia odpowiedniego gospodarza bakteryjnego, z którego można zebrać fagemidy oraz wyciąć i zsekwencjonować odpowiednią sekwencję DNA w celu zidentyfikowania odpowiednich, oddziałujących białek lub fragmentów białek. [ potrzebne źródło ]
Wykorzystanie faga pomocniczego można wyeliminować, stosując technologię „bakteryjnej linii komórkowej do pakowania”.
Elucję można przeprowadzić łącząc bufor elucyjny o niskim pH z sonifikacją, która oprócz rozluźnienia interakcji peptyd-cel służy również do oderwania cząsteczki docelowej od powierzchni immobilizacji. Ta na ultradźwiękach umożliwia jednoetapową selekcję peptydu o wysokim powinowactwie.
Aplikacje
Zastosowania technologii prezentacji fagowej obejmują określenie partnerów interakcji białka (które można by wykorzystać jako „przynętę” immobilizowanego faga z biblioteką DNA składającą się ze wszystkich sekwencji kodujących komórkę, tkankę lub organizm), tak aby funkcja lub mechanizm można określić funkcję tego białka. Prezentacja na fagach jest również szeroko stosowaną metodą in vitro (zwaną także inżynierią białek ). Jako taka, prezentacja na fagach jest użytecznym narzędziem w odkrywaniu leków . Służy do znajdowania nowych ligandów (inhibitory enzymów, agoniści i antagoniści receptorów) do białek docelowych. Technika ta jest również wykorzystywana do oznaczania antygenów nowotworowych (do wykorzystania w diagnostyce i ukierunkowaniu terapeutycznym) oraz do poszukiwania interakcji białko-DNA przy użyciu specjalnie skonstruowanych bibliotek DNA z losowymi segmentami. Ostatnio ekspozycję na fagach zastosowano również w kontekście leczenia raka – na przykład metodą adopcyjnego transferu komórek. W takich przypadkach prezentacja na fagach jest wykorzystywana do tworzenia i selekcji syntetycznych przeciwciał ukierunkowanych na białka powierzchniowe guza. Są one przekształcane w syntetyczne receptory dla limfocytów T pobranych od pacjenta, które są wykorzystywane do zwalczania choroby.
Konkurencyjne metody ewolucji białek in vitro obejmują prezentację drożdżową , prezentację bakteryjną , prezentację rybosomalną i prezentację mRNA . [ potrzebne źródło ]
Dojrzewanie przeciwciał in vitro
Wynalazek prezentacji przeciwciał na fagach zrewolucjonizował odkrycie leków opartych na przeciwciałach. Wstępne prace wykonały laboratoria Laboratorium Biologii Molekularnej MRC ( Greg Winter i John McCafferty ), Instytutu Badawczego Scripps (Richard Lerner i Carlos F. Barbas) oraz Niemieckiego Centrum Badań nad Rakiem (Frank Breitling i Stefan Dübel). W 1991 roku grupa Scripps zgłosiła pierwszą prezentację i selekcję ludzkich przeciwciał na fagu. W tym wstępnym badaniu opisano szybką izolację ludzkiego przeciwciała Fab , które się wiąże toksynę tężcową , a następnie metodę rozszerzono na szybkie klonowanie ludzkich przeciwciał anty-HIV-1 do projektowania szczepionek i terapii.
Prezentacja bibliotek przeciwciał na fagach stała się potężną metodą zarówno badania odpowiedzi immunologicznej, jak i metodą szybkiej selekcji i ewolucji ludzkich przeciwciał do terapii. Prezentacja przeciwciał na fagach została później wykorzystana przez Carlosa F. Barbasa z The Scripps Research Institute do stworzenia syntetycznych bibliotek ludzkich przeciwciał, zasady opatentowanej po raz pierwszy w 1990 roku przez Breitlinga i współpracowników (Patent CA 2035384), umożliwiając w ten sposób tworzenie ludzkich przeciwciał in vitro z syntetycznych elementy różnorodności.
Biblioteki przeciwciał prezentujące miliony różnych przeciwciał na fagu są często wykorzystywane w przemyśle farmaceutycznym do izolowania wysoce specyficznych przeciwciał terapeutycznych w celu opracowania leków opartych na przeciwciałach, głównie jako leków przeciwnowotworowych lub przeciwzapalnych. Jednym z najbardziej udanych był adalimumab , odkryty przez Cambridge Antibody Technology jako D2E7 oraz opracowany i wprowadzony na rynek przez Abbott Laboratories . Adalimumab, przeciwciało przeciwko TNF alfa , było pierwszym na świecie w pełni ludzkim przeciwciałem, którego roczna sprzedaż przekroczyła 1 miliard dolarów.
Protokół ogólny
Poniżej znajduje się sekwencja zdarzeń, które są śledzone w badaniach przesiewowych na fagach w celu zidentyfikowania polipeptydów, które wiążą się z wysokim powinowactwem z pożądanym białkiem docelowym lub sekwencją DNA: [ potrzebne źródło ]
- Docelowe białka lub sekwencje DNA są immobilizowane w studzienkach płytki do mikromiareczkowania .
- Wiele sekwencji genetycznych ulega ekspresji w bibliotece bakteriofagów w postaci fuzji z białkiem otoczki bakteriofaga, dzięki czemu są one prezentowane na powierzchni cząstki wirusowej. Wyświetlane białko odpowiada sekwencji genetycznej faga.
- Ta biblioteka prezentacji fagowej jest dodawana do szalki i po pozostawieniu faga do związania się, szalka jest myta.
- Białka eksponujące fagi, które oddziałują z cząsteczkami docelowymi, pozostają przyczepione do szalki, podczas gdy wszystkie inne są wypłukiwane.
- Przyłączony fag może być eluowany i użyty do wytworzenia większej liczby fagów przez infekcję odpowiednich gospodarzy bakteryjnych. Nowy fag stanowi wzbogaconą mieszaninę, zawierającą znacznie mniej faga nieistotnego (tj. niewiążącego) niż był obecny w początkowej mieszaninie.
- Etapy od 3 do 5 są ewentualnie powtarzane jeden lub więcej razy, dodatkowo wzbogacając bibliotekę fagową w białka wiążące.
- Po dalszej amplifikacji opartej na bakteriach, DNA w oddziałującym fagu jest sekwencjonowane w celu zidentyfikowania oddziałujących białek lub fragmentów białek.
Selekcja białka płaszcza
Fagi nitkowate
pIII
pIII jest białkiem, które określa zakaźność wirionu. pIII składa się z trzech domen (N1, N2 i CT) połączonych łącznikami bogatymi w glicynę. Domena N2 wiąże się z pilusem F podczas infekcji wirionem, uwalniając domenę N1, która następnie oddziałuje z białkiem TolA na powierzchni bakterii. Insercje w obrębie tego białka są zwykle dodawane w pozycji 249 (w obrębie regionu łącznika między CT a N2), w pozycji 198 (w domenie N2) i na N- końcu (wstawiony między N-końcową sekwencją sekrecyjną a N-końcem pIII). Jednakże, stosując miejsce BamHI znajdujące się w pozycji 198, należy uważać na niesparowaną resztę cysteiny (C201), która może powodować problemy podczas prezentacji fagowej, jeśli stosuje się nieobciętą wersję pIII.
Zaletą stosowania pIII zamiast pVIII jest to, że pIII umożliwia prezentację monowalentną, gdy stosuje się fagemid (plazmid pochodzący z fagów Ff ) w połączeniu z fagiem pomocniczym. Ponadto pIII umożliwia wstawienie większych sekwencji białkowych (>100 aminokwasów) i jest na nie bardziej tolerancyjny niż pVIII. Jednak użycie pIII jako partnera fuzyjnego może prowadzić do zmniejszenia zakaźności faga, co prowadzi do problemów, takich jak błąd selekcji spowodowany różnicą w szybkości wzrostu faga lub, co gorsza, niezdolność faga do zakażenia swojego gospodarza. Utraty infekcyjności faga można uniknąć stosując plazmid fagemidowy i faga pomocniczego tak, że powstały fag zawiera zarówno pIII typu dzikiego, jak i fuzję.
cDNA analizowano również przy użyciu pIII poprzez system dwóch komplementarnych suwaków leucynowych, Direct Interaction Rescue lub przez dodanie 8-10-aminokwasowego łącznika pomiędzy cDNA i pIII na C-końcu.
pVIII
pVIII jest głównym białkiem otoczki fagów Ff. Peptydy są zwykle połączone z N-końcem pVIII. Zwykle peptydy, które można połączyć z pVIII, mają długość 6-8 aminokwasów. Wydaje się, że ograniczenie rozmiaru ma mniej wspólnego z przeszkodą strukturalną spowodowaną przez dodaną sekcję, a bardziej z wykluczeniem rozmiaru spowodowanym przez pIV podczas eksportu białka płaszcza. Ponieważ na typowym fagu znajduje się około 2700 kopii białka, bardziej prawdopodobne jest, że białko będące przedmiotem zainteresowania będzie wyrażane poliwalentnie, nawet jeśli używany jest fagemid. To sprawia, że użycie tego białka jest niekorzystne dla odkrycia partnerów wiążących o wysokim powinowactwie.
Aby przezwyciężyć problem wielkości pVIII, zaprojektowano sztuczne białka płaszcza. Przykładem jest odwrócone białko sztucznego płaszcza (ACP) Weissa i Sidhu, które umożliwia prezentację dużych białek na C-końcu. ACP mogą jednak prezentować białko o masie 20 kDa tylko na niskich poziomach (przeważnie tylko jednowartościowe).
pVI
pVI jest szeroko stosowany do prezentacji bibliotek cDNA. Prezentacja bibliotek cDNA poprzez prezentację na fagach jest atrakcyjną alternatywą dla metody drożdży-2-hybrydowej do odkrywania oddziałujących białek i peptydów ze względu na jej wysoką przepustowość. PVI stosowano preferencyjnie w stosunku do pVIII i pIII do ekspresji bibliotek cDNA, ponieważ można dodać białko będące przedmiotem zainteresowania do C-końca pVI bez znacznego wpływu na rolę pVI w składaniu faga. Oznacza to, że kodon stop w cDNA nie stanowi już problemu. Jednak prezentacja cDNA na fagach jest zawsze ograniczona przez niezdolność większości prokariotów do wytwarzania modyfikacji potranslacyjnych obecnych w komórkach eukariotycznych lub przez nieprawidłowe fałdowanie białek wielodomenowych.
Podczas gdy pVI był użyteczny do analizy bibliotek cDNA, pIII i pVIII pozostają najczęściej wykorzystywanymi białkami płaszcza do prezentacji fagowej.
pVII i pIX
W eksperymencie przeprowadzonym w 1995 roku próbowano wyświetlić S-transferazę glutationową zarówno na pVII, jak i pIX, ale nie powiodło się. Jednak prezentacja tego białka na fagach została pomyślnie zakończona po dodaniu peryplazmatycznej sekwencji sygnałowej (pelB lub ompA) na N-końcu. W niedawnym badaniu wykazano, że AviTag, FLAG i His mogą być wyświetlane na pVII bez potrzeby stosowania sekwencji sygnałowej. Następnie wyrażano ekspresję jednołańcuchowych Fv (scFv) i jednołańcuchowych receptorów komórek T (scTCR) zarówno z sekwencją sygnałową, jak i bez niej.
PelB (aminokwasowa sekwencja sygnałowa, która kieruje białko do peryplazmy, gdzie peptydaza sygnałowa następnie odcina PelB) poprawiła poziom prezentacji fagowej w porównaniu z fuzjami pVII i pIX bez sekwencji sygnałowej. Doprowadziło to jednak do włączenia większej liczby genomów fagów pomocniczych zamiast genomów fagemidów. We wszystkich przypadkach poziomy prezentacji fagowej były niższe niż przy użyciu fuzji pIII. Jednak niższy wskaźnik może być korzystniejszy dla doboru spoiw, ponieważ niższy wskaźnik jest bliższy rzeczywistemu wyświetlaczowi jednowartościowemu. W pięciu z sześciu przypadków fuzje pVII i pIX bez pelB były bardziej wydajne niż fuzje pIII w testach selekcji powinowactwa. Artykuł stwierdza nawet, że platformy wyświetlaczy pVII i pIX mogą w dłuższej perspektywie przewyższyć pIII.
Zastosowanie pVII i pIX zamiast pIII może być również korzystne, ponieważ odzyskiwanie wirionów można przeprowadzić bez zrywania wiązania wirion-antygen, jeśli zastosowany pIII jest typu dzikiego. Zamiast tego można rozszczepić odcinek między kulką a antygenem w celu elucji. Ponieważ pIII jest nienaruszony, nie ma znaczenia, czy antygen pozostaje związany z fagiem.
fagi T7
Kwestia wykorzystania fagów Ff do prezentacji fagowej polega na tym, że wymagają one translokacji białka będącego przedmiotem zainteresowania przez wewnętrzną błonę bakteryjną, zanim zostaną złożone w faga. Niektóre białka nie mogą przejść tego procesu i dlatego nie mogą być prezentowane na powierzchni fagów Ff. W takich przypadkach zamiast tego stosuje się prezentację fagową T7. W prezentacji fagowej T7 białko, które ma być prezentowane, jest przyłączone do C-końca białka kapsydu genu 10 T7.
Wadą stosowania T7 jest to, że rozmiar białka, które może być eksprymowane na powierzchni, jest ograniczony do krótszych peptydów, ponieważ duże zmiany w genomie T7 nie mogą być uwzględnione, jak ma to miejsce w przypadku M13, gdzie fag po prostu wydłuża swój płaszcz, aby dopasować się do większego genomu w nim. Jednak może być użyteczna do wytwarzania dużej biblioteki białek do selekcji scFV, gdzie scFV ulega ekspresji na fagu M13, a antygeny ulegają ekspresji na powierzchni faga T7.
Zasoby i narzędzia bioinformatyczne
Bazy danych i narzędzia obliczeniowe dla mimotopów były ważną częścią badań nad prezentacją fagową. Bazy danych, programy i serwery internetowe są szeroko stosowane do wykluczania peptydów niezwiązanych z celem, charakteryzowania interakcji między małymi cząsteczkami a białkami oraz mapowania interakcji między białkami. Użytkownicy mogą wykorzystać trójwymiarową strukturę białka i peptydów wybranych z eksperymentu z prezentacją fagową do mapowania epitopów konformacyjnych. Niektóre z szybkich i wydajnych metod obliczeniowych są dostępne online.
Zobacz też
Konkurujące techniki:
Dalsza lektura
- Ledsgaard L, Kilstrup M, Karatt-Vellatt A, McCafferty J, Laustsen AH (2018). „Podstawy technologii prezentacji przeciwciał na fagach” (PDF) . Toksyny . 10 (6): 236. doi : 10.3390/toxins10060236 . PMC 6024766 . PMID 29890762 .
- Wybór a projektowanie w inżynierii chemicznej
- Biblioteka fagów ludzkich przeciwciał ETH-2
- Sidhu SS, Lowman HB, Cunningham BC, Wells JA (2000). „Wystawa na fagach do selekcji nowych peptydów wiążących”. Met. Enzymol . Metody w enzymologii. 328 : 333–63. doi : 10.1016/S0076-6879(00)28406-1 . ISBN 9780121822293 . PMID 11075354 .
Linki zewnętrzne
|
Zasoby biblioteczne dotyczące prezentacji Phage |