Spektrometria mas białek
Spektrometria masowa białek odnosi się do zastosowania spektrometrii masowej do badania białek . Spektrometria mas jest ważną metodą dokładnego określania masy i charakteryzowania białek, a do jej wielu zastosowań opracowano różnorodne metody i instrumenty. Jej zastosowania obejmują identyfikację białek i ich modyfikacje potranslacyjne , wyjaśnianie kompleksów białkowych, ich podjednostek i oddziaływań funkcjonalnych, a także globalny pomiar białek w proteomice . Można ją również wykorzystać do lokalizacji białek w różnych organellach i określenia interakcji między różnymi białkami, a także z lipidami błonowymi.
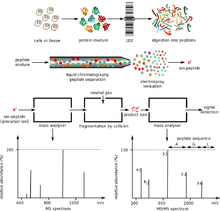
Dwie podstawowe metody stosowane do jonizacji białek w spektrometrii mas to jonizacja przez elektrorozpylanie (ESI) i desorpcja/jonizacja laserowa wspomagana matrycą (MALDI). Te techniki jonizacji są stosowane w połączeniu z analizatorami mas, takimi jak tandemowa spektrometria mas . Ogólnie rzecz biorąc, białka analizuje się albo metodą „ od góry do dołu ”, w której białka analizuje się w stanie nienaruszonym, albo metodą „ od dołu do góry ”. „podejście, w którym białka są najpierw trawione na fragmenty. Czasami można zastosować pośrednie podejście „od środka”, w którym analizowane są większe fragmenty peptydów.
Historia
Zastosowanie spektrometrii mas do badania białek spopularyzowano w latach 80. XX wieku po opracowaniu metod MALDI i ESI . Te techniki jonizacji odegrały znaczącą rolę w charakteryzowaniu białek. (MALDI) Jonizacja z desorpcją laserową wspomaganą matrycą została wymyślona pod koniec lat 80-tych przez Franza Hillenkampa i Michaela Karasa . Hillenkamp, Karas i ich współpracownicy byli w stanie zjonizować aminokwas alaninę , mieszając ją z aminokwasem tryptofanem i naświetlano laserem impulsowym o długości fali 266 nm. Choć przełom był ważny, przełom nastąpił dopiero w 1987 r. W 1987 r. Koichi Tanaka zastosował „metodę ultradrobnego metalu i ciekłej matrycy” i zjonizowane biomolekuły o wielkości 34 472 Da białkowej karboksypeptydazy-A.
W 1968 roku Malcolm Dole opisał pierwsze zastosowanie jonizacji przez elektrorozpylanie za pomocą spektrometrii mas. Mniej więcej w tym samym czasie, gdy spopularyzowano MALDI, cytowano Johna Bennetta Fenna za rozwój jonizacji przez elektrorozpylanie. Koichi Tanaka wraz z Johnem Fennem i Kurtem Wüthrichem otrzymał Nagrodę Nobla w dziedzinie chemii w 2002 roku „za opracowanie metod identyfikacji i analizy struktury makrocząsteczek biologicznych”. Te metody jonizacji znacznie ułatwiły badanie białek metodą spektrometrii mas. W rezultacie spektrometria mas białek odgrywa obecnie wiodącą rolę w charakteryzowaniu białek.
Metody i podejścia
Techniki
Spektrometria mas białek wymaga, aby białka w roztworze lub w stanie stałym zostały przekształcone w postać zjonizowaną w fazie gazowej, zanim zostaną wstrzyknięte i przyspieszone w polu elektrycznym lub magnetycznym w celu analizy. Dwie podstawowe metody jonizacji białek to jonizacja przez elektrorozpylanie (ESI) i desorpcja/jonizacja laserowa wspomagana matrycą (MALDI). W elektrorozpylaniu jony powstają z białek w roztworze, co pozwala na jonizację delikatnych cząsteczek w stanie nienaruszonym, czasami zachowując interakcje niekowalencyjne. W MALDI białka są osadzone w matrycy, zwykle w postaci stałej, a jony powstają w wyniku impulsów światła laserowego. Electrospray wytwarza więcej wielokrotnie naładowanych jonów niż MALDI, umożliwiając pomiar białek o dużej masie i lepszą fragmentację w celu identyfikacji, podczas gdy MALDI jest szybki i mniej podatny na wpływ zanieczyszczeń, buforów i dodatków.
Analizę masy całego białka przeprowadza się głównie przy użyciu MS czasu przelotu (TOF) lub rezonansu cyklotronowego z transformacją Fouriera (FT-ICR). Preferowane są tu te dwa typy przyrządów ze względu na szeroki zakres mas, a w przypadku FT-ICR dużą dokładność masową. Jonizacja białka przez elektrorozpylanie często skutkuje powstaniem wielu naładowanych cząstek o masie 800 < m/z <2000, a powstałe widmo można rozłożyć w celu określenia średniej masy białka z dokładnością do 50 ppm lub lepszą przy użyciu przyrządów TOF lub pułapek jonowych .
Analiza masowa peptydów proteolitycznych jest popularną metodą charakteryzowania białek, ponieważ do charakteryzacji można zastosować tańsze konstrukcje instrumentów. Dodatkowo przygotowanie próbki jest łatwiejsze, gdy całe białka zostaną strawione na mniejsze fragmenty peptydowe. Najpowszechniej stosowanym instrumentem do analizy masy peptydów są instrumenty MALDI-TOF, ponieważ pozwalają one na uzyskanie odcisków palców masy peptydów (PMF) w szybkim tempie (1 PMF można analizować w około 10 sekund). Wielostopniowy kwadrupolowy czas przelotu i kwadrupolowa pułapka jonowa również znajdują zastosowanie w tym zastosowaniu.
Tandemowa spektrometria mas (MS/MS) służy do pomiaru widm fragmentacji i identyfikacji białek z dużą szybkością i dokładnością. Dysocjacja wywołana kolizją jest wykorzystywana w głównych zastosowaniach do generowania zestawu fragmentów z określonego jonu peptydowego. W procesie fragmentacji powstają przede wszystkim produkty rozszczepienia, które rozrywają wiązania peptydowe. Ze względu na prostotę fragmentacji możliwe jest wykorzystanie obserwowanych mas fragmentów w celu dopasowania do bazy danych przewidywanych mas dla jednej z wielu podanych sekwencji peptydowych. Niedawno zbadano tandemowy MS jonów całych białek za pomocą dysocjacji wychwytu elektronów i wykazał w zasadzie obszerne informacje o sekwencji, ale nie jest to w powszechnej praktyce.
Podchodzi do
Zgodnie z wydajnością i zakresem mas dostępnych spektrometrów mas, do charakteryzowania białek stosuje się dwa podejścia. W pierwszym przypadku nienaruszone białka są jonizowane jedną z dwóch technik opisanych powyżej, a następnie wprowadzane do analizatora masy. Podejście to określa się jako strategię analizy białek „ od góry do dołu ”, ponieważ polega na rozpoczęciu od całej masy, a następnie jej rozłożeniu. Podejście odgórne ogranicza się jednak głównie do niskoprzepustowych badań pojedynczych białek ze względu na problemy związane z obsługą całych białek, ich heterogenicznością i złożonością ich analiz.
W drugim podejściu, określanym jako MS „ od dołu do góry ”, białka są trawione enzymatycznie na mniejsze peptydy przy użyciu proteazy , takiej jak trypsyna . Następnie peptydy te wprowadza się do spektrometru mas i identyfikuje za pomocą peptydowego odcisku palca lub tandemowej spektrometrii mas . Dlatego też podejście to wykorzystuje identyfikację na poziomie peptydu, aby wnioskować o istnieniu białek złożonych od nowa ponowne wykrywanie. Mniejsze i bardziej jednolite fragmenty są łatwiejsze do analizy niż nienaruszone białka i można je również określić z dużą dokładnością, dlatego to podejście „oddolne” jest preferowaną metodą badań w proteomice. Kolejnym podejściem, które zaczyna być przydatne, jest podejście pośrednie „od środka”, w którym analizuje się peptydy proteolityczne większe niż typowe peptydy trypsynowe.
Frakcjonowanie białek i peptydów
Białka będące przedmiotem zainteresowania są zwykle częścią złożonej mieszaniny wielu białek i cząsteczek, które współistnieją w środowisku biologicznym. Stwarza to dwa istotne problemy. Po pierwsze, dwie techniki jonizacji stosowane w przypadku dużych cząsteczek działają dobrze tylko wtedy, gdy mieszanina zawiera mniej więcej równe ilości materiału, podczas gdy w próbkach biologicznych różne białka są zwykle obecne w znacznie różniących się ilościach. Jeśli taką mieszaninę jonizuje się za pomocą elektrorozpylania lub MALDI , gatunki liczniejsze mają tendencję do „topienia” lub tłumienia sygnałów pochodzących od gatunków mniej licznych. Po drugie, widmo masowe złożonej mieszaniny jest bardzo trudne do interpretacji ze względu na przytłaczającą liczbę składników mieszaniny. Sytuację pogarsza fakt, że enzymatyczne trawienie białka powoduje powstanie dużej liczby produktów peptydowych.
W świetle tych problemów do rozdzielania białek powszechnie stosuje się metody jedno- i dwuwymiarowej elektroforezy żelowej oraz wysokosprawnej chromatografii cieczowej. Pierwsza metoda frakcjonuje całe białka poprzez dwuwymiarową elektroforezę żelową . Pierwszym wymiarem żelu 2D jest ogniskowanie izoelektryczne (IEF). W tym wymiarze białko rozdzielane jest na podstawie jego punktu izoelektrycznego (pI), a drugim wymiarem jest elektroforeza w żelu poliakryloamidowym SDS (SDS-PAGE). Wymiar ten oddziela białko według jego masy cząsteczkowej. Po zakończeniu tego etapu następuje trawienie w żelu. W niektórych sytuacjach może być konieczne połączenie obu tych technik. Plamy żelowe zidentyfikowane na żelu 2D są zwykle przypisane jednemu białku. Jeśli pożądana jest tożsamość białka, zwykle stosuje się metodę trawienia w żelu stosuje się, podczas którego wycina się żądaną plamkę białkową i trawi proteolitycznie. Masy peptydów powstałe w wyniku trawienia można określić metodą spektrometrii mas z wykorzystaniem peptydowego odcisku palca . Jeżeli informacja ta nie pozwala na jednoznaczną identyfikację białka, jego peptydy można poddać tandemowej spektrometrii mas w celu sekwencjonowania de novo . Niewielkie zmiany masy i ładunku można wykryć za pomocą 2D-PAGE. Wadą tej techniki jest mały zakres dynamiczny w porównaniu z innymi metodami, niektóre białka są nadal trudne do rozdzielenia ze względu na ich kwasowość, zasadowość, hydrofobowość i rozmiar (zbyt duży lub zbyt mały).
Druga metoda, wysokosprawna chromatografia cieczowa, służy do frakcjonowania peptydów po trawieniu enzymatycznym. Charakterystyka mieszanin białek za pomocą HPLC/MS nazywana jest również proteomią shotgun i MuDPIT (technologia wielowymiarowej identyfikacji białek). Mieszaninę peptydów powstałą w wyniku trawienia mieszaniny białek frakcjonuje się w jednym lub dwóch etapach chromatografii cieczowej. Eluent z etapu chromatografii można albo bezpośrednio wprowadzić do spektrometru mas poprzez jonizację przez elektrorozpylanie, albo rozprowadzić na szeregu małych punktów w celu późniejszej analizy mas przy użyciu MALDI.
Aplikacje
Identyfikacja białek
Istnieją dwa główne sposoby wykorzystania MS do identyfikacji białek. Odcisk palca masy peptydów wykorzystuje masy peptydów proteolitycznych jako dane wejściowe do przeszukiwania bazy danych przewidywanych mas, które powstałyby w wyniku trawienia listy znanych białek. Jeżeli sekwencja białka na liście referencyjnej powoduje powstanie znacznej liczby przewidywanych mas odpowiadających wartościom doświadczalnym, istnieją pewne dowody na to, że białko to było obecne w pierwotnej próbce. Etapy oczyszczania ograniczają zatem wydajność metody pobierania odcisków palców masy peptydów. Alternatywnie, peptydy można fragmentować za pomocą MS/MS, aby je dokładniej zidentyfikować.
MS jest także preferowaną metodą identyfikacji modyfikacji potranslacyjnych w białkach w porównaniu z innymi podejściami, takimi jak metody oparte na przeciwciałach.
de novo (peptydów).
de novo do spektrometrii mas zazwyczaj przeprowadza się bez wcześniejszej wiedzy o sekwencji aminokwasów. Jest to proces przypisywania aminokwasów z mas fragmentów peptydowych białka . Sekwencjonowanie de novo okazało się skuteczne w potwierdzaniu i rozszerzaniu wyników przeszukiwania baz danych.
Ponieważ sekwencjonowanie de novo opiera się na masie, a niektóre aminokwasy mają identyczne masy (np. leucyna i izoleucyna ), dokładne ręczne sekwencjonowanie może być trudne. Dlatego może okazać się konieczne wykorzystanie aplikacji do wyszukiwania homologii sekwencji do jednoczesnej pracy pomiędzy przeszukiwaniem bazy danych a od nowa , aby zaradzić temu nieodłącznemu ograniczeniu.
Przeszukiwanie bazy danych ma tę zaletę, że szybko identyfikuje sekwencje, pod warunkiem, że zostały już udokumentowane w bazie danych. Inne nieodłączne ograniczenia przeszukiwania baz danych obejmują modyfikacje/mutacje sekwencji (niektóre przeszukiwania baz danych nie uwzględniają odpowiednio zmian w „udokumentowanej” sekwencji, w związku z czym mogą przeoczyć cenne informacje), nieznane (jeśli sekwencja nie jest udokumentowana, nie zostanie znaleziona ), fałszywe alarmy oraz niekompletne i uszkodzone dane.
Bibliotekę widm peptydów z adnotacjami można również zastosować jako odniesienie do identyfikacji białka/peptydu. Oferuje wyjątkową siłę w postaci ograniczonej przestrzeni wyszukiwania i zwiększonej specyficzności. Ograniczenia obejmują, że widma nieuwzględnione w bibliotece nie zostaną zidentyfikowane, widma zebrane z różnych typów spektrometrów mas mogą mieć dość odmienne cechy, a widma referencyjne w bibliotece mogą zawierać piki szumu, co może prowadzić do fałszywie pozytywnych identyfikacji. Opisano wiele różnych podejść algorytmicznych do identyfikacji peptydów i białek na podstawie tandemowej spektrometrii mas (MS/MS), de novo i wyszukiwanie w oparciu o znaczniki sekwencji.
Prezentacja antygenu
Prezentacja antygenu jest pierwszym krokiem w edukacji układu odpornościowego w zakresie rozpoznawania nowych patogenów. W tym celu komórki prezentujące antygen udostępniają układowi odpornościowemu fragmenty białek poprzez cząsteczki MHC . Jednak nie wszystkie fragmenty białek wiążą się z cząsteczkami MHC konkretnego osobnika. Za pomocą spektrometrii mas można określić prawdziwe spektrum cząsteczek prezentowanych układowi odpornościowemu.
Ilościowe oznaczenie białka
Wiele metod pozwala na ilościową ocenę białek za pomocą spektrometrii mas, a najnowsze postępy umożliwiły ilościowe oznaczenie tysięcy białek w pojedynczych komórkach. Zazwyczaj stabilne (np. nieradioaktywne) cięższe izotopy węgla ( 13 C) lub azotu ( 15 N) włącza się do jednej próbki, podczas gdy druga jest znakowana odpowiednimi lekkimi izotopami (np. 12 C i 14 N). Obie próbki miesza się przed analizą. Peptydy pochodzące z różnych próbek można rozróżnić na podstawie różnicy mas. Stosunek ich intensywności pików odpowiada względnemu stosunkowi obfitości peptydów (i białek). Najpopularniejsze metody znakowania izotopowego to SILAC (znakowanie stabilnych izotopów aminokwasami w hodowli komórkowej), znakowanie 18O katalizowane trypsyną, ICAT (znakowanie powinowactwa kodowane izotopowo), iTRAQ (znaczniki izobaryczne do oznaczania ilościowego względnego i bezwzględnego). „Półilościową” spektrometrię mas można przeprowadzić bez znakowania próbek. Zwykle odbywa się to za pomocą analizy MALDI (w trybie liniowym). Intensywność piku lub powierzchnia piku poszczególnych cząsteczek (zwykle białek) jest tutaj skorelowana z ilością białka w próbce. Jednakże indywidualny sygnał zależy od pierwotnej struktury białka, złożoności próbki i ustawień instrumentu. Inne rodzaje ilościowej spektrometrii mas „bez znaczników” wykorzystują zliczenia widmowe (lub zliczenia peptydów) strawionych białek jako sposób określenia względnych ilości białka.
Określanie struktury białek
Charakterystyki wskazujące na trójwymiarową strukturę białek można badać za pomocą spektrometrii mas na różne sposoby. Stosując sieciowanie chemiczne do łączenia części białka znajdujących się blisko siebie w przestrzeni, ale daleko od siebie w sekwencji, można wywnioskować informacje o ogólnej strukturze. Śledząc wymianę protonów amidowych na deuter z rozpuszczalnika, można zbadać dostępność różnych części białka dla rozpuszczalnika. Wymiana wodór-deuter Spektrometria mas jest stosowana do badania białek i ich konformacji od ponad 20 lat. Ten typ analizy strukturalnej białek może być odpowiedni w przypadku białek, które stanowią wyzwanie dla innych metod strukturalnych. Inną interesującą możliwością badań strukturalnych białek jest znakowanie kowalencyjne indukowane laserem. W tej technice miejsca białka wystawione na działanie rozpuszczalnika są modyfikowane rodnikami hydroksylowymi. Jego połączenie z szybkim mieszaniem wykorzystano w badaniach zwijania białek.
Proteogenomika
W tym, co obecnie powszechnie określa się mianem proteogenomiki , peptydy identyfikowane za pomocą spektrometrii mas są wykorzystywane do ulepszania adnotacji genów (na przykład miejsc startu genów) i adnotacji białek. Równoległa analiza genomu i proteomu ułatwia odkrycie modyfikacji potranslacyjnych i zdarzeń proteolitycznych, szczególnie przy porównywaniu wielu gatunków.