Sekwencjonowanie peptydów de novo
W spektrometrii mas sekwencjonowanie peptydów de novo jest metodą, w której sekwencja aminokwasowa peptydu jest określana za pomocą tandemowej spektrometrii mas .
Znajomość sekwencji aminokwasowej peptydów pochodzących z trawienia białka jest niezbędna do badania funkcji biologicznych białka. W dawnych czasach osiągano to za pomocą degradacji Edmana . Obecnie analiza za pomocą tandemowego spektrometru mas jest bardziej powszechną metodą rozwiązywania sekwencjonowania peptydów. Zasadniczo istnieją dwa podejścia: przeszukiwanie bazy danych i sekwencjonowanie de novo. Przeszukiwanie bazy danych jest prostą wersją, ponieważ dane widma masowego nieznanego peptydu są przesyłane i uruchamiane w celu znalezienia dopasowania ze znaną sekwencją peptydu, wybrany zostanie peptyd o najwyższym wyniku dopasowania. To podejście nie rozpoznaje nowych peptydów, ponieważ może pasować tylko do istniejących sekwencji w bazie danych. Sekwencjonowanie de novo to przypisanie fragmentów jonów z widma masowego. Do interpretacji używane są różne algorytmy, a większość instrumentów jest dostarczana z programami do sekwencjonowania de novo.
Fragmentacja peptydów
Peptydy są protonowane w trybie jonów dodatnich. Proton początkowo lokalizuje się na N-końcu lub łańcuchu bocznym reszty zasadowej, ale z powodu wewnętrznej solwatacji może poruszać się wzdłuż szkieletu, łamiąc się w różnych miejscach, co skutkuje różnymi fragmentami. Zasady fragmentacji są dobrze wyjaśnione w niektórych publikacjach.
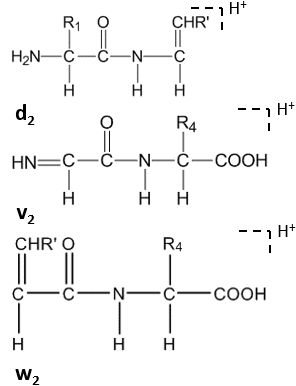
Trzy różne typy wiązań szkieletowych mogą zostać rozerwane, tworząc fragmenty peptydowe: wiązanie alkilokarbonylowe (CHR-CO), wiązanie peptydoamidowe (CO-NH) i wiązanie aminoalkilowe (NH-CHR).
Różne rodzaje jonów fragmentacyjnych
Kiedy wiązania szkieletowe ulegają rozszczepieniu, powstaje sześć różnych typów sekwencji jonów, jak pokazano na ryc. 1. N-końcowe naładowane jony fragmentacyjne są klasyfikowane jako a, b lub c, podczas gdy naładowane jony C-końcowe są klasyfikowane jako x, y lub z. Indeks dolny n to liczba reszt aminokwasowych. Nazewnictwo zostało po raz pierwszy zaproponowane przez Roepstorffa i Fohlmana, następnie Biemann je zmodyfikował i ta wersja stała się najpowszechniej akceptowaną.
Wśród tych jonów sekwencyjnych najczęściej spotykanymi typami jonów są jony a, b i y, zwłaszcza w niskoenergetycznych spektrometrach mas z dysocjacją wywołaną kolizją (CID), ponieważ wiązanie peptydoamidowe (CO-NH) jest najbardziej wrażliwe i utrata CO z jonów b.
Masa jonów b = Σ (masy pozostałości) + 1 (H + )
Masa jonów y = Σ (masy pozostałości) + 19 (H 2 O+H + )
Masa jonów a = masa jonów b – 28 (CO)
Rozszczepienie podwójnego szkieletu wytwarza jony wewnętrzne, typu acylowego, jak H 2 N-CHR 2 -CO-NH-CHR 3 -CO+ lub typu imonowego, jak H 2 N-CHR 2 -CO-NH + =CHR 3 . Jony te zwykle zakłócają widma.
Dalsze rozszczepianie zachodzi w wysokoenergetycznym CID w łańcuchu bocznym reszt C-końcowych, tworząc jony dn , vn , wn .
Podsumowanie zasad fragmentacji
Większość jonów fragmentacyjnych to jony b lub y. jony a są również często postrzegane przez utratę CO z jonów b.
Jony satelitarne(w n , v n , d n -jony) powstają w wyniku wysokoenergetycznego CID.
Jony zawierające Ser, Thr, Asp i Glu powodują neutralną utratę molekularną wody (-18).
Jony zawierające Asn-, Gln-, Lys-, Arg generują obojętny ubytek cząsteczkowy amoniaku (-17).
Obojętna utrata amoniaku z Arg prowadzi do jonów fragmentacyjnych (y-17) lub (b-17) o większej obfitości niż odpowiadające im jony.
Gdy C-koniec ma resztę zasadową, peptyd wytwarza jon (bn -1 +18).
Komplementarną parę jonową można zaobserwować w widmach wielokrotnie naładowanych jonów. W tym przypadku według pary jonowej suma ich indeksów dolnych jest równa całkowitej liczbie reszt aminokwasowych w nieznanym peptydzie.
Jeśli C-końcem jest Arg lub Lys, w widmie można znaleźć jon y1 , aby to udowodnić.
Metody fragmentacji peptydów
W niskoenergetycznej dysocjacji wywołanej kolizją (CID) jony b i y są głównymi jonami produkcyjnymi. Ponadto obserwuje się utratę amoniaku (-17 Da) we fragmencie zawierającym aminokwasy RKNQ. Utratę wody (-18 Da) można zaobserwować we fragmencie zawierającym aminokwasy STED. Na widmach nie widać żadnych jonów satelitarnych.
W wysokoenergetycznym CID można zaobserwować wszystkie rodzaje jonów fragmentacyjnych, ale nie ma strat amoniaku ani wody.
W dysocjacji z przeniesieniem elektronu (ETD) i dysocjacji z wychwytem elektronów (ECD) dominującymi jonami są jony c, y, z+1, z+2, a czasami w.
W przypadku rozpadu poźródłowego (PSD) w MALDI jony a, b, y są najczęstszymi jonami produkcyjnymi.
Czynniki wpływające na fragmentację to stan naładowania (im wyższy stan naładowania, tym mniej energii potrzeba do fragmentacji), masa peptydu (im większa masa, tym więcej energii potrzeba), energia indukowana (wyższa energia prowadzi do większej fragmentacji), pierwotna sekwencja aminokwasowa, tryb dysocjacji i gaz kolizyjny.
Wytyczne dotyczące interpretacji
W celu interpretacji najpierw poszukaj jonów imonowych pojedynczych aminokwasów (H 2 N + = CHR 2 ). Odpowiednie jony imonowe dla aminokwasów wymieniono w tabeli 1. Pomiń kilka pików na końcu widma o dużej masie. Są to jony, które ulegają stratom cząsteczek obojętnych (H 2 O, NH 3 , CO 2 , HCOOH) z jonów [M+H] + . Znajdź różnice masy przy 28 Da, ponieważ jony b mogą tworzyć jony a przez utratę CO. Poszukaj jonów b 2 na końcu widma o niskiej masie, co pomaga również zidentyfikować jony y n-2 . Masy b2 - jonów wymieniono w Tabeli 2, jak również pojedyncze aminokwasy, które mają masę równą masie b2 - jonów. Masa jonu b 2 = masa dwóch reszt aminokwasowych + 1.
Zidentyfikuj serię jonów sekwencji na podstawie tej samej różnicy mas, która pasuje do jednej z mas reszt aminokwasowych (patrz Tabela 1). Na przykład różnice masy między a n i a n-1 , b n i b n-1 , c n i c n-1 są takie same. Zidentyfikuj jon y n-1 na końcu widma o dużej masie. Następnie kontynuuj identyfikowanie jonów yn -2 , yn -3 ... przez dopasowanie różnic masy do mas reszt aminokwasowych (patrz Tabela 1). Poszukaj odpowiednich b-jonów zidentyfikowanych jonów y. Masa jonów b+y jest masą peptydu +2 Da. Po zidentyfikowaniu szeregu jonów y i szeregu jonów b, przypisz sekwencję aminokwasów i sprawdź masę. Inną metodą jest najpierw zidentyfikowanie jonów b, a następnie znalezienie odpowiednich jonów y.
Algorytmy i oprogramowanie
Ręczne sekwencjonowanie de novo jest pracochłonne i czasochłonne. Zwykle algorytmy lub programy dostarczane z instrumentem spektrometru mas są stosowane do interpretacji widm.
Opracowanie algorytmów sekwencjonowania de novo
Stara metoda polega na wymienieniu wszystkich możliwych peptydów dla jonu prekursora w widmie masowym i dopasowaniu widma masowego dla każdego kandydata do widma eksperymentalnego. Możliwy peptyd, który ma najbardziej podobne widmo, będzie miał największą szansę na uzyskanie właściwej sekwencji. Jednak liczba możliwych peptydów może być duża. Na przykład peptyd prekursorowy o masie cząsteczkowej 774 ma 21 909 046 możliwych peptydów. Nawet jeśli odbywa się to na komputerze, zajmuje to dużo czasu.
Inna metoda nazywa się „podsekwencjonowaniem”, która zamiast wymieniać całą sekwencję możliwych peptydów, dopasowuje krótkie sekwencje peptydów, które reprezentują tylko część całego peptydu. Gdy zostaną znalezione sekwencje, które bardzo pasują do jonów fragmentów w widmie eksperymentalnym, są one wydłużane o reszty jedna po drugiej, aby znaleźć najlepiej pasujące.
W trzeciej metodzie stosuje się graficzne przedstawienie danych, w którym jony fragmentów, które mają takie same różnice masy jednej reszty aminokwasowej, są połączone liniami. W ten sposób łatwiej uzyskać wyraźny obraz serii jonów tego samego typu. Ta metoda może być pomocna w ręcznym sekwencjonowaniu peptydów de novo, ale nie działa w przypadku warunków o wysokiej przepustowości.
Czwartą metodą, uważaną za skuteczną, jest teoria grafów. O zastosowaniu teorii grafów w sekwencjonowaniu peptydów de novo po raz pierwszy wspomniał Bartels. Piki w widmie są przekształcane w wierzchołki na grafie zwanym „wykresem widma”. Jeśli dwa wierzchołki mają taką samą różnicę masy jednego lub kilku aminokwasów, zostanie zastosowana skierowana krawędź. Algorytm SeqMS, algorytm Lutefisk, algorytm Sherenga to tylko niektóre przykłady tego typu.
Głęboka nauka
Niedawno do rozwiązania problemu sekwencjonowania peptydów de novo zastosowano techniki głębokiego uczenia. Pierwszym przełomem było DeepNovo, które przyjęło splotową strukturę sieci neuronowej, osiągnęło znaczną poprawę dokładności sekwencji i umożliwiło kompletną sekwencję białek bez wspomagania baz danych. surowe widmo. Problem sekwencjonowania peptydów de novo jest następnie przedstawiany jako problem przewidywania sekwencji. Biorąc pod uwagę wcześniej przewidywaną częściową sekwencję peptydu, modele sekwencjonowania peptydów de novo oparte na sieci neuronowej będą wielokrotnie generować najbardziej prawdopodobny następny aminokwas, aż masa przewidywanego peptydu będzie odpowiadać masie prekursora. W czasie wnioskowania można zastosować strategie wyszukiwania, takie jak przeszukiwanie wiązki, w celu zbadania większej przestrzeni poszukiwań przy jednoczesnym utrzymaniu niskich kosztów obliczeniowych. W porównaniu z poprzednimi metodami modele oparte na sieciach neuronowych wykazały znacznie lepszą dokładność i czułość. Co więcej, dzięki starannemu zaprojektowaniu modelu algorytmy sekwencjonowania peptydów de novo oparte na głębokim uczeniu mogą być również wystarczająco szybkie, aby uzyskać sekwencjonowanie peptydów de novo w czasie rzeczywistym. Oprogramowanie PEAKS uwzględnia uczenie się sieci neuronowych w swoich algorytmach sekwencjonowania de novo.
Pakiety oprogramowania
Jak opisali Andreotti i in. w 2012 roku Antilope jest połączeniem relaksacji Lagrange'a i adaptacją k najkrótszych ścieżek jena. Opiera się na metodzie „wykresu widma” i zawiera różne funkcje oceny, a pod względem czasu działania i dokładności może być porównywalny z „popularnymi najnowocześniejszymi programami ” PepNovo i NovoHMM.
Grossmanna i in. przedstawił AUDENS w 2005 roku jako zautomatyzowane narzędzie do sekwencjonowania peptydów de novo, zawierające moduł wstępnego przetwarzania, który może rozpoznawać szczyty sygnału i szczyty szumu.
Lutefisk może rozwiązać sekwencjonowanie de novo z widm masowych CID. W tym algorytmie najpierw znajdują się znaczące jony, a następnie określa się listę dowodów N- i C-końcowych. Na podstawie listy sekwencji generuje kompletne sekwencje w widmach i porównuje je z widmem eksperymentalnym. Jednak wynik może zawierać kilka kandydatów na sekwencje, które mają tylko niewielką różnicę, więc trudno jest znaleźć właściwą sekwencję peptydową. Drugi program, CIDentify, który jest zmodyfikowaną wersją algorytmu FASTA Billa Pearsona autorstwa Alexa Taylora, może zostać zastosowany do rozróżnienia tych niepewnych podobnych kandydatów.
Mo i in. przedstawił algorytm MSNovo w 2007 roku i udowodnił, że działa on „lepiej niż istniejące narzędzia de novo na wielu zestawach danych”. Algorytm ten umożliwia interpretację sekwencjonowania de novo spektrometrów masowych LCQ, LTQ oraz pojedynczych, podwójnych i potrójnie naładowanych jonów. W odróżnieniu od innych algorytmów zastosował nowatorską funkcję punktacji i używał tablicy mas zamiast wykresu widma.
Fisher i in. zaproponował metodę sekwencjonowania de novo NovoHMM. Ukryty model Markowa (HMM) jest stosowany jako nowy sposób rozwiązywania sekwencjonowania de novo w ramach bayesowskich. Zamiast oceniać pojedyncze symbole sekwencji, ta metoda uwzględnia późniejsze prawdopodobieństwa dla aminokwasów. W artykule udowodniono, że ta metoda ma lepszą wydajność niż inne popularne metody sekwencjonowania peptydów de novo, takie jak PepNovo, na podstawie wielu przykładowych widm.
PEAKS to kompletny pakiet oprogramowania do interpretacji widm masowych peptydów. Obejmuje sekwencjonowanie de novo, przeszukiwanie bazy danych, identyfikację PTM, wyszukiwanie homologii i kwantyfikację w analizie danych. Ma i in. opisali nowy model i algorytm sekwencjonowania de novo w PEAKS i porównali wydajność z Lutefiskiem kilku peptydów trypsynowych standardowych białek za pomocą kwadrupolowego spektrometru masowego czasu przelotu (Q-TOF).
PepNovo jest wysokowydajnym narzędziem do sekwencjonowania peptydów de novo i wykorzystuje sieć probabilistyczną jako metodę oceny. Interpretacja jednego widma zajmuje zwykle mniej niż 0,2 sekundy. Opisany przez Franka i in. , PepNovo działa lepiej niż kilka popularnych algorytmów, takich jak Sherenga, PEAKS, Lutefisk. Teraz dostępna jest nowa wersja PepNovo+.
Chi i in. przedstawił pNovo + w 2013 roku jako nowe narzędzie do sekwencjonowania peptydów de novo przy użyciu komplementarnych tandemowych widm masowych HCD i ETD. W tej metodzie algorytm składowy pDAG znacznie przyspiesza czas akwizycji sekwencjonowania peptydów średnio do 0,018 s, czyli trzy razy szybciej niż inne popularne oprogramowanie do sekwencjonowania de novo.
Jak opisali Jeong i in. , w porównaniu z innymi narzędziami do sekwencjonowania peptydów do novo, które działają dobrze tylko na niektórych typach widm, UniNovo jest bardziej uniwersalnym narzędziem, które ma dobrą wydajność na różnych typach widm lub parach widm, takich jak CID, ETD, HCD, CID/ETD, itp. Ma lepszą dokładność niż PepNovo+ czy PEAKS. Ponadto generuje wskaźnik błędów zgłoszonych sekwencji peptydowych.
Ma opublikował Novor w 2015 roku jako silnik sekwencjonowania peptydów de novo w czasie rzeczywistym. Narzędzie ma poprawić szybkość de novo o rząd wielkości i zachować podobną dokładność, jak inne narzędzia de novo na rynku. Na laptopie Macbook Pro Novor osiągnął ponad 300 widm MS/MS na sekundę.
Pevtsov i in. porównali wydajność powyższych pięciu algorytmów sekwencjonowania de novo: AUDENS, Lutefisk, NovoHMM, PepNovo i PEAKS. Dane ze spektrometru mas QSTAR i LCQ wykorzystano w analizie i oceniono na podstawie wartości względnej odległości sekwencji (RSD), która była podobieństwem między sekwencjonowaniem peptydów de novo i prawdziwą sekwencją peptydów obliczoną metodą programowania dynamicznego. Wyniki pokazały, że wszystkie algorytmy miały lepszą wydajność w danych QSTAR niż w danych LCQ, podczas gdy PEAKS jako najlepszy miał wskaźnik sukcesu 49,7% w danych QSTAR, a NovoHMM jako najlepszy miał wskaźnik sukcesu 18,3% w danych LCQ. Kolejność wyników w danych QSTAR była następująca: PEAKS > Lutefisk, PepNovo > AUDENS, NovoHMM, a w danych LCQ była następująca: NovoHMM > PepNovo, SZCZYTY > Lutefisk > AUDENS. Porównując zakres jakości widma, PEAKS i NovoHMM wykazały również najlepszą wydajność w obu danych spośród wszystkich 5 algorytmów. PEAKS i NovoHMM miały również najlepszą czułość zarówno w danych QSTAR, jak i LCQ. Jednak żaden oceniany algorytm nie przekroczył 50% dokładnej identyfikacji dla obu zestawów danych.
Niedawny postęp w spektrometrach mas umożliwił generowanie widm masowych o ultrawysokiej rozdzielczości [1] . Poprawiona dokładność, wraz ze zwiększoną ilością generowanych danych spektrometrii mas, przyciąga zainteresowanie zastosowaniem technik głębokiego uczenia się do sekwencjonowania peptydów de novo. W 2017 Tran i in. zaproponował DeepNovo, pierwsze oprogramowanie do sekwencjonowania de novo oparte na głębokim uczeniu się. Analiza porównawcza w oryginalnej publikacji wykazała, że DeepNovo znacznie przewyższał poprzednie metody, w tym PEAKS, Novor i PepNovo. DeepNovo jest zaimplementowane w pythonie z frameworkiem Tensorflow. Aby przedstawić widmo masowe jako wejście o stałym wymiarze do sieci neuronowej, DeepNovo zdyskretyzowało każde widmo do wektora o długości 150 000. Ta niepotrzebnie duża reprezentacja widma i użycie procesora jednowątkowego w oryginalnej implementacji uniemożliwia DeepNovo wykonywanie sekwencjonowania peptydów w czasie rzeczywistym. Aby jeszcze bardziej poprawić wydajność modeli sekwencjonowania peptydów de novo, Qiao i in. zaproponował PointNovo w 2020 roku. PointNovo to oprogramowanie Pythona zaimplementowane w ramach PyTorch, które eliminuje zajmującą miejsce reprezentację wektorów widma przyjętą przez DeepNovo. W porównaniu z DeepNovo, PointNovo udało się osiągnąć lepszą dokładność i wydajność w tym samym czasie, bezpośrednio reprezentując widmo jako zbiór par m/z i intensywności.