SZCZYTY
| Deweloperzy | Rozwiązania Bioinformatyczne Inc |
|---|---|
| Wersja stabilna | SZCZYTY 8,5 / 12 października 2017 r |
| System operacyjny | Okna |
| Typ | Identyfikacja białek na podstawie widma masowego, sekwencjonowanie de novo, przeszukiwanie bazy danych i oznaczanie ilościowe |
| Licencja | Własne oprogramowanie komercyjne |
| Strona internetowa | http://www.bioinfor.com/ |
PEAKS to oprogramowanie proteomiczne do tandemowej spektrometrii mas przeznaczone do sekwencjonowania peptydów , identyfikacji białek i oznaczania ilościowego.
Opis
PEAKS jest powszechnie używany do identyfikacji peptydów (identyfikator białka) poprzez sekwencjonowanie peptydów de novo wspomagane przeszukiwaniem bazy danych wyszukiwarek . PEAKS zintegrował również charakterystykę PTM i mutacji poprzez automatyczne znacznikach sekwencji peptydów (SPIDER) i identyfikację PTM.
PEAKS zapewnia między innymi kompletną sekwencję dla każdego peptydu, wyniki ufności dla poszczególnych przypisań aminokwasów, proste raportowanie do analizy o wysokiej przepustowości.
Oprogramowanie posiada możliwość porównywania wyników wielu wyszukiwarek. PEAKS inChorus automatycznie sprawdza wyniki testów z innymi wyszukiwarkami identyfikatorów białek, takimi jak Sequest, OMSSA, X!Tandem i Mascot. Takie podejście chroni przed fałszywie dodatnimi przypisaniami peptydów.
PEAKS Q to dodatkowe narzędzie do ilościowego oznaczania białek, wspierające techniki znakowania ( ICAT , iTRAQ , SILAC , TMT , 018 , itd.) i bez znaczników .
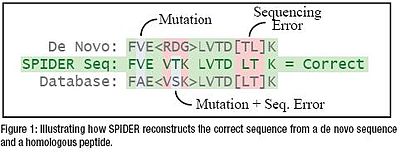
SPIDER to narzędzie wyszukiwania oparte na znacznikach sekwencji w ramach PEAKS, które zajmuje się możliwymi nakładającymi się błędami sekwencjonowania de novo i mutacjami homologii. Rekonstruuje rzeczywistą sekwencję peptydową, automatycznie i wydajnie łącząc znacznik sekwencji de novo i homolog.
Zbiór algorytmów wykorzystywanych w oprogramowaniu PEAKS został zaadaptowany i skonfigurowany w ramach specjalistycznego projektu PEAKS AB, który okazał się pierwszą metodą automatycznego sekwencjonowania przeciwciał monoklonalnych.