prezentacja mRNA
Prezentacja mRNA to technika prezentacji stosowana do ewolucji białek i/lub peptydów in vitro w celu stworzenia cząsteczek, które mogą wiązać się z pożądanym celem. W wyniku tego procesu powstają przetłumaczone peptydy lub białka , które są związane z ich prekursorem mRNA poprzez wiązanie puromycyny . Kompleks następnie wiąże się z unieruchomionym celem w etapie selekcji ( chromatografia powinowactwa ). Fuzje mRNA-białko, które dobrze się wiążą, są następnie poddawane odwrotnej transkrypcji do cDNA i ich sekwencji amplifikowany w reakcji łańcuchowej polimerazy . Rezultatem jest nukleotydowa , która koduje peptyd o wysokim powinowactwie do cząsteczki będącej przedmiotem zainteresowania.
Puromycyna jest analogiem końca 3' tyrozylo-tRNA, którego część struktury naśladuje cząsteczkę adenozyny , a druga część naśladuje cząsteczkę tyrozyny . W porównaniu z rozszczepialnym wiązaniem estrowym w tyrozylo-tRNA, puromycyna ma nieulegające hydrolizie wiązanie amidowe. W rezultacie puromycyna zakłóca translację i powoduje przedwczesne uwalnianie produktów translacji.

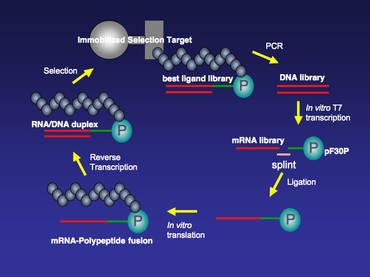
Wszystkie matryce mRNA stosowane w technologii prezentacji mRNA mają na końcu 3' puromycynę. W miarę postępu translacji rybosom porusza się wzdłuż matrycy mRNA, a gdy dotrze do końca 3' matrycy, połączona puromycyna wejdzie w miejsce A rybosomu i zostanie włączona do powstającego peptydu. Fuzja mRNA-polipeptyd jest następnie uwalniana z rybosomu (ryc. 1).
Aby zsyntetyzować fuzję mRNA-polipeptyd, połączona puromycyna nie jest jedyną modyfikacją matrycy mRNA. Oligonukleotydy i inne odstępniki muszą być rekrutowane wraz z puromycyną, aby zapewnić elastyczność i odpowiednią długość, aby puromycyna mogła wejść do miejsca A. Idealnie, łącznik między końcem 3' mRNA a puromycyną musi być elastyczny i wystarczająco długi, aby umożliwić puromycynie wejście do miejsca A po translacji ostatniego kodonu. Umożliwia to wydajną produkcję wysokiej jakości fuzji mRNA-polipeptyd o pełnej długości. Rihe Liu i in. zoptymalizowali odstępnik oligonukleotydowy 3'-puromycyny. Poinformowali, że dA25 w połączeniu z Spacer 9 (Glen Research) i dAdCdCP na końcu 5' działały najlepiej w reakcji fuzji. Odkryli, że łączniki dłuższe niż 40 nukleotydów i krótsze niż 16 nukleotydów wykazywały znacznie zmniejszoną wydajność tworzenia fuzji. Ponadto, gdy sekwencja rUrUP prezentowała się w sąsiedztwie puromycyny, fuzja nie tworzyła się wydajnie.
Oprócz zapewniania elastyczności i długości, część poli dA łącznika umożliwia również dalsze oczyszczanie fuzji mRNA-polipeptyd ze względu na jej wysokie powinowactwo do żywicy celulozowej dT. Fuzje mRNA-polipeptyd można selekcjonować względem unieruchomionych celów selekcyjnych przez kilka rund z rosnącą ostrością. Po każdej rundzie selekcji, te elementy biblioteki, które pozostają związane z unieruchomionym obiektem docelowym, metodą PCR , a elementy niewiążące są wypłukiwane.
metoda
Synteza biblioteki prezentacji mRNA rozpoczyna się od syntezy biblioteki DNA. Bibliotekę DNA dla dowolnego białka lub małego peptydu będącego przedmiotem zainteresowania można zsyntetyzować przez syntezę w fazie stałej, a następnie amplifikację PCR. Zwykle każdy członek tej biblioteki DNA ma miejsce transkrypcji polimerazy RNA T7 i miejsce wiązania rybosomu na końcu 5'. Region promotora T7 umożliwia transkrypcję T7 na dużą skalę in vitro na transkrypcję biblioteki DNA do biblioteki mRNA, która dostarcza szablonów do późniejszej reakcji translacji in vitro . Miejsce wiązania rybosomu w nieulegającym translacji regionie 5' (5' UTR) jest zaprojektowane zgodnie z in vitro , który ma być zastosowany. Istnieją dwa popularne komercyjnie dostępne systemy translacji in vitro . Jednym z nich jest E. coli S30 Extract System (Promega), który wymaga sekwencji Shine-Dalgarno w 5' UTR jako miejsca wiązania rybosomu; drugim jest Red Nova Lysate (Novagen), który potrzebuje rybosomalnego miejsca wiązania ΔTMV.
Gdy biblioteka mRNA zostanie wygenerowana, zostanie ona oczyszczona za pomocą mocznika-PAGE i zligowana przy użyciu ligazy DNA T4 z łącznikiem rozdzielającym DNA zawierającym puromycynę na końcu 3'. Na tym etapie ligacji kawałek mRNA jest ligowany z jednoniciowym DNA przy pomocy ligazy DNA T4. Nie jest to standardowa reakcja ligacji ligazy DNA T4, w której dwie części dwuniciowego DNA są łączone ze sobą. Aby zwiększyć wydajność tej specjalnej ligacji, można zastosować jednoniciową szynę DNA, aby wspomóc reakcję ligacji. Koniec 5' szyny jest zaprojektowany tak, aby był komplementarny do końca 3' mRNA, a koniec 3' szyny jest zaprojektowany tak, aby był komplementarny do końca 5' łącznika rozdzielającego DNA, który zwykle składa się z poli nukleotydy dA (ryc. 2).
Zligowana biblioteka mRNA-DNA-puromycyna jest poddawana translacji w Red Nova Lysate (Novagen) lub E. coli S30 Extract System (Promega), w wyniku czego powstają polipeptydy połączone kowalencyjnie w cis z kodującym mRNA. Translację in vitro można również przeprowadzić w systemie PURE (synteza białek przy użyciu elementów rekombinowanych). System PURE to E. coli bezkomórkowy system translacji, w którym obecne są tylko niezbędne komponenty translacji. Niektóre składniki, takie jak aminokwasy i syntazy aminoacylo-tRNA (AARS), można pominąć w systemie. Zamiast tego do systemu PURE można dodać chemicznie acylowany tRNA. Wykazano, że niektóre nienaturalne aminokwasy, takie jak acylowany N-metyloaminokwasem tRNA, mogą być włączane do peptydów lub fuzji mRNA-polipeptyd w systemie PURE.
Po translacji, jednoniciowe części mRNA fuzji zostaną przekształcone w heterodupleks RNA/DNA przez odwrotną transkryptazę w celu wyeliminowania wszelkich niepożądanych struktur drugorzędowych RNA i uczynienia części fuzji kwasu nukleinowego bardziej stabilną. Ten etap jest standardową reakcją odwrotnej transkrypcji. Na przykład można to zrobić za pomocą Superscript II (GIBCO-BRL) zgodnie z protokołem producenta.
Fuzje mRNA/DNA-polipeptyd można selekcjonować na unieruchomionych celach selekcyjnych przez kilka rund (Figura 3). Przez kilka pierwszych rund selekcji może istnieć stosunkowo wysokie tło, które można zminimalizować poprzez zwiększenie surowości selekcji, takiej jak dostosowanie stężenia soli, ilości detergentu i/lub temperatury podczas okresu wiązania celu/fuzji. Po selekcji wiązania, te elementy biblioteki, które pozostają związane z unieruchomionym celem, są amplifikowane metodą PCR. Etap amplifikacji PCR wzbogaci populację z biblioteki prezentacji mRNA, która ma większe powinowactwo do unieruchomionego celu. Podatny na błędy PCR można również przeprowadzić pomiędzy każdą rundą selekcji, aby jeszcze bardziej zwiększyć różnorodność biblioteki prezentacji mRNA i zredukować tło selekcji.
Niedawno opublikowano mniej czasochłonny protokół prezentacji mRNA.
Zalety
Chociaż istnieje wiele innych technologii prezentacji molekularnej, takich jak prezentacja fagowa , prezentacja bakteryjna , prezentacja drożdżowa i prezentacja rybosomalna , technologia prezentacji mRNA ma wiele zalet w stosunku do innych. Pierwsze trzy wymienione biblioteki prezentacji biologicznej mają polipeptydy lub białka eksprymowane na powierzchni odpowiedniego mikroorganizmu, a towarzysząca informacja kodująca dla każdego polipeptydu lub białka jest możliwa do odzyskania z genomu mikroorganizmu. Jednak rozmiar biblioteki dla tych trzech in vivo systemów wyświetlania jest ograniczona wydajnością transformacji każdego organizmu. Na przykład rozmiar biblioteki do prezentacji fagowej i bakteryjnej jest ograniczony do 1-10 × 10 ^ 9 różnych członków. Rozmiar biblioteki drożdży jest jeszcze mniejszy. Co więcej, te oparte na komórkach systemy wyświetlania pozwalają jedynie na przeszukiwanie i wzbogacanie peptydów/białek zawierających naturalne aminokwasy. W przeciwieństwie do tego, prezentacja mRNA i prezentacja rybosomalna są in vitro metody selekcji. Pozwalają na rozmiar biblioteki tak duży, jak 10 ^ 15 różnych członków. Duży rozmiar biblioteki zwiększa prawdopodobieństwo wybrania bardzo rzadkich sekwencji, a także poprawia różnorodność wybranych sekwencji. Ponadto in vitro usuwają niepożądaną presję selekcyjną, taką jak słaba ekspresja białka i szybka degradacja białka, co może zmniejszać różnorodność wybranych sekwencji. Wreszcie, in vitro pozwalają na zastosowanie technik mutagenezy i rekombinacji in vitro w całym procesie selekcji.
Chociaż zarówno prezentacja rybosomalna, jak i prezentacja mRNA są in vitro metody selekcji, prezentacja mRNA ma pewną przewagę nad technologią prezentacji rybosomalnej. Prezentacja mRNA wykorzystuje kowalencyjne kompleksy mRNA-polipeptyd połączone przez puromycynę; podczas gdy prezentacja rybosomalna wykorzystuje zatrzymane, niekowalencyjne kompleksy rybosom-mRNA-polipeptyd. W przypadku prezentacji rybosomów rygorystyczność selekcji jest ograniczona, aby utrzymać rybosom-mRNA-polipeptyd w kompleksie z powodu niekowalencyjnych kompleksów rybosom-mRNA-polipeptyd. Może to powodować trudności w zmniejszaniu wiązania tła podczas cyklu selekcji. Ponadto polipeptydy poddane selekcji w systemie prezentacji rybosomów są przyłączone do ogromnego kompleksu rRNA-białko, rybosomu, który ma masę cząsteczkową ponad 2 000 000 Da. Może wystąpić nieprzewidywalna interakcja między celem selekcji a rybosomem, co może prowadzić do utraty potencjalnych substancji wiążących podczas cyklu selekcji. Natomiast łącznik DNA puromycyny stosowany w technologii prezentacji mRNA jest znacznie mniejszy w porównaniu z rybosomem. Ten łącznik może mieć mniejsze szanse na interakcję z unieruchomionym celem selekcji. Zatem technologia wyświetlania mRNA z większym prawdopodobieństwem da mniej stronnicze wyniki.
Aplikacja
W 1997 roku Roberts i Szostak wykazali, że fuzje między syntetycznym mRNA i jego zakodowanym epitopem myc można wzbogacić z puli fuzji o przypadkowej sekwencji mRNA-polipeptyd przez immunoprecypitację.
Dziewięć lat później Fukuda i współpracownicy wybrali metodę prezentacji mRNA do ewolucji in vitro jednołańcuchowych fragmentów przeciwciał Fv (scFv). Wybrali sześć różnych mutantów scFv z pięcioma mutacjami konsensusowymi. Jednak analiza kinetyczna tych mutantów wykazała, że ich specyficzność względem antygenu pozostała podobna do swoistości typu dzikiego. Wykazali jednak, że dwie z pięciu mutacji konsensusowych znajdowały się w regionach determinujących komplementarność (CDR). Doszli do wniosku, że prezentacja mRNA ma potencjał do szybkiej sztucznej ewolucji diagnostycznych i terapeutycznych przeciwciał o wysokim powinowactwie poprzez optymalizację ich CDR.
Roberts i współpracownicy wykazali, że nienaturalne oligomery peptydowe składające się z N-podstawionego aminokwasu można zsyntetyzować jako fuzje mRNA-polipeptyd. Peptydy zawierające N-podstawione aminokwasy są związane z dobrą stabilnością proteolityczną i lepszymi właściwościami farmakokinetycznymi. Ta praca wskazuje, że technologia prezentacji mRNA ma potencjał do selekcji lekopodobnych peptydów do zastosowań terapeutycznych odpornych na proteolizę.