Peptydoglikan
Peptydoglikan lub mureina to unikalna duża makrocząsteczka, polisacharyd , składający się z cukrów i aminokwasów , który tworzy siatkową warstwę peptydoglikanu na zewnątrz błony komórkowej , sztywnej ściany komórkowej (murein sacculus) charakterystycznej dla większości bakterii ( domena Bacteria ). Składnik cukrowy składa się z naprzemiennych reszt N -acetyloglukozaminy (NAG) połączonych wiązaniami β-(1,4) i kwasu N -acetylomuraminowego (NAM). Do kwasu N -acetylomuraminowego przyłączony jest łańcuch oligopeptydowy składający się z trzech do pięciu aminokwasów. Łańcuch peptydowy może być usieciowany z łańcuchem peptydowym innej nici, tworząc trójwymiarową warstwę przypominającą siatkę. ścianie komórkowej bakterii, zapewniając wytrzymałość strukturalną, a także przeciwdziałając ciśnieniu osmotycznemu cytoplazmy . To powtarzające się łączenie skutkuje gęstą warstwą peptydoglikanu, która ma kluczowe znaczenie dla utrzymania formy komórki i wytrzymywania wysokich ciśnień osmotycznych, i jest regularnie zastępowana przez produkcję peptydoglikanu. Hydroliza i synteza peptydoglikanu to dwa procesy, które muszą zachodzić, aby komórki mogły rosnąć i namnażać się, technika przeprowadzana w trzech etapach: obcinanie obecnego materiału, wstawianie nowego materiału i ponowne sieciowanie istniejącego materiału w nowy materiał.
Warstwa peptydoglikanu jest znacznie grubsza u bakterii Gram-dodatnich (20 do 80 nanometrów) niż u bakterii Gram-ujemnych (7 do 8 nanometrów). W zależności od warunków wzrostu pH, peptydoglikan tworzy około 40 do 90% suchej masy ściany komórkowej bakterii Gram-dodatnich, ale tylko około 10% szczepów Gram-ujemnych. Zatem obecność wysokiego poziomu peptydoglikanu jest głównym wyznacznikiem charakterystyki bakterii jako Gram-dodatnich . W przypadku szczepów Gram-dodatnich jest to ważne w rolach przywiązania i serotypowaniu cele. Zarówno w przypadku bakterii Gram-dodatnich, jak i Gram-ujemnych, cząsteczki o wielkości około 2 nm mogą przechodzić przez peptydoglikan.
Za pomocą mikroskopu trudno jest stwierdzić, czy organizm jest Gram-dodatni, czy Gram-ujemny; Wymagane jest barwienie metodą Grama, stworzone przez Hansa Christiana Grama w 1884 roku. Bakterie barwi się kilkoma barwnikami, takimi jak fiolet krystaliczny, alkohol jodowy i safranina, stosując procedurę barwienia Grama. Komórki Gram-dodatnie po barwieniu są fioletowe, podczas gdy komórki Gram-ujemne są czerwone.
Struktura
Warstwa peptydoglikanu w ścianie komórkowej bakterii jest strukturą sieci krystalicznej utworzonej z liniowych łańcuchów dwóch naprzemiennych aminokwasów , a mianowicie N -acetyloglukozaminy (GlcNAc lub NAG) i kwasu N -acetylomuraminowego (MurNAc lub NAM). Naprzemienne cukry są połączone wiązaniem β-(1,4) -glikozydowym . Każdy MurNAc jest przyłączony do krótkiego (4- do 5-resztowego) aminokwasowego , zawierającego L -alaninę , kwas D -glutaminowy , mezo kwas -diaminopimelinowy i D -alanina w przypadku Escherichia coli (bakterie Gram-ujemne) lub L -alanina , D -glutamina , L -lizyna i D -alanina z mostkiem 5- glicynowym między tetrapeptydami w przypadku Staphylococcus aureus (bakteria Gram-dodatnia). Peptydoglikan jest jednym z najważniejszych źródeł D-aminokwasów w przyrodzie.
Otaczając wewnętrzną błonę, warstwa peptydoglikanu chroni komórkę przed lizą spowodowaną ciśnieniem turgoru komórki. Kiedy ściana komórkowa rośnie, zachowuje swój kształt przez całe życie, więc kształt pręta pozostanie kształtem pręta, a kształt kulisty pozostanie kształtem kulistym na całe życie. Dzieje się tak, ponieważ świeżo dodany materiał syntezy przegrody przekształca się w półkulistą ścianę dla komórek potomnych.
Sieciowanie między aminokwasami w różnych liniowych łańcuchach aminocukrowych zachodzi za pomocą enzymu DD-transpeptydazy i skutkuje trójwymiarową strukturą, która jest mocna i sztywna. Specyficzna sekwencja aminokwasów i struktura cząsteczkowa różnią się w zależności od gatunku bakterii .
Opisano różne typy peptydoglikanów ścian komórkowych bakterii i ich implikacje taksonomiczne. Archaea ( domena Archaea ) nie zawierają peptydoglikanu (mureiny). Niektóre Archaea zawierają pseudopeptydoglikan (pseudomureinę, patrz poniżej).
Struktura peptydoglikanu. NAG = N -acetyloglukozamina (zwana także GlcNAc lub NAGA), NAM = kwas N -acetylomuraminowy (zwany także MurNAc lub NAMA).
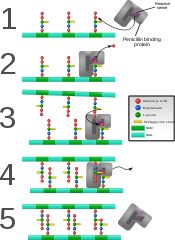
Białko wiążące penicylinę tworzące wiązania poprzeczne w nowo powstałej ścianie komórkowej bakterii.
Peptydoglikan bierze udział w rozszczepieniu binarnym podczas reprodukcji komórek bakteryjnych. Bakterie i mykoplazmy typu L , oba pozbawione ścian komórkowych peptydoglikanu, nie rozmnażają się przez rozszczepienie binarne, ale przez mechanizm pączkowania.
W toku wczesnej ewolucji sukcesywne tworzenie się granic (błon, ścian) chroniących pierwsze struktury życia przed ich środowiskiem musiało mieć zasadnicze znaczenie dla powstania pierwszych komórek (celularyzacja).
Wynalezienie sztywnych ścian komórkowych peptydoglikanu (mureiny) u bakterii (domena Bacteria ) było prawdopodobnie warunkiem ich przetrwania, ekstensywnego promieniowania i kolonizacji praktycznie wszystkich siedlisk geosfery i hydrosfery.
Biosynteza
Monomery peptydoglikanów są syntetyzowane w cytosolu , a następnie przyłączane do baktoprenolu będącego nośnikiem błonowym . Baktoprenol transportuje monomery peptydoglikanu przez błonę komórkową, gdzie są one wstawiane do istniejącego peptydoglikanu.
- W pierwszym etapie syntezy peptydoglikanu glutamina , która jest aminokwasem, przekazuje grupę aminową do cukru, fruktozo-6-fosforanu . Ta reakcja, katalizowana przez EC 2.6.1.16 (GlmS), zamienia fruktozo-6-fosforan w 6-fosforan glukozaminy.
- W drugim etapie grupa acetylowa jest przenoszona z acetylo-CoA do grupy aminowej 6-fosforanu glukozaminy, tworząc N-acetylo-glukozamino-6-fosforan. Ta reakcja to EC 5.4.2.10 , katalizowana przez GlmM.
- W trzecim etapie procesu syntezy N -acetylo-glukozamino-6-fosforan ulega izomeryzacji, która zmieni N -acetylo-glukozamino-6-fosforan w N-acetylo-glukozamino-1-fosforan. To jest EC 2.3.1.157 , katalizowane przez GlmU.
- W kroku 4 N -acetylo-glukozamino-1-fosforan, który jest teraz monofosforanem, atakuje UTP . Trójfosforan urydyny, będący nukleotydem pirymidynowym , ma zdolność działania jako źródło energii. W tej konkretnej reakcji, po tym jak monofosforan zaatakował UTP, wydziela się nieorganiczny pirofosforan, który jest zastępowany przez monofosforan, tworząc UDP-N-acetyloglukozaminę (2,4). (Kiedy UDP jest używany jako źródło energii, wydziela nieorganiczny fosforan.) Ten początkowy etap służy do stworzenia prekursora NAG w peptydoglikanie. To jest WE 2.7.7.23 , również katalizowany przez GlmU, który jest enzymem dwufunkcyjnym.
- W etapie 5 część UDP-N-acetyloglukozaminy (UDP-GlcNAc) jest przekształcana w UDP-MurNAc (kwas UDP-N-acetylomuraminowy) przez dodanie grupy laktylowej do glukozaminy. Również w tej reakcji grupa hydroksylowa C3 usunie fosforan z węgla alfa fosfoenolopirogronianu . Tworzy to tak zwaną pochodną enolu. EC 2.5.1.7 , katalizowany przez MurA.
- W etapie 6 enol jest redukowany do „ugrupowania laktylowego” przez NADPH w etapie szóstym. EC 1.3.1.98 , katalizowany przez MurB.
- W kroku 7 UDP-MurNAc jest przekształcany w pentapeptyd UDP-MurNAc przez dodanie pięciu aminokwasów, zwykle w tym dipeptydu D -alanylo- D -alaniny. Jest to ciąg trzech reakcji: EC 6.3.2.8 autorstwa MurC, EC 6.3.2.9 autorstwa MurD i EC 6.3.2.13 autorstwa MurE.
Każda z tych reakcji wymaga źródła energii ATP. To wszystko jest określane jako etap pierwszy.
Etap drugi zachodzi w błonie cytoplazmatycznej. To właśnie w błonie komórkowej nośnik lipidowy zwany baktoprenolem przenosi prekursory peptydoglikanu przez błonę komórkową.
- Fosforan undekaprenylu zaatakuje penta UDP-MurNAc, tworząc penta PP-MurNac, która jest teraz lipidem (lipidem I). EC 2.7.8.13 autorstwa MraY.
- UDP-GlcNAc jest następnie transportowany do MurNAc, tworząc Lipid-PP-MurNAc penta-GlcNAc ( lipid II ), disacharyd, również prekursor peptydoglikanu. EC 2.4.1.227 autorstwa MurG.
- Lipid II jest transportowany przez błonę przez flippazę (MurJ), odkrycie dokonane w 2014 roku po dziesięcioleciach poszukiwań. Gdy już się tam znajdzie, jest dodawany do rosnącego łańcucha glikanu przez enzym glikozylotransferazę peptydoglikanu (GTase, EC 2.4.1.129). Ta reakcja jest znana jako transglikozylacja. W reakcji grupa hydroksylowa GlcNAc przyłączy się do MurNAc w glikanie, co spowoduje wyparcie lipidu-PP z łańcucha glikanu.
- W ostatnim etapie transpeptydaza DD (TPaza, EC 3.4.16.4) łączy sieciowo poszczególne łańcuchy glikanów. Białko to jest również znane jako białko wiążące penicylinę . Niektóre wersje enzymu pełnią również funkcję glikozylotransferazy, podczas gdy inne pozostawiają to zadanie odrębnemu enzymowi.
pseudopeptydoglikan
U niektórych archeonów , tj. przedstawicieli Methanobacteriales iw rodzaju Methanopyrus , znaleziono pseudopeptydoglikan (pseudomureinę). W pseudopeptydoglikanie reszty cukrowe to N -acetyloglukozamina z wiązaniami β-(1,3) i kwas N -acetylotalosaminuronowy . To sprawia, że ściany komórkowe takich archeonów są niewrażliwe na lizozym . Opisano biosyntezę pseudopeptydoglikanu.
Rozpoznawanie przez układ odpornościowy
Rozpoznawanie peptydoglikanów jest procesem konserwatywnym ewolucyjnie. Ogólna struktura jest podobna u różnych gatunków bakterii, ale różne modyfikacje mogą zwiększyć różnorodność. Należą do nich modyfikacje długości polimerów cukrowych, modyfikacje struktur cukrowych, zmiany usieciowania lub substytucje aminokwasów (głównie w trzeciej pozycji). Celem tych modyfikacji jest zmiana właściwości ściany komórkowej, która odgrywa istotną rolę w patogenezie .
Peptydoglikany mogą być rozkładane przez kilka enzymów ( lizozym , glukozaminidaza, endopeptydaza …), wytwarzając fragmenty immunostymulujące (czasami nazywane muropeptydami), które są krytyczne w pośredniczeniu w interakcjach gospodarz-patogen . Należą do nich MDP ( dipeptyd muramylowy ), NAG ( N-acetyloglukozamina ) czy iE-DAP (kwas γ-d-glutamylo-mezo-diaminopimelinowy).
Peptydoglikan z bakterii jelitowych (zarówno patogenów, jak i komensali) przekracza barierę jelitową nawet w warunkach fizjologicznych. Mechanizmy, przez które peptydoglikan lub jego fragmenty dostają się do komórek gospodarza, mogą być bezpośrednie (niezależne od nośnika) lub pośrednie (zależne od nośnika) i pośredniczą w nich bakterie (układy wydzielnicze, pęcherzyki błonowe) lub komórka gospodarza ( receptor pośredniczone, transportery peptydowe). Bakteryjne systemy wydzielnicze to kompleksy białkowe stosowane do dostarczania czynników wirulencji przez otoczkę komórki bakteryjnej do środowiska zewnętrznego. Wewnątrzkomórkowe patogeny bakteryjne atakują komórki eukariotyczne (co może prowadzić do powstawania fagolizosomów i/lub aktywacji autofagii ) lub bakterie mogą zostać pochłonięte przez fagocyty ( makrofagi , monocyty , neutrofile …). Fagosom zawierający bakterie może następnie łączyć się z endosomami i lizosomami , prowadząc do degradacji bakterii i wytwarzania polimerycznych fragmentów peptydoglikanu i muropeptydów.
Receptory
Wrodzony układ odpornościowy wykrywa nienaruszone fragmenty peptydoglikanu i peptydoglikanu za pomocą licznych PRR ( receptorów rozpoznawania wzorców ), które są wydzielane, wyrażane wewnątrzkomórkowo lub wyrażane na powierzchni komórki.
Białka rozpoznające peptydoglikany
PGLYRP są konserwowane od owadów po ssaki . Ssaki wytwarzają cztery wydzielane rozpuszczalne białka rozpoznające peptydoglikan ( PGLYRP-1 , PGLYRP-2 , PGLYRP-3 i PGLYRP-4 ), które rozpoznają pentapeptyd lub tetrapeptyd muramylu. Mogą również wiązać się z LPS i innymi cząsteczkami, wykorzystując miejsca wiązania poza rowkiem wiążącym peptydoglikan. Po rozpoznaniu peptydoglikanu, PGLYRP aktywują oksydazę polifenolową (PPO), szlaki sygnałowe Toll lub niedobór odporności (IMD). Prowadzi to do produkcji peptydów przeciwdrobnoustrojowych (AMP).
Każdy z ssaczych PGLYRP wykazuje unikalne wzorce ekspresji tkankowej. PGLYRP-1 ulega ekspresji głównie w ziarnistościach neutrofili i eozynofili . PGLYRP-3 i 4 są wyrażane przez kilka tkanek, takich jak skóra, gruczoły potowe, oczy lub przewód pokarmowy. homodimery i heterodimery połączone wiązaniami disiarczkowymi, które są niezbędne do ich działania bakteriobójczego. Ich wiązanie z peptydoglikanami ściany komórkowej bakterii może indukować śmierć komórki bakteryjnej poprzez interakcję z różnymi bakteryjnymi białkami regulującymi transkrypcję. PGLYRP mogą pomagać w zabijaniu bakterii poprzez współpracę z innymi PRR w celu zwiększenia rozpoznawania bakterii przez fagocyty.
PGLYRP-2 jest głównie wyrażany w wątrobie i wydzielany do krążenia. Ponadto jego ekspresja może być indukowana w keratynocytach skóry , komórkach nabłonka jamy ustnej i jelit . W przeciwieństwie do innych PGLYRP, PGLYRP-2 nie ma bezpośredniego działania bakteriobójczego. Posiada aktywność amidazy peptydoglikanowej, hydrolizuje wiązanie laktyloamidowe pomiędzy MurNAc a pierwszym aminokwasem peptydu macierzystego peptydoglikanu. Sugeruje się, że funkcją PGLYRP-2 jest zapobieganie nadmiernej aktywacji układu odpornościowego i stanom zapalnym -indukowane uszkodzenie tkanki w odpowiedzi na ligandy NOD2 (patrz poniżej), ponieważ te muropeptydy nie mogą już być rozpoznawane przez NOD2 po oddzieleniu składnika peptydowego od MurNAc. Coraz więcej dowodów sugeruje, że członkowie rodziny białek rozpoznających peptydoglikan odgrywają dominującą rolę w tolerancji komórek nabłonka jelitowego na mikroflorę komensalną. Wykazano, że ekspresja PGLYRP-2 i 4 może wpływać na skład mikrobioty jelitowej .
Ostatnio odkryto, że PGLYRP (a także receptory NOD-podobne i transportery peptydoglikanów) ulegają silnej ekspresji w rozwijającym się mózgu myszy . PGLYRP-2 ulega silnej ekspresji w neuronach kilku regionów mózgu, w tym w korze przedczołowej , hipokampie i móżdżku , co wskazuje na potencjalny bezpośredni wpływ peptydoglikanu na neurony. PGLYRP-2 ulega silnej ekspresji również w korze mózgowej małych dzieci, ale nie w większości dorosłych tkanek korowych. PGLYRP-1 jest również wyrażany w mózgu i nadal ulega ekspresji w wieku dorosłym.
Receptory typu NOD
Prawdopodobnie najbardziej znanymi receptorami peptydoglikanu są receptory typu NOD (NLR), głównie NOD1 i NOD2 . Receptor NOD1 jest aktywowany po związaniu iE-DAP (kwas γ-d-glutamylo-mezo-diaminopimelinowy), podczas gdy NOD2 rozpoznaje MDP (dipeptyd muramylowy), poprzez ich domeny LRR . Aktywacja prowadzi do samooligomeryzacji, co skutkuje aktywacją dwóch kaskad sygnalizacyjnych. Jeden wyzwala aktywację NF-kB (poprzez RIP2, TAK1 i IKK ), drugi prowadzi do MAPK kaskada sygnalizacyjna. Aktywacja tych szlaków indukuje produkcję zapalnych cytokin i chemokin .
NOD1 jest wyrażany przez różne typy komórek, w tym fagocyty mieloidalne, komórki nabłonkowe i neurony. NOD2 ulega ekspresji w monocytach i makrofagach, nabłonkowych komórkach jelitowych, komórkach Panetha , komórkach dendrytycznych , osteoblastach , keratynocytach i innych typach komórek nabłonkowych. Jako cytozolowe , NOD1 i NOD2 muszą albo wykrywać bakterie, które dostają się do cytozolu, albo peptydoglikan musi ulec degradacji w celu wytworzenia fragmentów, które muszą zostać przetransportowane do cytozolu, aby te czujniki działały.
Ostatnio wykazano, że NLRP3 jest aktywowany przez peptydoglikan poprzez mechanizm niezależny od NOD1 i NOD2. Stwierdzono, że w makrofagach N-acetyloglukozamina wytwarzana przez degradację peptydoglikanu hamuje aktywność heksokinazy i indukuje jej uwalnianie z błony mitochondrialnej . Promuje inflamasomu NLRP3 poprzez mechanizm wyzwalany przez zwiększoną przepuszczalność błony mitochondrialnej.
NLRP1 jest również uważany za cytoplazmatyczny czujnik peptydoglikanu. Może wykrywać MDP i promować IL-1 poprzez wiązanie NOD2.
Receptory lektyny typu C (CLR)
Lektyny typu C są zróżnicowaną nadrodziną białek zależnych głównie od Ca2 + , które wiążą różne węglowodany (w tym szkielet glikanu peptydoglikanu) i działają jako wrodzone receptory odpornościowe. Białka CLR, które wiążą się z peptydoglikanem, obejmują MBL ( lektyna wiążąca mannozę ), fikoliny , Reg3A (białko z rodziny genów regeneracji 3A) i PTCLec1. U ssaków inicjują szlak lektynowy kaskady dopełniacza .
Receptory toll-podobne
Rola TLR w bezpośrednim rozpoznawaniu peptydoglikanu jest kontrowersyjna. W niektórych badaniach stwierdzono, że peptydoglikan jest wykrywany przez TLR2 . Ale ta aktywność indukująca TLR2 może być spowodowana lipoproteinami ściany komórkowej i kwasami lipotejchojowymi , które zwykle współoczyszczają się z peptydoglikanem. Również różnice w strukturze peptydoglikanu w bakteriach z gatunku na gatunek mogą przyczynić się do różnych wyników na ten temat.
Jako szczepionka lub adiuwant
Peptydoglikan jest aktywny immunologicznie i może stymulować komórki układu odpornościowego do zwiększania ekspresji cytokin i wzmacniania swoistej odpowiedzi zależnej od przeciwciał w połączeniu ze szczepionką lub jako sam adiuwant . MDP, który jest podstawową jednostką peptydoglikanu, był początkowo stosowany jako aktywny składnik adiuwanta Freunda . Peptydoglikan ze Staphylococcus aureus zastosowano jako szczepionkę do ochrony myszy, wykazując, że po wstrzyknięciu szczepionki przez 40 tygodni myszy przeżyły prowokację S. aureus przy zwiększonej dawce śmiertelnej .
Hamowanie i degradacja
Niektóre leki przeciwbakteryjne, takie jak penicylina , zakłócają wytwarzanie peptydoglikanu przez wiązanie się z enzymami bakteryjnymi znanymi jako białka wiążące penicylinę lub transpeptydazy DD . Białka wiążące penicylinę tworzą wiązania między sieciującymi wiązaniami oligopeptydowymi w peptydoglikanie. Aby komórka bakteryjna mogła się rozmnażać przez rozszczepienie binarne , ponad milion podjednostek peptydoglikanu (NAM-NAG + oligopeptyd) musi być przyłączonych do istniejących podjednostek. Mutacje w genach kodujących transpeptydazy, które prowadzą do zmniejszenia interakcji z antybiotykiem, są istotnym źródłem pojawiającej się oporności na antybiotyki . Ponieważ peptydoglikanu nie ma również w bakteriach L i mykoplazmach, oba są oporne na penicylinę.
Celować można również w inne etapy syntezy peptydoglikanu. Miejscowy antybiotyk bacytracyna ma na celu wykorzystanie pirofosforanu C55-izoprenylu . Lantybiotyki , w tym środek konserwujący żywność nizyna , atakują lipid II.
Lizozym , który znajduje się we łzach i jest częścią wrodzonego układu odpornościowego organizmu, działa przeciwbakteryjnie poprzez rozrywanie wiązań β-(1,4)-glikozydowych w peptydoglikanie (patrz wyżej). Lizozym skuteczniej działa na bakterie Gram-dodatnie , w których ściana komórkowa peptydoglikanu jest odsłonięta, niż na bakterie Gram-ujemne , które mają zewnętrzną warstwę LPS pokrywa warstwę peptydoglikanu. Kilka modyfikacji peptydoglikanu bakteryjnego może skutkować odpornością na degradację przez lizozym. Na podatność bakterii na degradację istotny wpływ ma również ekspozycja na antybiotyki . Odsłonięte bakterie syntetyzują peptydoglikan, który zawiera krótsze łańcuchy cukrowe, które są słabo usieciowane i ten peptydoglikan jest następnie łatwiej degradowany przez lizozym.
Zobacz też
Linki zewnętrzne
- Schematyczne przedstawienie struktur peptydoglikanu.
- Struktura hydrolazy 6-fosforanowej MurNAc (MurQ) z Haemophilus influenzae ze związanym inhibitorem.