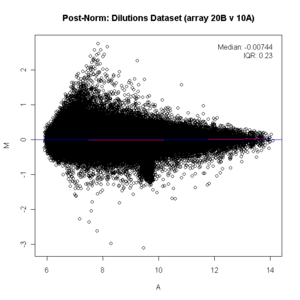
Działka MA
W biologii obliczeniowej wykres MA jest zastosowaniem wykresu Blanda-Altmana do wizualnej reprezentacji danych genomowych . Wykres wizualizuje różnice między pomiarami wykonanymi w dwóch próbkach, przekształcając dane na skale M (stosunek logarytmiczny) i A ( średnia średnia ), a następnie wykreśla te wartości. Chociaż pierwotnie stosowano je w kontekście danych dotyczących ekspresji genów z dwukanałowej mikromacierzy DNA , wykresy MA są również wykorzystywane do wizualizacji analizy sekwencjonowania o wysokiej przepustowości .
Wyjaśnienie
z mikromacierzy są często normalizowane w macierzach w celu kontroli systematycznych odchyleń w sprzęganiu barwników i wydajności hybrydyzacji, a także innych technicznych odchyleń w sondach DNA i końcówce drukującej używanej do wykrywania macierzy. Minimalizując te systematyczne zmiany, można znaleźć prawdziwe różnice biologiczne. Aby określić, czy potrzebna jest normalizacja, można wykreślić Cy5 (R) w funkcji intensywności Cy3 (G) i sprawdzić, czy nachylenie linii wynosi około 1. Ulepszona metoda, która jest w zasadzie skalowanym, 45-stopniowym obrotem R względem Wykres G jest wykresem MA. Wykres MA jest wykresem rozkładu stosunku intensywności koloru czerwonego do zielonego („M”) wykreślonym przez średnią intensywność („A”). M i A są określone następującymi równaniami.
M jest zatem binarnym logarytmem stosunku intensywności (lub różnicą między logarytmami intensywności), a A jest średnią logarytmiczną intensywnością dla kropki na wykresie. Wykresy MA są następnie wykorzystywane do wizualizacji zależnego od intensywności stosunku surowych danych z mikromacierzy (mikromacierze zwykle wykazują tutaj błąd systematyczny, przy czym wyższe A skutkuje wyższym |M|, tj. im jaśniejsza plamka, tym bardziej prawdopodobne jest zaobserwowanie różnicy między próbką a kontrolą). Wykres MA umieszcza zmienną M na osi y , a A na osi x i daje szybki przegląd rozkładu danych.
W wielu eksperymentach z ekspresją genów na mikromacierzach bazowym założeniem jest to, że większość genów nie zauważyłaby żadnej zmiany w swojej ekspresji; dlatego większość punktów na y ( M ) znajdowałaby się w punkcie 0, ponieważ log(1) wynosi 0. Jeśli tak nie jest, wówczas do danych należy zastosować metodę normalizacji , taką jak LOESS , przed Analiza statystyczna. (Na poniższym diagramie zobacz czerwoną linię biegnącą poniżej znaku zerowego przed normalizacją, powinna być prosta. Ponieważ nie jest prosta, dane powinny zostać znormalizowane. Po normalizacji czerwona linia jest prosta na linii zerowej i pokazuje jako różowy/czarny.)
Pakiety
Kilka pakietów Bioconductor dla oprogramowania R zapewnia możliwość tworzenia wykresów MA. Należą do nich affy (ma.plot, mva.pairs), limma (plotMA), marray (maPlot) i edgeR (maPlot)
Podobne wykresy „RA” można wygenerować za pomocą funkcji raPlot w pakiecie caroline CRAN R.
Interaktywny wykres MA do filtrowania genów według wartości M, A i p, wyszukiwania według nazw lub za pomocą lassa i zapisywania wybranych genów jest dostępny jako R-Shiny code Enhanced-MA- Plot .
Przykład w języku programowania R
biblioteka ( affy ) if ( wymagane ( affydata )) { dane ( rozcieńczenie ) } y <- ( wyraż ( rozcieńczenie ) [, c ( "20B" , "10A" )]) x11 () map.plot ( rowMeans ( log2 ( y )), log2 ( y [, 1 ])
- log2 ( y [, 2 ]), cex = 1 ) tytuł ( "Dilutions Dataset (array 20B v 10A)" ) biblioteka ( preprocessCore ) # wykonaj normalizację kwantyli x <- normalize.quantiles ( y ) x11 () ma. wykres ( rowMeans ( log2 ( x )), log2 ( x [, 1 ])
- log2 ( x [, 2 ]), cex = 1 ) tytuł ( "Post Norm: Zestaw danych rozcieńczeń (tablica 20B v 10A)" )
Zobacz też
- Bibliografia _ McCarthy, DJ; Smyth, GK (11 listopada 2009). „edgeR: pakiet Bioconductor do analizy różnicowej ekspresji cyfrowych danych dotyczących ekspresji genów” . Bioinformatyka . 26 (1): 139–140. doi : 10.1093/bioinformatyka/btp616 . PMC 2796818 . PMID 19910308 .
- ^ Miłość, Michał I; Hubera, Wolfganga; Anders, Simon (5 grudnia 2014). „Moderowane oszacowanie zmiany krotności i dyspersji danych seq RNA za pomocą DESeq2” . Biologia genomu . 15 (12): 550. doi : 10.1186/s13059-014-0550-8 . PMC 4302049 . PMID 25516281 .
- ^ YH Yang , S Dudoit , P Luu, DM Lin, V Peng, J Ngai, TP Speed . (2002). Normalizacja danych z mikromacierzy cDNA: solidna metoda złożona, zajmująca się systematyczną zmiennością pojedynczego i wielu szkiełek. Badania nad kwasami nukleinowymi , tom. 30 (4) s. e15.
- ^ Dudoit, S , Yang, YH , Callow, MJ, Prędkość, TP . (2002). Statystyczne metody identyfikacji genów o zróżnicowanej ekspresji w eksperymentach z replikacją mikromacierzy cDNA. Stan. Grzech. 12:1 111-139