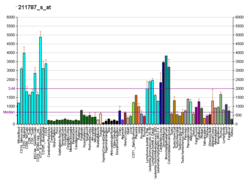
EIF4A1
| EIF4A1 | |||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
 | |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| Identyfikatory | |||||||||||||||||||||||||||||||||||||||||||||||
| , DDX2A, EIF-4A, EIF4A, eIF-4A-I, eIF4A-I, eukariotyczny czynnik inicjacji translacji 4A1 | |||||||||||||||||||||||||||||||||||||||||||||||
| Identyfikatory zewnętrzne | |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| Wikidane | |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
Eukariotyczny czynnik inicjacji 4A-I (znany również jako eIF4A1 lub DDX2A) jest białkiem cytozolowym o masie cząsteczkowej 46 kDa , które u ludzi jest kodowane przez gen EIF4A1 znajdujący się na chromosomie 17. Jest najbardziej rozpowszechnionym członkiem rodziny eIF4A Helikazy RNA zależne od ATP i odgrywają kluczową rolę w inicjacji zależnej od czapeczki translacji białek eukariotycznych jako składnik kompleksu inicjacji translacji eIF4F . eIF4A1 rozwija drugorzędową strukturę RNA w obrębie 5'-UTR mRNA , krytyczny etap niezbędny do rekrutacji kompleksu preinicjacyjnego 43S , a tym samym do translacji białka u eukariotów . Po raz pierwszy został scharakteryzowany w 1982 roku przez Grifo i in. , który oczyścił go z lizatu retikulocytów króliczych .
Tło
Regulacja translacji transkryptów mRNA na białko jest jednym z najlepszych sposobów, w jaki komórka może zmienić swoją reakcję na środowisko, ponieważ zmiany w transkrypcji genów często wymagają znacznie więcej czasu. Translację białek można podzielić na cztery fazy: aktywację, inicjację, wydłużenie i terminację. Spośród tych etapów inicjacja jest tym, nad którym komórki mają największą kontrolę. Jest to etap ograniczający szybkość syntezy białek, kontrolowany przez niezliczone białka znane jako eukariotyczne czynniki inicjujące lub eIF. Względna obfitość tych czynników lub ich względna indywidualna aktywność zapewniają komórkom eukariotycznym szeroką kontrolę nad szybkością inicjacji, a tym samym syntezą białek. eIF są regulowane w ramach dobrze znanych wewnątrzkomórkowych szlaków sygnalizacyjnych, takich jak szlak PI3K/AKT/mTOR , jednak inne biochemiczne warstwy regulacji, takie jak złożoność drugorzędowej struktury RNA w 5′-UTR, stają się oczywiste wraz z dalszymi badaniami.
Podrodzina eIF4A u ssaków składa się z trzech paralogów , eIF4A1, eIF4A2 i eIF4A3 . eIF4A1 i eIF4A2 mają 90% podobieństwa sekwencji i oba są białkami cytoplazmatycznymi, podczas gdy eIF4A3 jest zlokalizowany w jądrze i ma tylko 60% homologii . Historycznie eIF4A1 i eIF4A2 były uważane za wymienne, ponieważ obserwowano to w in vitro , ale dalsze badania wykazały, że eIF4A1 występuje częściej w dzielących się komórkach, podczas gdy eIF4A2 występuje częściej w komórkach niedzielących się, a ponadto nowsze dowody sugerują że mogą pełnić funkcjonalnie różne role in vivo .
Struktura
eIF4A1 jest członkiem rodziny helikaz RNA DEAD box . Helikazy RNA to enzymy, które wykorzystują energię uwolnioną z hydrolizy ATP do manipulowania drugorzędową strukturą RNA, a rodzina pudełek DEAD jest największą rodziną helikaz RNA. Nazwa „DEAD box” odnosi się do kluczowej sekwencji aminokwasowej DEAD na motywie II helikazy, która uczestniczy w trifosforanu nukleozydu (w przypadku eIF4A1, ATP ). Inne konserwatywne motywy , wspólne dla wszystkich białek z rodziny eIF4A, to motywy Q, I, Ia, Ib, III, IV, V i VI. Motywy Ia, Ib, IV i V wiążą RNA, motywy I, II i III pośredniczą w ATPazy zależnej od RNA , a motyw VI jest wymagany zarówno do wiązania RNA, jak i hydrolizy ATP.

Rodzina pudełek DEAD charakteryzuje się strukturalnie wysoce konserwowanym rdzeniem helikazy składającym się z dwóch domen podobnych do RecA , połączonych elastycznym regionem zawiasowym, wokół którego białko może się otwierać i zamykać po hydrolizie ATP. Szczelina utworzona między tymi dwiema domenami tworzy kieszeń wiążącą ATP. Cząsteczka RNA wiąże się przeciwnie do tej kieszeni wiążącej, rozciągając się w poprzek każdej z domen. Ten rdzeń jest otoczony przez zmienne domeny pomocnicze, które nadają im unikalną funkcję każdej helikazy RNA, częściowo umożliwiając specyficzne wiązanie z białkami pomocniczymi.
Funkcjonować
eIF4A1 jest helikazą RNA zależną od ATP, jednak dokładny charakter jej zależności od ATP w zakresie jej funkcji jest nadal przedmiotem dyskusji. Chociaż po związaniu ATP, późniejsza hydroliza indukuje zmiany konformacyjne w eIF4A1, wykazano, że inne helikazy RNA typu DEAD-box posiadają aktywność helikazy w obecności nieulegających hydrolizie analogów ATP, co sugeruje, że wiązanie, a nie hydroliza, jest ważniejszym elementem w czynność regulująca.
eIF4A1 jest składnikiem kompleksu inicjacji translacji eIF4F wraz z eIF4E , 5'-końcowym białkiem wiążącym czapeczkę i eIF4G , białkiem rusztowania, które utrzymuje razem eIF4A i eIF4E. Kompleksowi eIF4F często towarzyszą dodatkowe białka eIF4B i eIF4H , z których każde może w różny sposób zwiększać aktywność eIF4A1. Po transkrypcji mRNA z DNA i translokacji do cytoplazmy, a cytozolowy PABP jest związany z ogonem poli (A) powstającego mRNA, jego 5'-czapka zwiąże się z eIF4E, a PABP z eIF4G. eIF4A1 następnie rozwinie drugorzędową strukturę RNA od 5 'do 3', gdy PIC 43S jest rekrutowany do kompleksu eIF4F. 43S PIC skanuje również rozwinięty mRNA od 5' do 3', aż dotrze do kodonu start AUG , po czym podjednostka rybosomu 60S zostanie zwerbowana do rozpoczęcia procesu wydłużania.

(A) Wiązanie mRNA z kompleksem eIF4F. Należy zauważyć, że eIF4B i eIF4H mogą również wiązać się z eIF4A1, aby stymulować jego aktywność. (B) eIF4A1 rozwijanie drugorzędowej struktury mRNA i rekrutacja 43S PIC. (C) Podjednostka rybosomu 40S skanuje 5'-UTR transkryptu mRNA w poszukiwaniu kodonu start. (D) Rekrutacja podjednostki rybosomu 60S i początek wydłużania.
Rozporządzenie
Transkrypcja eIF4A1 jest sterowana przez czynnik transkrypcyjny MYC . Sama aktywność helikazy eIF4A1 jest słaba, jednak ta cecha nakłada praktyczne ograniczenie na eIF4A1, ponieważ niespecyficzna, „niezamierzona” aktywność helikazy w komórce byłaby szkodliwa dla funkcji niektórych endogennych, niezbędnych struktur RNA. Jego skuteczność znacznie wzrasta w obecności eIF4B i eIF4H, partnerów wiążących, którzy modulują jego aktywność. Kiedy eIF4B wiąże się z eIF4A1, aktywność helikazy eIF4A1 wzrasta ponad 100-krotnie, ale gdy zamiast tego wiąże się eIF4H, wzrost nie jest tak duży, co sugeruje, że różne względne stężenia tych białek pomocniczych mogą nadawać dalszy poziom regulacji wydajność eIF4A1.
I odwrotnie, aktywność eIF4A1 jest tłumiona, gdy jest związana z PDCD4 , supresorem nowotworu , który sam jest modulowany przez mTOR i miR-21 . PCDC4 jest zwykle zlokalizowany w jądrze w zdrowych komórkach, jednak w warunkach rakotwórczych przemieszcza się do jądra i wiążą się z nim dwie oddzielne cząsteczki eIF4A1, hamując zdolność eIF4A1 do wiązania się z RNA poprzez blokowanie cząsteczek w ich nieaktywnej konformacji, zapobiegając w ten sposób wiązaniu się z eIF4G.
Rola w chorobie
Rak
Dysregulacja translacji jest cechą charakterystyczną złośliwej transformacji komórek nowotworowych . Komórki nowotworowe w rosnących guzach stają się „uzależnione” od podwyższonego poziomu translacji białek, a szczególnie zależne od regulowanej w górę translacji proonkogennych mRNA. Te proonkogenne mRNA mają charakterystycznie dłuższe 5'-UTR z bardziej złożonymi strukturami drugorzędowymi, a regulacja w górę eIF4A1 jest powiązana z kilkoma ludzkimi nowotworami (patrz tabela). Biorąc pod uwagę ogólny trend nadekspresji eIF4A1 prowadzącej do raka, istnieje zainteresowanie opracowaniem inhibitorów tego enzymu. Kilka naturalnych związków zostało zidentyfikowanych jako potencjalne inhibitory rozwoju, chociaż hamują one zarówno eIF4A1, jak i eIF4A2 niespecyficznie. Należą do nich między innymi hippuristanol , silvestrol i pateamina A. W szczególności Silvestrol jest pochodną rokaglatu, a ta klasa związków może być żywotnymi inhibitorami eIF4A.
| Typ raka | eIF4A1 Dysregulacja / asocjacja |
|---|---|
| Rak wątrobowokomórkowy | nadekspresja |
| Czerniak | nadekspresja |
| Niedrobnokomórkowy rak płuc (NSCLC) | Wyrażenie związane z przerzutami |
| Rak endometrium | Nadekspresja w atypowym rozroście |
| Rak szyjki macicy | nadekspresja; zmniejszona ekspresja po brachyterapii związana z lepszym wynikiem |
| Rak piersi | Ekspresja związana ze złym wynikiem w chorobie z ujemnym receptorem estrogenu |
Infekcje wirusowe
Wirusy polegają na przejmowaniu maszynerii komórkowej zainfekowanych komórek w celu stworzenia własnych białek wirusowych i umożliwienia im dalszego infekowania nowych komórek. Dlatego ich zdolność do manipulowania eIF, takimi jak eIF4A1, znacząco wpływa na ich zjadliwość . Na przykład wirus cytomegalii opiera się na eIF4A, aby napędzać syntezę białek. Białko wirusowe pUL69 stabilizuje tworzenie eIF4F poprzez wiązanie z eIF4A, proces, w którym eIF4E zapobiega dysocjacji od kompleksu eIF4F. W związku z tym eIF4E nie może już być sekwestrowany przez swój negatywny regulator, 4EBP . Ponadto wirus cytomegalii stymuluje syntezę wszystkich elementów kompleksu eIF4F w celu napędzania syntezy białek. Inne wirusy, takie jak Cotesia plutellae bracovirus (CpBV), które faworyzują translację niezależną od czapeczki, wykorzystają eIF4A1 w odwrotnym kontekście, poprzez sekwestrację eIF4A1 z dala od kompleksu eIF4F z wirusowymi partnerami wiążącymi, w tym przypadku białkiem zwanym CpBV15β, w ten sposób hamowanie endogennej translacji mRNA zależnej od czapeczki i faworyzowanie translacji białek wirusowych. Związki wymienione w powyższej sekcji dotyczącej raka, hippuristanolu, silvestrolu, pateaminy A, pochodnych rokaglatu itp., można również zastosować jako domniemane inhibitory wirusów.
Dalsza lektura
- Reddy NS, Roth WW, Bragg PW, Wahba AJ (październik 1988). „Izolacja i mapowanie genu czynnika inicjacji syntezy białek 4A i jego ekspresji podczas różnicowania mysich komórek erytroleukemii”. gen . 70 (2): 231–43. doi : 10.1016/0378-1119(88)90195-3 . PMID 3215517 .
- Kukimoto I, Watanabe S, Taniguchi K, Ogata T, Yoshiike K, Kanda T (kwiecień 1997). „Charakterystyka sklonowanego promotora genu ludzkiego czynnika inicjacji 4AI”. Komunikaty dotyczące badań biochemicznych i biofizycznych . 233 (3): 844–7. doi : 10.1006/bbrc.1997.6555 . PMID 9168945 .
- Imataka H, Sonenberg N (grudzień 1997). „Ludzki eukariotyczny czynnik inicjacji translacji 4G (eIF4G) posiada dwa oddzielne i niezależne miejsca wiązania dla eIF4A” . Biologia molekularna i komórkowa . 17 (12): 6940–7. doi : 10.1128/mcb.17.12.6940 . PMC232551 . _ PMID 9372926 .
- Gradi A, Imataka H, Svitkin YV, Rom E, Raught B, Morino S, Sonenberg N (styczeń 1998). „Nowy funkcjonalny ludzki eukariotyczny czynnik inicjujący translację 4G” . Biologia molekularna i komórkowa . 18 (1): 334–42. doi : 10.1128/mcb.18.1.334 . PMC 121501 . PMID 9418880 .
- Craig AW, Haghighat A, Yu AT, Sonenberg N (kwiecień 1998). „Interakcja białka wiążącego poliadenylan z homologiem eIF4G PAIP poprawia translację”. Natura . 392 (6675): 520–3. Bibcode : 1998Natur.392..520C . doi : 10.1038/33198 . PMID 9548260 . S2CID 10891925 .
- Henis-Korenblit S, Strumpf NL, Goldstaub D, Kimchi A (styczeń 2000). „Nowa forma białka DAP5 gromadzi się w komórkach apoptotycznych w wyniku rozszczepienia kaspazy i translacji za pośrednictwem wewnętrznego miejsca wejścia rybosomu” . Biologia molekularna i komórkowa . 20 (2): 496–506. doi : 10.1128/MCB.20.2.496-506.2000 . PMC85113 . _ PMID 10611228 .
- Quinn CM, Wiles AP, El-Shanawany T, Catchpole I, Alnadaf T, Ford MJ i in. (grudzień 1999). „Ludzki gen czynnika inicjacji eukariotycznej 4AI (EIF4A1) zawiera wiele elementów regulatorowych, które kierują ekspresją genu reporterowego wysokiego poziomu w liniach komórkowych ssaków”. Genomika . 62 (3): 468–76. doi : 10.1006/geno.1999.6031 . PMID 10644445 .
- Cuesta R, Xi Q, Schneider RJ (lipiec 2000). „Translacja specyficzna dla adenowirusa przez przemieszczenie kinazy Mnk1 z kompleksu inicjacji czapeczki eIF4F” . Dziennik EMBO . 19 (13): 3465–74. doi : 10.1093/emboj/19.13.3465 . PMC 313943 . PMID 10880459 .
- Mendell JT, Medghalchi SM, Lake RG, Noensie EN, Dietz HC (grudzień 2000). „Nowe ortologi Upf2p sugerują funkcjonalny związek między inicjacją translacji a nonsensownymi kompleksami nadzoru” . Biologia molekularna i komórkowa . 20 (23): 8944–57. doi : 10.1128/MCB.20.23.8944-8957.2000 . PMC86549 . _ PMID 11073994 .
- Li W, Belsham GJ, Proud CG (sierpień 2001). „Eukariotyczne czynniki inicjujące 4A (eIF4A) i 4G (eIF4G) wzajemnie oddziałują w stosunku 1: 1 in vivo” . Journal of Biological Chemistry . 276 (31): 29111–5. doi : 10.1074/jbc.C100284200 . PMID 11408474 .
- Du MX, Johnson RB, Sun XL, Staschke KA, Colacino J, Wang QM (kwiecień 2002). „Porównawcza charakterystyka dwóch helikaz RNA typu DEAD-box w nadrodzinie II: ludzki czynnik inicjujący translację 4A i helikaza niestrukturalnego białka 3 (NS3) wirusa zapalenia wątroby typu C” . Dziennik biochemiczny . 363 (część 1): 147–55. doi : 10.1042/0264-6021:3630147 . PMC 1222461 . PMID 11903057 .
- Bohnsack MT, Regener K, Schwappach B, Saffrich R, Paraskeva E, Hartmann E, Görlich D (listopad 2002). „Exp5 eksportuje eEF1A przez tRNA z jąder i współdziała z innymi szlakami transportowymi, aby ograniczyć translację do cytoplazmy” . Dziennik EMBO . 21 (22): 6205–15. doi : 10.1093/emboj/cdf613 . PMC 137205 . PMID 12426392 .
- Yang HS, Cho MH, Zakowicz H, Hegamyer G, Sonenberg N, Colburn NH (maj 2004). „Nowa funkcja domen MA-3 w transformacji i supresji translacji Pdcd4 jest niezbędna do jego wiązania z eukariotycznym czynnikiem inicjującym translację 4A” . Biologia molekularna i komórkowa . 24 (9): 3894–906. doi : 10.1128/MCB.24.9.3894-3906.2004 . PMC 387765 . PMID 15082783 .
- Mingot JM, Bohnsack MT, Jäkle U, Görlich D (sierpień 2004). „Exportin 7 definiuje nową ogólną ścieżkę eksportu jądrowego” . Dziennik EMBO . 23 (16): 3227–36. doi : 10.1038/sj.emboj.7600338 . PMC 514512 . PMID 15282546 .
- Hinton TM, Coldwell MJ, Carpenter GA, Morley SJ, Pain VM (styczeń 2007). „Analiza funkcjonalna poszczególnych aktywności wiążących białka rusztowania eIF4G” . Journal of Biological Chemistry . 282 (3): 1695–708. doi : 10.1074/jbc.M602780200 . PMID 17130132 .
- Ewing RM, Chu P, Elisma F, Li H, Taylor P, Climie S i in. (2007). „Mapowanie na dużą skalę ludzkich interakcji białko-białko za pomocą spektrometrii mas” . Biologia systemów molekularnych . 3 (1): 89. doi : 10.1038/msb4100134 . PMC 1847948 . PMID 17353931 .