Niepełne sortowanie linii rodowych
Niekompletne sortowanie linii , określane również jako hemiplazja , głęboka koalescencja , zachowanie polimorfizmu przodków lub polimorfizm międzygatunkowy , opisuje zjawisko w genetyce populacji , gdy kopie genów przodków nie łączą się (patrząc wstecz w czasie) we wspólną kopię przodków, aż do głębiej niż poprzednio zdarzenia specjacyjne. Jest to spowodowane sortowaniem rodowodowym polimorfizmów genetycznych, które zostały zachowane w kolejnych węzłach drzewa gatunku. Innymi słowy, drzewo wytwarzane przez pojedynczy gen różni się od drzewa na poziomie populacji lub gatunku, tworząc niezgodne drzewo. Niezależnie od mechanizmu, wynik jest taki, że wygenerowane drzewo poziomu gatunku może się różnić w zależności od wybranych genów użytych do oceny. Kontrastuje to z całkowitym sortowaniem rodowodowym , w którym drzewo wytworzone przez gen jest takie samo jak drzewo na poziomie populacji lub gatunku. Oba są powszechnymi wynikami w analizie filogenetycznej, chociaż zależy to od genu, organizmu i techniki pobierania próbek.
Pojęcie
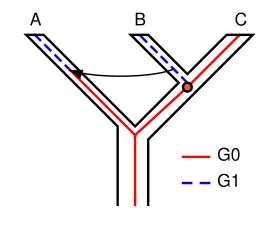
Koncepcja niepełnego sortowania linii ma pewne ważne implikacje dla technik filogenetycznych. Utrzymywanie się polimorfizmów w różnych zdarzeniach specjacji może powodować niepełne sortowanie linii. Załóżmy, że zachodzą dwa kolejne zdarzenia specjacyjne, w których gatunek przodka daje początek najpierw gatunkowi A, a następnie gatunkowi B i C. Podczas badania pojedynczego genu może on mieć wiele wersji ( allele ) powodujące pojawienie się różnych znaków (polimorfizmy). W przykładzie pokazanym na rycinie 1 gen G ma dwie wersje (allele), G0 i G1. Przodek A, B i C pierwotnie miał tylko jedną wersję genu G, G0. W pewnym momencie nastąpiła mutacja i populacja przodków stała się polimorficzna, przy czym niektóre osoby miały G0, a inne G1. Kiedy gatunek A oddzielił się, zachował tylko G1, podczas gdy przodek B i C pozostał polimorficzny. Kiedy B i C rozeszły się, B zachował tylko G1, a C tylko G0; żadne z nich nie było teraz polimorficzne w G. Drzewo genu G pokazuje A i B jako siostry, podczas gdy drzewo gatunków pokazuje B i C jako siostry. Jeśli filogeneza tych gatunków jest oparta na genie G, nie będzie odzwierciedlać rzeczywistych relacji między gatunkami. Innymi słowy, najbardziej spokrewnione gatunki niekoniecznie odziedziczą najbardziej spokrewnione geny. Jest to oczywiście uproszczony przykład niekompletnego sortowania linii, aw prawdziwych badaniach jest to zwykle bardziej złożone i zawiera więcej genów i gatunków.
Jednak inne mechanizmy mogą prowadzić do tej samej pozornej niezgodności, na przykład allele mogą przekraczać granice gatunków poprzez hybrydyzację, a DNA mogą być przenoszone między gatunkami przez wirusy. Jest to zilustrowane na rycinie 2. Tutaj przodek A, B i C oraz przodek B i C miał tylko wersję G0 genu G. Mutacja wystąpiła na rozbieżności B i C, a B nabył zmutowany wersja G1. Jakiś czas później strzałka pokazuje, że G1 został przeniesiony z B do A w jakiś sposób (np. hybrydyzacja lub poziomy transfer genów). Badanie tylko końcowych stanów G w trzech gatunkach sprawia, że wydaje się, że A i B są siostrami, a nie B i C, jak na rycinie 1, ale na rycinie 2 nie jest to spowodowane niepełnym sortowaniem linii.
Implikacje
Niekompletne sortowanie linii ma ważne implikacje dla badań filogenetycznych. Istnieje szansa, że podczas tworzenia drzewa filogenetycznego może ono nie przypominać rzeczywistych relacji z powodu tego niepełnego sortowania linii. Jednak przepływ genów między liniami poprzez hybrydyzację lub poziomy transfer genów mogą wytworzyć to samo sprzeczne drzewo filogenetyczne. Rozróżnienie tych różnych procesów może wydawać się trudne, ale prowadzi się wiele badań i różnych podejść statystycznych, aby uzyskać lepszy wgląd w tę ewolucyjną dynamikę. Jednym z rozwiązań mających na celu zmniejszenie skutków niepełnego sortowania linii jest wykorzystanie wielu genów do tworzenia filogenezy gatunków lub populacji. Im więcej użytych genów, tym bardziej niezawodna staje się filogeneza.
W organizmach diploidalnych
Niepełne sortowanie linii rodowych często ma miejsce podczas rozmnażania płciowego , ponieważ gatunku nie można prześledzić wstecz do jednej osoby lub pary hodowlanej. Kiedy populacje plemion organizmów są duże (tj. tysiące), każdy gen ma pewną różnorodność, a drzewo genów składa się z innych wcześniej istniejących linii. Jeśli populacja jest większa, te rodowody przodków przetrwają dłużej. Kiedy otrzymasz duże populacje przodków wraz z wydarzeniami specjacji w ściśle określonym czasie, te różne fragmenty DNA zachowują sprzeczne powiązania. Utrudnia to określenie wspólnego przodka lub punktów rozgałęzień.
W ewolucji naczelnych
Podczas badania naczelnych szympansy i bonobo są ze sobą bardziej spokrewnione niż jakiekolwiek inne taksony, a zatem są taksonami siostrzanymi . Jednak w przypadku 1,6% genomu bonobo sekwencje są bliżej spokrewnione z homologami ludzkimi niż z szympansami, co prawdopodobnie wynika z niepełnego sortowania linii. Badanie ponad 23 000 dopasowań sekwencji DNA w rodzinie Hominidae (wielkie małpy człekokształtne, w tym ludzie) wykazało, że około 23% nie potwierdza znanego siostrzanego związku szympansów i ludzi.
W ewolucji człowieka
W ewolucji człowieka niekompletne sortowanie linii jest używane do tworzenia diagramów linii homininów, które mogły nie zostać uporządkowane w tym samym czasie, gdy specjacja miała miejsce w prehistorii. Ze względu na pojawienie się testów genetycznych i sekwencjonowania genomu naukowcy odkryli, że powiązania genetyczne między liniami homininów mogą nie zgadzać się z wcześniejszym zrozumieniem ich pokrewieństwa na podstawie cech fizycznych. Ponadto rozbieżność ostatniego wspólnego przodka (LCA) niekoniecznie musi wystąpić w tym samym czasie co specjacja. Sortowanie rodowodowe to metoda, która pozwala paleoantropologom badać powiązania i rozbieżności genetyczne, które mogą nie pasować do ich poprzednich modeli specjacji opartych wyłącznie na filogenezie .
Niekompletne sortowanie rodowodowe ludzkiego drzewa genealogicznego jest obszarem wielkiego zainteresowania. Istnieje wiele niewiadomych, gdy rozważa się zarówno przejście od archaicznych ludzi do współczesnych ludzi, jak i rozbieżność innych małp człekokształtnych z linii homininów.
Dywergencja małpy i hominina / człowieka
Niepełne sortowanie rodowodowe oznacza, że średni czas rozbieżności między genami może różnić się od czasu rozbieżności między gatunkami. Modele sugerują, że średni czas rozbieżności między genami w genomie człowieka i szympansa jest starszy niż podział między ludźmi i gorylami. Oznacza to, że wspólny przodek ludzi i szympansów pozostawił ślady materiału genetycznego, który był obecny u wspólnego przodka ludzi, szympansów i goryli. Jednak drzewo genetyczne różni się nieco od drzewa gatunkowego lub filogenetycznego. W drzewie filogenezy, kiedy patrzymy na ewolucyjny związek między człowiekiem, bonobo szympansa i goryla, wyniki pokazują, że oddzielenie bonobo i szympansa nastąpiło w bliskiej odległości od wspólnego przodka przodka szympansa bonobo i ludzi, co wskazuje, że ludzie i szympansy mieli wspólnego przodka przez kilka milionów lat po separacji od goryli. Stwarza to zjawisko, jakim jest niepełne sortowanie linii. Obecnie naukowcy polegają na fragmentach DNA, aby badać ewolucyjne relacje między ludźmi a ich odpowiednikami w nadziei, że dostarczą informacji na temat specjacji i procesów przodków z genomów różnych typów ludzi.
W wirusach

Niepełne sortowanie linii jest powszechną cechą filodynamiki wirusów , gdzie filogeneza reprezentowana przez przenoszenie choroby z jednej osoby na drugą, czyli drzewo na poziomie populacji, często nie odpowiada drzewu utworzonemu na podstawie analizy genetycznej z powodu do wąskich gardeł populacji , które są nieodłączną cechą wirusowego przenoszenia chorób. Rysunek 3 ilustruje, w jaki sposób może to nastąpić. Ma to związek z przestępczym przenoszeniem wirusa HIV gdzie w niektórych sprawach karnych analiza filogenetyczna jednego lub dwóch genów ze szczepów oskarżonego i ofiary została wykorzystana do wywnioskowania transmisji; jednak powszechność niepełnego sortowania linii oznacza, że transmisji nie można wywnioskować wyłącznie na podstawie takiej podstawowej analizy.
W językoznawstwie
Jacques i List (2019) pokazują, że koncepcję niepełnego sortowania linii można zastosować do wyjaśnienia zjawisk niepodobnych do drzew w ewolucji języka. Kalyan i François (2019), zwolennicy metody historycznej glottometrii , modelu kwestionującego przydatność modelu drzewa w językoznawstwie historycznym, zgadzają się, że „Glottometria historyczna nie kwestionuje modelu drzewa genealogicznego po uwzględnieniu niepełnego sortowania linii. "
Zobacz też
- Gibon – ostatni wspólny przodek człowieka
- Sokół Laggara
- Teoria koalescencji
- Wielogatunkowy proces koalescencyjny
- Pochodzenie (ewolucja)
Linki zewnętrzne
- Venema, D. (2013-08-01). „Podstawy ewolucji: niekompletne sortowanie linii i rozmiary populacji przodków” . BioLogos . Źródło 29 czerwca 2018 r .
- Maddison, Wayne P.; Knowles, L. Lacey; Collins, Tim (2006). „Wnioskowanie o filogenezie pomimo niepełnego sortowania linii” . Biologia systematyczna . 55 (1): 21–30. doi : 10.1080/10635150500354928 . ISSN 1076-836X . PMID 16507521 .
- Joly, Szymon; McLenachan, Patricia A.; Lockhart, Peter J. (2009). „Statystyczne podejście do rozróżniania hybrydyzacji i niepełnego sortowania linii”. Amerykański przyrodnik . 174 (2): E54-E70. doi : 10.1086/600082 . PMID 19519219 . S2CID 205991354 .
- Carstens, Bryan C.; Knowles, L. Lacey; Collins, Tim (2007). „Szacowanie filogenezy gatunków na podstawie prawdopodobieństw drzew genetycznych pomimo niepełnego sortowania linii: przykład z koników polnych Melanoplus” . Biologia systematyczna . 56 (3): 400–411. doi : 10.1080/10635150701405560 . ISSN 1076-836X . PMID 17520504 .
- Scornavacca, C.; Galtier, N. (2017). „Niekompletne sortowanie linii w filogenetyce ssaków” . Biologia systematyczna . 66 (1): 112–120. doi : 10.1093/sysbio/syw082 . PMID 28173480 .