SOGA2
| Identyfikatory | |||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| MTCL1 | |||||||||||||||||||||||||||||||||||||||||||||||
| , CCDC165, KIAA0802, SOGA2, czynnik sieciowania mikrotubul 1 | |||||||||||||||||||||||||||||||||||||||||||||||
| Identyfikatory zewnętrzne | |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| Wikidane | |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
SOGA2 , znany również jako supresor związanej z autofagią glukozy 2 lub CCDC165 , jest białkiem , które u ludzi jest kodowane przez gen SOGA2 . SOGA2 ma dwóch ludzkich paralogów , SOGA1 i SOGA3 . U ludzi sekwencja kodująca gen ma długość 151 349 par zasad, mRNA ma 6092 par zasad, a sekwencja białkowa ma 1586 aminokwasów. Gen SOGA2 jest konserwowany u goryla, pawiana, galago, szczura, myszy, kota i innych. Istnieje odległa ochrona obserwowana w organizmach, takich jak zięby zebry i anole. SOGA2 ulega wszechobecnej ekspresji u ludzi, ze szczególnie wysoką ekspresją w mózgu (zwłaszcza móżdżku i hipokampie), jelicie grubym, przysadce mózgowej, jelicie cienkim, rdzeń kręgowy , jądra i mózg płodu.
Gen
Umiejscowienie
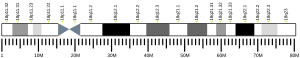
SOGA2 znajduje się od 8717369 do 8832775 na krótkim ramieniu chromosomu 18 (18p11.22).
Homologia i ewolucja
Paralogi
Istnieją dwa główne paralogi SOGA2: ludzkie białko SOGA1 i ludzkie białko SOGA3 . Wykazano, że SOGA1 bierze udział w supresji glukozy przez autofagię. Szybkość, z jaką ortologi odbiegają od człowieka SOGA2 (mierzona jako % identyczności), określa przybliżone zdarzenie duplikacji SOGA1 z SOGA2 na ~ 254,1 MYA, a zdarzenie duplikacji SOGA3 z SOGA2 ~ 329,1 MYA.
| nazwa białka | numer dostępowy | długość sekwencji (aa) | identyczność sekwencji z ludzkim białkiem | notatki |
| SOGA3 | NP_001012279.1 | 947 | 58% | konserwowane w ~ 500 N-końcowych aa |
| SOGA1 izoforma 2 | NP_954650.2 | 1016 aa | 65% | konserwowane w pierwszych ~ 900 aa |
| SOGA1 izoforma 1 | NP_542194.2 | 1661 | 41% | konserwowane na całej długości sekwencji z wyjątkiem ~ 950-1150 |
ortologi
U eukariontów zidentyfikowano wiele ortologów.
| Nazwa zwyczajowa | nazwa białka | rozbieżność z linii ludzkiej (MYA) | numer dostępowy | długość sekwencji (aa) | identyczność sekwencji z ludzkim białkiem | różnice w domenach białkowych |
|---|---|---|---|---|---|---|
| goryl | białko SOGA2 | 8.8 | XP_004059220.1 | 1586 | 99% | |
| pawian | białko SOGA2 | 29 | XP_003914218 | 1587 | 98% | |
| galago | białko SOGA3 | 74 | XP_003801047.1 | 1583 | 88% | Brak DUF4201 |
| szczur | CCDC165 | 92,3 | XP_237548.6 | 2060 | 81% | Brak DUF4201 |
| mysz | SOGA2 | 92,3 | NP_001107570.1 | 1893 | 80% | |
| kot domowy | białko SOGA2 | 94,2 | XP_003995077.1 | 1700 | 84% | Brak DUF4201 |
| krowa | CCDC166 | 94,2 | XP_581047.5 | 1525 | 74% | Brak DUF4201 |
| Słoń afrykański | CCDC167-podobny | 98,7 | XP_003406836.1 | 1544 | 73% | |
| zebry Finch | białko SOGA2 | 296 | XP_002193121.1 | 1598 | 69% | Brak DUF4201 |
| Ptactwo Czerwonej Dżungli | CCDC165 | 296 | XP_423729.3 | 1600 | 70% | Brak DUF4201 |
| Anol Karolina | niescharakteryzowane białko podobne do KIAA0802 | 296 | XP_003225723.1 | 1839 | 67% | Brak DUF4201 |
Odległe homologi
| Nazwa zwyczajowa | nazwa białka | rozbieżność z linii ludzkiej (MYA) | numer dostępowy | długość sekwencji (aa) | identyczność sekwencji z ludzkim białkiem | różnice w domenach białkowych |
|---|---|---|---|---|---|---|
| Tropikalna żaba szponiasta | niescharakteryzowane białko podobne do C20orf117 | 371,2 | XP_002942331.1 | 1584 | 39% | |
| fioletowy jeżowiec | niescharakteryzowane białko LOC578090 | 742,9 | XP_783370.2 | 1587 | 47% | Brak DUF4201 |
| wszy ciała | Centromerowe białko E, domniemane | 782,7 | XP_002429877.1 | 2086 | 30% | brak współdzielonych domen |
| południowy komar domowy | hipotetyczne białko konserwowane | 782,7 | XP_001843754.1 | 1878 | 32% | brak współdzielonych domen |
| wieprzowina | białko z rodziny powtórzeń antygenu powierzchniowego | 937,5 | XP_003380263.1 | 2030 | 36% | brak współdzielonych domen |
Domeny homologiczne
SOGA2 jest konserwowana najdalej wstecz w swoim regionie N-końcowym, gdzie zawiera trzy domeny o nieznanej funkcji.

Białko
Skład wewnętrzny białka
SOGA2 jest bogaty w glicynę (stosunek składu SOGA2 do przeciętnego białka ludzkiego wynosi 1,723), glutaminian (r = 1,647) i argininę (r = 1,357). Ma również niższy niż zwykle skład tyrozyny (r = 0,3406), izoleucyny (r = 0,4430), fenyloalaniny (r = 0,5808) i waliny (r = 0,6161).
Struktura pierwotna i izoformy
SOGA2 ma 4 izoformy: Q9Y4B5-1, Q9Y4B5-2, Q9Y4B5-3, Q9Y4B5-4.
Domeny i motywy
SOGA2 zawiera domenę nieznanej funkcji 4201 (DUF4201) z aa 16-235. Ta domena jest specyficzna dla rodziny białek zawierających domenę zwiniętej cewki u eukariontów. Zawiera również dwie kopie Domain of Unknown Function 3166 (DUF3166): jedną z aa 140-235 i jedną z aa 269-364.
Modyfikacje potranslacyjne
Oczekuje się, że SOGA2 przejdzie szereg modyfikacji potranslacyjnych. Modyfikacje ludzkiego SOGA2, które są wspólne dla ortologów, obejmują:
- Sumoilacja przy aminokwasach 87, 152, 235, 392 i 1379.
- Sulfinowanie przy tyrozynach 14 i 1249.
- Fosforylacja w wielu miejscach, zaznaczona na poniższej grafice:
Struktura drugorzędowa
Konsensus oprogramowania prognostycznego PELE, GOR4 i SOSUICoil jest taki, że drugorzędowa struktura SOGA2 jest zdominowana przez helisy alfa z przeplatanymi obszarami zwoju losowego. GOR4 wskazał, że SOGA2 jest zdominowany przez alfa-helisy; przewidział zaledwie 5,61% reszt w konformacji wydłużonej nici (równoległej lub antyrównoległej arkusza Beta), w przeciwieństwie do 47,79% helisy alfa i 46,6% zwojów losowych.
Struktura trzeciorzędowa
SOGA2 ma wspólne cechy sekwencji w swoim wysoce konserwatywnym regionie N-końcowym. Ta homologia umożliwia przewidywanie jego trzeciorzędowej struktury na podstawie homologii do opublikowanych struktur 3D za pośrednictwem struktury Phyre2 i NCBI.
Ekspresja genu
Promotor
Poniżej przedstawiono promotor ludzkiego SOGA2.
Dane dotyczące ekspresji genów
Profil EST pokazuje, że u ludzi SOGA2 ulega silnej ekspresji w wielu miejscach w całym ciele, w tym w kościach, mózgu, uchu, oku i wielu innych. Istnieje duża liczba transkryptów w próbkach raka wątroby. Dane z ludzkich mikromacierzy pokazują, że ekspresja SOGA2 jest umiarkowana, ze szczególnie wysoką ekspresją w mózgu (szczególnie w móżdżku i hipokampie), okrężnicy, przysadce mózgowej, jelicie cienkim, rdzeniu kręgowym, jądrach i mózgu płodu . Dane mikromacierzy specyficzne dla tkanki mózgowej pokazują, że SOGA2 ma wysoką ekspresję w tylnym płacie półkul móżdżku i tylnym płacie robaka w mózgu myszy . W większości innych obszarów mózgu ekspresja jest niska.
Warianty transkrypcji
U ludzi gen SOGA2 wytwarza 17 różnych transkryptów, z których 8 tworzy produkt białkowy (jeden ulega rozpadowi, w którym pośredniczy nonsens). Głównym transkryptem u ludzi jest transkrypt ID ENST00000359865 lub SOGA2-001.
Funkcjonować
Możliwe czynniki transkrypcyjne
Możliwe czynniki transkrypcyjne dla ludzkiego SOGA2 obejmują:
- Współczynnik rozpoznawania modulatora 2
- Białko wiążące element reagujący na cAMP 1
- alternatywny wariant splicingu FOXP1
- Gen podobny do MDS1/EVI1 1
- Ikaros 2, możliwy regulator różnicowania limfocytów
Interakcje
z koimmunoprecypitacją kompleksu białkowego ( Co-IP ) ujawniły białka oddziałujące, takie jak regulatory śmierci komórkowej , transportery kasety wiążącej ATP (ABC) i białka wiążące kinazę białkową A.
540 oddziałujących białek obejmuje ABCF1 , ACTB , ACTL6A , BCLAF1 , BCLAF1 , CHEK1 i MAGEE2.
Analiza K-najbliższego sąsiada przeprowadzona przez wilka pSort wskazuje, że u ludzi SOGA2 skupia się głównie w jądrze, cytoplazmie i przestrzeni cytojądrowej. Istnieje niewielka szansa, że lokalizuje się w aparacie Golgiego.
Za pośrednictwem bazy danych STRING zidentyfikowano również szereg interakcji białkowych, w tym MARK2 , MARK4 i PPP2R2B .
Znaczenie kliniczne
SOGA2 nie ma obecnie znanych skojarzeń ani mutacji chorobowych.
Dalsza lektura
- Strausberg RL, Feingold EA, Grouse LH i in. (2003). „Generowanie i wstępna analiza ponad 15 000 pełnej długości sekwencji cDNA człowieka i myszy” . proc. Natl. Acad. nauka USA . 99 (26): 16899–903. Bibcode : 2002PNAS...9916899M . doi : 10.1073/pnas.242603899 . PMC 139241 . PMID 12477932 .
- Brajenovic M, Joberty G, Küster B i in. (2004). „Kompleksowa analiza proteomiczna kompleksów ludzkich białek Par ujawnia wzajemnie połączoną sieć białek” . J. Biol. chemia . 279 (13): 12804-11. doi : 10.1074/jbc.M312171200 . PMID 14676191 .
- Ota T, Suzuki Y, Nishikawa T i in. (2004). „Kompletne sekwencjonowanie i charakterystyka 21 243 pełnej długości ludzkich cDNA” . Nat. Genet . 36 (1): 40–5. doi : 10.1038/ng1285 . PMID 14702039 .
- Gerhard DS, Wagner L, Feingold EA i in. (2004). „Stan, jakość i ekspansja projektu cDNA pełnej długości NIH: kolekcja genów ssaków (MGC)” . Genom Res . 14 (10B): 2121–7. doi : 10.1101/gr.2596504 . PMC 528928 . PMID 15489334 .
- Nusbaum C, Zody MC, Borowsky ML i in. (2005). „Sekwencja DNA i analiza ludzkiego chromosomu 18” . Natura . 437 (7058): 551–5. Bibcode : 2005Natur.437..551N . doi : 10.1038/natura03983 . PMID 16177791 .
- Nousiainen M, Silljé HH, Sauer G i in. (2006). „Analiza fosfoproteomu ludzkiego wrzeciona mitotycznego” . proc. Natl. Acad. nauka USA . 103 (14): 5391–6. Bibcode : 2006PNAS..103.5391N . doi : 10.1073/pnas.0507066103 . PMC 1459365 . PMID 16565220 .
- Beausoleil SA, Villén J, Gerber SA i in. (2006). „Oparte na prawdopodobieństwie podejście do wysokowydajnej analizy fosforylacji białek i lokalizacji miejsca”. Nat. Biotechnologia . 24 (10): 1285–92. doi : 10.1038/nbt1240 . PMID 16964243 . S2CID 14294292 .
- Olsen JV, Blagoev B, Gnad F i in. (2006). „Globalna, in vivo i specyficzna dla miejsca dynamika fosforylacji w sieciach sygnalizacyjnych” . komórka . 127 (3): 635–48. doi : 10.1016/j.cell.2006.09.026 . PMID 17081983 . S2CID 7827573 .