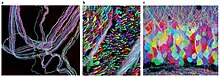
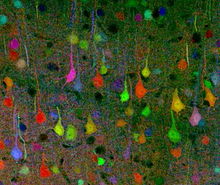
Brainbow
Brainbow to proces, dzięki któremu poszczególne neurony w mózgu można odróżnić od sąsiednich neuronów za pomocą białek fluorescencyjnych. Poprzez losowe wyrażanie różnych proporcji czerwonych, zielonych i niebieskich pochodnych zielonego białka fluorescencyjnego w poszczególnych neuronach, możliwe jest oznaczenie każdego neuronu charakterystycznym kolorem. Proces ten wniósł znaczący wkład w dziedzinę konektomiki neuronowej .
Technika ta została pierwotnie opracowana w 2007 roku przez zespół kierowany przez Jeffa W. Lichtmana i Joshuę R. Sanesa z Uniwersytetu Harvarda . Oryginalna technika została ostatnio dostosowana do użytku z innymi modelowymi organizmami badawczymi, w tym muszką owocową ( Drosophila melanogaster ) , danio pręgowanym ( Danio rerio ) i Arabidopsis thaliana .
Podczas gdy wcześniejsze techniki znakowania pozwalały na mapowanie tylko kilku neuronów, ta nowa metoda pozwala na jednoczesne i zróżnicowane oświetlenie ponad 100 różnie mapowanych neuronów w ten sposób. Prowadzi to do jego charakterystycznego, wielobarwnego wyglądu na obrazach, zyskując swoją nazwę i zdobywając nagrody w konkursach fotografii naukowej. [ potrzebne źródło ]
Historia i rozwój
Brainbow został początkowo opracowany przez Jeffa W. Lichtmana i Joshuę R. Sanesa na Uniwersytecie Waszyngtońskim w St. Louis , chociaż od tego czasu przenieśli się na Uniwersytet Harvarda . Zespół skonstruował Brainbow przy użyciu dwuetapowego procesu: najpierw wygenerowano specyficzny konstrukt genetyczny , który można było ponownie połączyć w wielu układach, aby uzyskać jeden z trzech lub czterech kolorów w oparciu o konkretne wdrażane białka fluorescencyjne (XFP). Następnie wiele kopii tego samego transgenicznego konstruktu wstawiono do genomu gatunku docelowego, co spowodowało losową ekspresję różnych proporcji XFP, a następnie spowodowało, że różne komórki wykazywały różnorodne kolorowe odcienie.
Brainbow został pierwotnie stworzony jako ulepszenie bardziej tradycyjnych technik neuroobrazowania , takich jak barwienie Golgiego i wstrzykiwanie barwnika, z których oba stanowiły poważne ograniczenia dla naukowców w ich zdolności do wizualizacji skomplikowanej architektury obwodów nerwowych w mózgu . Podczas gdy starsze techniki były w stanie zabarwić komórki jedynie wąskim zakresem kolorów, często wykorzystując transgeniczne myszy dwu- i trójkolorowe do ujawnienia ograniczonych informacji dotyczących struktur neuronalnych, Brainbow jest znacznie bardziej elastyczny, ponieważ ma zdolność znakowania fluorescencyjnego pojedyncze neurony o około 100 różnych odcieniach, dzięki czemu naukowcy mogą zidentyfikować, a nawet rozróżnić procesy dendrytyczne i aksonalne . Ujawniając tak szczegółowe informacje na temat połączeń i wzorców neuronalnych, czasami nawet in vivo, naukowcy często są w stanie wywnioskować informacje dotyczące interakcji neuronalnych i ich późniejszego wpływu na zachowanie i funkcjonowanie. W ten sposób Brainbow wypełnił pustkę pozostawioną przez poprzednie metody neuroobrazowania.
Wraz z niedawnym pojawieniem się Brainbow w neurobiologii naukowcy są teraz w stanie konstruować specyficzne mapy obwodów neuronalnych i lepiej badać, w jaki sposób odnoszą się one do różnych czynności umysłowych i powiązanych z nimi zachowań (np. Brainbow ujawnia informacje o wzajemnych połączeniach między neuronami i ich późniejszych interakcjach, które wpływają ogólna funkcjonalność mózgu). Jako dalszą ekstrapolację tej metody, Brainbow może być zatem wykorzystany do badania zarówno zaburzeń neurologicznych, jak i psychicznych poprzez analizę różnic w mapach neuronowych.
Metody
Techniki Brainbow opierają się na rekombinacji Cre-Lox , w której rekombinaza białkowa Cre steruje inwersją lub wycięciem DNA między miejscami loxP. Oryginalna metoda Brainbow obejmuje zarówno Brainbow-1, jak i Brainbow-2, które wykorzystują różne formy rekombinacji cre/lox. Brainbow-3, zmodyfikowana wersja Brainbow-1, została opracowana w 2013 roku. Dla wszystkich podtypów Brainbow ekspresja danego XFP jest zdarzeniem stochastycznym lub losowym.
Brainbow-1 wykorzystuje konstrukty DNA z różnymi genami białek fluorescencyjnych (XFP) oddzielonymi zmutowanymi i kanonicznymi formami loxP. Stwarza to zestaw wzajemnie wykluczających się możliwości wycinania, ponieważ rekombinacja za pośrednictwem cre zachodzi tylko między identycznymi miejscami loxP. Po wystąpieniu rekombinacji białko fluorescencyjne, które pozostaje bezpośrednio po promotorze, ulega unikalnej ekspresji. Zatem konstrukt z czterema XFP oddzielonymi trzema różnymi miejscami loxP, trzema zdarzeniami wycięcia i oryginalnym konstruktem może wytwarzać cztery różne białka fluorescencyjne.
Brainbow-2 wykorzystuje wycięcie i inwersję Cre, aby umożliwić wiele możliwości ekspresji w danym konstrukcie. W jednym segmencie DNA z dwoma przeciwnie zorientowanymi XFP, Cre wywoła losowe zdarzenie inwersji, które pozostawia jedno białko fluorescencyjne we właściwej orientacji do ekspresji. Jeśli dwie z tych odwracalnych sekwencji są wyrównane, możliwe są trzy różne zdarzenia inwersji. Gdy brane są również pod uwagę zdarzenia wycinania, jedno z czterech białek fluorescencyjnych będzie wyrażane dla danej kombinacji wycięć i inwersji Cre.
Brainbow-3 zachowuje format Brainbow-1 loxP, ale zastępuje geny RFP, YFP i CFP mOrange2, EGFP i mKate2. Wybrano mO2, EGFP i mK2, ponieważ ich widma wzbudzenia i emisji fluorescencji zachodzą na siebie w minimalnym stopniu, a także dlatego, że mają minimalną homologię sekwencji, co pozwala na zaprojektowanie selektywnych przeciwciał, które można wykorzystać do ich wykrywania w protokołach immunohistochemicznych . Brainbow-3 rozwiązuje również problem nierównomiernego wypełniania neuronów XFP za pomocą farnezylowanych pochodnych XFP, które są bardziej równomiernie przemieszczane do błon neuronalnych.
Brainbow jest wdrażany in vivo przez skrzyżowanie dwóch szczepów organizmów transgenicznych : jednego, który wyraża białko Cre, i drugiego, który został transfekowany kilkoma wersjami konstruktu loxP/XFP. Korzystanie z wielu kopii transgenu pozwala XFP łączyć się w sposób, który może dać jeden z około 100 różnych kolorów. W ten sposób każdy neuron jest oznaczony innym odcieniem w oparciu o daną kombinatoryczną i stochastyczną ekspresję białek fluorescencyjnych.
W celu wyjaśnienia zróżnicowanych wzorców ekspresji XFP w widocznej formie, skrawki mózgu są obrazowane za pomocą mikroskopii konfokalnej . Po wystawieniu na działanie fotonu o określonej długości fali wzbudzenia, każdy fluorofor emituje sygnał, który jest zbierany w kanale czerwonym, zielonym lub niebieskim, a wynikowa kombinacja światła jest analizowana za pomocą oprogramowania do analizy danych. Nałożenie różnokolorowych neuronów umożliwia wizualne rozplątanie skomplikowanych obwodów neuronowych.
Brainbow był dotychczas testowany głównie na myszach; jednak podstawowa technika opisana powyżej została również zmodyfikowana do użytku w nowszych badaniach od czasu pojawienia się oryginalnej metody wprowadzonej w 2007 roku.
myszy

Mózg myszy ma 75 000 000 neuronów i jest bardziej podobny do ludzkiego mózgu niż drosophila i inne organizmy powszechnie używane do modelowania tej techniki, takie jak C. elegans . Myszy były pierwszymi organizmami, u których z powodzeniem zastosowano metodę neuroobrazowania Brainbow. Livet i in. (2007) opracowali dwie wersje myszy Brainbow przy użyciu Brainbow-1 i Brainbow-2, które opisano powyżej. Używając tych metod do stworzenia kompletnej mapy i śledzenia aksonów mięśnia myszy, konieczne jest zebranie dziesiątek tysięcy obrazów i zestawienie ich w stosy, aby stworzyć kompletny schemat. Następnie możliwe jest prześledzenie każdego aksonu motorycznego i jego styków synaptycznych w celu skonstruowania kompletnego konektomu mięśnia.
Więcej przykładów neuronów zbadanych techniką Brainbow u transgenicznych myszy znajduje się w mięśniach ucha unerwiających nerw ruchowy, drogach aksonów w pniu mózgu i zakręcie zębatym hipokampa .
Drosophila
Złożoność mózgu Drosophila, składającego się z około 100 000 neuronów, czyni go doskonałym kandydatem do wdrożenia technik neurofizjologicznych i neurobiologicznych, takich jak Brainbow. W rzeczywistości Stefanie Hampel i in. (2011) połączyli Brainbow w połączeniu z narzędziami do celowania genetycznego w celu identyfikacji poszczególnych neuronów w mózgu Drosophila i różnych linii neuronalnych. Jednym z narzędzi do kierowania genetycznego był GAL4/UAS , który kontroluje ekspresję UAS-Brainbow i kieruje ekspresję do małych grup neuronów. Wykorzystanie metod „Flip Out” zwiększyło rozdzielczość komórkową konstruktu reporterowego. Ekspresja białek fluorescencyjnych, podobnie jak w przypadku oryginalnego Brainbow, zależała od rekombinacji Cre odpowiadającej dopasowanym miejscom lox. Hampel i in. (2011) opracowali również własną odmianę Brainbow (dBrainbow), opartą na znakowaniu przeciwciałami epitopów, a nie endogennej fluorescencji. Dwie kopie ich konstruktu dają sześć jasnych, dających się rozdzielić kolorów. To, wraz z uproszczeniami w przypisywaniu kolorów, umożliwiło im obserwację trajektorii każdego neuronu na duże odległości. W szczególności prześledzili neurony ruchowe od płata anteny do połączeń nerwowo-mięśniowych, co pozwoliło im zidentyfikować określone cele mięśniowe poszczególnych neuronów.
Ostatecznie technika ta zapewnia możliwość skutecznego mapowania obwodów neuronalnych u Drosophila, dzięki czemu naukowcy mogą odkryć więcej informacji na temat struktury mózgu tego bezkręgowca i jego związku z wynikającym z tego zachowaniem.
Ograniczenia
Jak każda technika neuroobrazowania , Brainbow ma szereg ograniczeń, które wynikają z metod wymaganych do jej wykonania. Na przykład proces hodowli co najmniej dwóch szczepów zwierząt transgenicznych z embrionalnych komórek macierzystych jest zarówno czasochłonny, jak i złożony. Nawet jeśli uda się stworzyć dwa transgeniczne , nie wszystkie ich potomstwo wykaże rekombinację. Dlatego wymaga to szeroko zakrojonego planowania przed wykonaniem eksperymentu.
Ponadto, ze względu na losowy charakter ekspresji białek fluorescencyjnych, naukowcy nie są w stanie precyzyjnie kontrolować znakowania obwodów nerwowych, co może skutkować słabą identyfikacją określonych neuronów.
Wykorzystanie łuku mózgowego w populacjach ssaków jest również utrudnione przez niesamowitą różnorodność neuronów ośrodkowego układu nerwowego . Sama gęstość neuronów w połączeniu z obecnością długich ciągów aksonów utrudnia oglądanie większych obszarów OUN w wysokiej rozdzielczości. Brainbow jest najbardziej przydatny podczas badania rozdzielczości pojedynczej komórki na tle złożonego środowiska wielokomórkowego. Jednak ze względu na ograniczenia rozdzielczości mikroskopii optycznej ostateczna identyfikacja połączeń synaptycznych między neuronami nie jest łatwa. Problemu tego można w pewnym stopniu uniknąć, stosując markery synaptyczne jako uzupełnienie mikroskopii optycznej do oglądania połączeń synaptycznych.