Cis-naturalny antysensowny transkrypt
Naturalne transkrypty antysensowne (NAT) to grupa RNA zakodowanych w komórce, które mają komplementarność transkryptu z innymi transkryptami RNA. Zidentyfikowano je u wielu eukariotów , w tym ludzi, myszy, drożdży i Arabidopsis thaliana . Ta klasa RNA obejmuje zarówno RNA kodujące, jak i niekodujące białka. Obecne dowody sugerują różne role regulacyjne dla NAT, takie jak interferencja RNA (RNAi), alternatywny splicing , imprinting genomowy i inaktywacja chromosomu X. NAT są ogólnie podzielone na dwie kategorie w zależności od tego, czy działają w systemie cis, czy trans. Trans-NAT są transkrybowane z innego miejsca niż ich cele i zwykle mają komplementarność z wieloma transkryptami z pewnymi niedopasowaniami. MikroRNA (miRNA) są przykładem trans-NAT, które mogą celować w wiele transkryptów z kilkoma niedopasowaniami. Cis-naturalne transkrypty antysensowne ( cis-NAT ) z drugiej strony są transkrybowane z tego samego locus genomowego , co ich cel, ale z przeciwnej nici DNA i tworzą idealne pary.
Orientacja
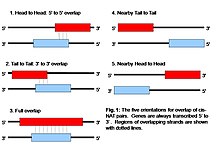
Cis-NAT mają różne orientacje i różne długości nakładania się par. Do tej pory zidentyfikowano pięć orientacji dla cis-NAT. Najczęstszą orientacją jest głowa do głowy, gdzie końce 5' obu transkryptów są wyrównane. Ta orientacja skutkowałaby największym powaleniem ekspresji genów, gdyby kolizja transkrypcyjna była przyczyną hamowania transkryptu. Istnieją jednak badania, które sugerują, że orientacje od ogona do ogona są najczęstszymi parami NAT. Inne, takie jak ogon do ogona, zachodzenie na siebie, blisko głowa do głowy i blisko ogon do ogona, są rzadziej spotykane. Całkowicie nakładające się NAT obejmują gen antysensowny, który jest umieszczony całkowicie jeden nad drugim. Pobliskie orientacje głowa do głowy i ogon do ogona są fizycznie oddzielone od siebie, ale znajdują się bardzo blisko siebie. Obecne dowody sugerują, że istnieje nadreprezentacja par NAT w genach, które mają aktywność katalityczną. W szczególności w tych genach może być coś, co czyni je bardziej podatnymi na tego typu regulacje.
Podejście identyfikacyjne
Identyfikacja NAT w całych genomach jest możliwa dzięki dużemu zbiorowi danych sekwencyjnych dostępnych z wielu organizmów. Metody in silico do wykrywania NAT mają kilka niedociągnięć w zależności od źródła informacji o sekwencji. Badania wykorzystujące mRNA mają sekwencje, których orientacja jest znana, ale ilość dostępnych informacji o sekwencji mRNA jest niewielka. Przewidywane modele genów wykorzystujące algorytmy przeszkolone do wyszukiwania genów dają większe pokrycie genomu kosztem zaufania do zidentyfikowanego genu. Innym zasobem są obszerne znaczników wyrażonej sekwencji (EST), ale tym małym sekwencjom należy najpierw przypisać orientację, zanim będzie można z nich wydobyć przydatne informacje. W niektórych badaniach wykorzystano specjalne informacje o sekwencji w EST, takie jak sygnał poli (A), ogon poli (A) i miejsca splicingu, aby zarówno filtrować EST, jak i nadać im prawidłową orientację transkrypcji. Kombinacje różnych źródeł sekwencji próbują zmaksymalizować pokrycie, a także zachować integralność danych.
Pary NAT są identyfikowane, gdy tworzą nakładające się klastry. Istnieje zmienność wartości odcięcia stosowanych w różnych badaniach, ale generalnie uważa się, że za minimum dla transkryptów i nakładających się klastrów uważa się około 20 nukleotydów nakładających się sekwencji. Ponadto transkrypty muszą być mapowane tylko na jedną inną cząsteczkę mRNA, aby można ją było uznać za parę NAT. Obecnie istnieje wiele zasobów internetowych i oprogramowania, które można wykorzystać do wyszukiwania par antysensownych. Baza danych NATsdb lub Natural Antisense Transcript to bogate narzędzie do wyszukiwania par antysensownych z wielu organizmów.
Mechanizmy
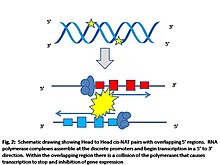
Mechanizmy molekularne stojące za regulacyjną rolą cis-NAT nie są obecnie dobrze poznane. Zaproponowano trzy modele wyjaśniające wpływ regulacyjny cis-NAT na ekspresję genów. Pierwszy model wskazuje, że parowanie zasad między cis-NAT i jego komplementarnym transkryptem powoduje obniżenie ekspresji mRNA. Założeniem tego modelu jest dokładne dopasowanie co najmniej 6 par zasad między parą cis-NAT w celu wytworzenia dwuniciowego RNA. Podstawą drugiego modelu są modyfikacje epigenetyczne, takie jak metylacja DNA i posttranslacyjna modyfikacja histonów rdzeniowych. Chociaż nie jest to jeszcze jasno zrozumiane, uważa się, że odwrotny transkrypt prowadzi kompleksy metylacyjne i/lub kompleksy modyfikujące histony do regionów promotorowych sensownego transkryptu i powoduje hamowanie ekspresji z genu. Obecnie nie wiadomo, jakie cechy cis-NAT są kluczowe dla epigenetycznego modelu regulacji. Ostatnim proponowanym modelem, który zyskał uznanie dzięki niedawnym dowodom eksperymentalnym, jest model kolizji transkrypcyjnej. Podczas procesu transkrypcji cis-NAT kompleksy transkrypcyjne gromadzą się w regionach promotorowych genu. Polimerazy RNA rozpoczną następnie transkrypcję genu w miejscu inicjacji transkrypcji, układając nukleotydy w kierunku od 5' do 3'. W obszarach nakładania się cis-NAT polimerazy RNA zderzają się i zatrzymują w miejscu katastrofy. Transkrypcja jest hamowana, ponieważ polimerazy RNA przedwcześnie zatrzymują się, a ich niekompletne transkrypty ulegają degradacji.
Znaczenie
Regulacja wielu procesów biologicznych, takich jak rozwój, metabolizm i wiele innych, wymaga starannej koordynacji między wieloma różnymi genami; jest to zwykle określane jako sieć regulacji genów . Fala zainteresowania sieciami regulacyjnymi genów została wywołana pojawieniem się zsekwencjonowanych genomów wielu organizmów. Następnym krokiem jest wykorzystanie tych informacji do ustalenia, w jaki sposób geny współpracują ze sobą, a nie tylko w izolacji. Podczas procesów rozwojowych ssaków dochodzi do inaktywacji dodatkowego chromosomu X u samic. Wykazano, że para NAT zwana Xist i Tsix bierze udział w hipermetylacji chromosomu. Wykazano, że aż 20–30% genów ssaków jest celem miRNA , co podkreśla znaczenie tych cząsteczek jako regulatorów w wielu genach. Ewolucyjne powody wykorzystania RNA do regulacji genów mogą polegać na tym, że jest to mniej kosztowne i szybsze niż synteza białek niepotrzebnych komórce. Mogło to mieć selektywną przewagę dla wczesnych eukariontów z tego typu regulacją transkrypcji.
Choroba

Transkrypcja antysensowna może przyczyniać się do choroby poprzez zmiany chromosomalne, które skutkują wytwarzaniem nieprawidłowych transkryptów antysensownych. Udokumentowany przypadek udziału cis-NAT w chorobie człowieka pochodzi z dziedzicznej postaci α- talasemii , w której dochodzi do wyciszenia genu hemoglobiny α-2 poprzez działanie cis-NAT. Uważa się, że w złośliwych komórkach nowotworowych z aktywowanymi elementami transpozycyjnymi tworzy się duża ilość szumu transkrypcyjnego. Jest prawdopodobne, że nieprawidłowe transkrypty antysensownego RNA wynikające z tego szumu transkrypcyjnego mogą powodować stochastyczną metylację wysp CpG związanych z onkogenami i genami supresorowymi nowotworów . To hamowanie jeszcze bardziej pogłębiłoby złośliwość komórek, ponieważ tracą one kluczowe geny regulatorowe. Przyglądając się regulowanym w górę transkryptom antysensownym w komórkach nowotworowych, naukowcy są w stanie szukać większej liczby potencjalnych genów supresorowych nowotworów. Również nieprawidłowe cis-NAT są zaangażowane w choroby neurologiczne, takie jak choroba Parkinsona .