Przenieś RNA
| Identyfikatory | |
|---|---|
| tRNA | |
| Symbol | T |
| Rfam | RF00005 |
| Inne dane | |
| typ RNA | gen , tRNA |
| Struktury PDB | PDBe 3icq, 1asy, 1asz, 1il2, 2tra, 3tra, 486d, 1fir, 1yfg, 3eph, 3epj, 3epk, 3epl, 1efw, 1c0a, 2ake, 2azx, 2dr2, 1f7u, 1f7v, 3foz, 2hgp, 2j00, 2j02, 2ow8 , 2v46, 2v48, 2wdg, 2wdh, 2wdk, 2wdm, 2wh1 |
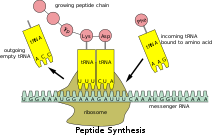
Transfer RNA (w skrócie tRNA i wcześniej określany jako sRNA , dla rozpuszczalnego RNA ) to cząsteczka adaptorowa złożona z RNA , zazwyczaj o długości od 76 do 90 nukleotydów (u eukariontów), która służy jako fizyczne połączenie między mRNA a aminokwasem sekwencja białek. Geny tRNA z bakterii są zazwyczaj krótsze (średnia = 77,6 pz) niż tRNA z Archaea (średnia = 83,1 pz) i eukariontów (średnia = 84,7 pz). Dojrzały tRNA ma odwrotny wzór, przy czym tRNA z bakterii jest zwykle dłuższy (mediana = 77,6 nt) niż tRNA z Archaea (mediana = 76,8 nt), przy czym eukarionty wykazują najkrótsze dojrzałe tRNA (mediana = 74,5 nt). Transfer RNA (tRNA) dokonuje tego, przenosząc aminokwas do mechanizmu syntezy białka w komórce zwanej rybosomem . Komplementacja 3-nukleotydowego kodonu w informacyjnym RNA (mRNA) przez 3-nukleotydowy antykodon tRNA powoduje syntezę białek opartą na kodzie mRNA. Jako takie, tRNA są niezbędnym składnikiem translacji , biologicznej syntezy nowych białek zgodnie z kodem genetycznym .
Przegląd
Podczas gdy specyficzna sekwencja nukleotydowa mRNA określa, które aminokwasy są włączone do produktu białkowego genu, z którego transkrybowany jest mRNA, rolą tRNA jest określenie, która sekwencja z kodu genetycznego odpowiada któremu aminokwasowi. MRNA koduje białko jako serię sąsiadujących ze sobą kodonów, z których każdy jest rozpoznawany przez określony tRNA. Jeden koniec tRNA pasuje do kodu genetycznego w sekwencji trzech nukleotydów zwanej antykodonem . Antykodon tworzy z kodonem trzy komplementarne pary zasad w mRNA podczas biosyntezy białek.
Na drugim końcu tRNA znajduje się kowalencyjne przyłączenie do aminokwasu odpowiadającego sekwencji antykodonu. Każdy typ cząsteczki tRNA może być przyłączony tylko do jednego rodzaju aminokwasu, więc każdy organizm ma wiele typów tRNA. Ponieważ kod genetyczny zawiera wiele kodonów, które określają ten sam aminokwas, istnieje kilka cząsteczek tRNA niosących różne antykodony, które niosą ten sam aminokwas.
Przyłączenie kowalencyjne do końca 3' tRNA jest katalizowane przez enzymy zwane syntetazami aminoacylo-tRNA . Podczas syntezy białek tRNA z dołączonymi aminokwasami są dostarczane do rybosomu przez białka zwane czynnikami elongacyjnymi , które pomagają w asocjacji tRNA z rybosomem, syntezie nowego polipeptydu i translokacji (ruchu) rybosomu wzdłuż mRNA. Jeśli antykodon tRNA pasuje do mRNA, inny tRNA już związany z rybosomem przenosi rosnący łańcuch polipeptydowy z jego końca 3' na aminokwas przyłączony do końca 3' nowo dostarczonego tRNA, reakcja jest katalizowana przez rybosom. Może to być duża liczba pojedynczych nukleotydów w cząsteczce tRNA modyfikowane chemicznie , często przez metylację lub deamidację . Te niezwykłe zasady czasami wpływają na interakcję tRNA z rybosomami , a czasami występują w antykodonie , zmieniając właściwości parowania zasad.
Struktura
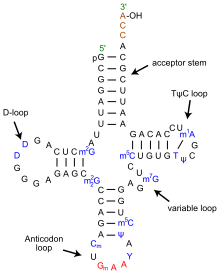
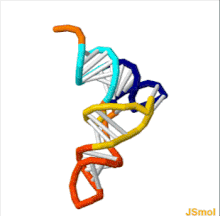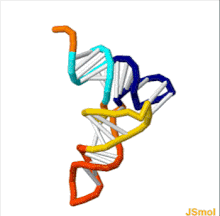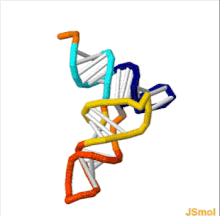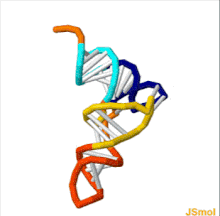
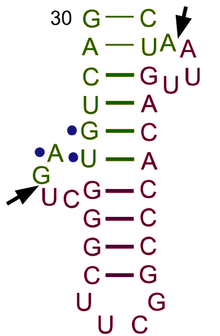
Strukturę tRNA można rozłożyć na strukturę pierwszorzędową , drugorzędową (zwykle wizualizowaną jako struktura koniczyny ) i trzeciorzędową (wszystkie tRNA mają podobną trójwymiarową strukturę w kształcie litery L, która pozwala im dopasować się do miejsc P i A ). rybosomu ) . Struktura koniczyny staje się trójwymiarową strukturą w kształcie litery L poprzez współosiowe ułożenie helis, co jest powszechną trzeciorzędową strukturą RNA motyw. Długość każdego ramienia, a także „średnica” pętli w cząsteczce tRNA różnią się w zależności od gatunku. Struktura tRNA składa się z następujących elementów:
- Rdzeń akceptorowy to rdzeń o długości od 7 do 9 par zasad (bp) utworzony przez sparowanie zasad 5'-końcowego nukleotydu z 3'-końcowym nukleotydem (który zawiera 3'-końcową grupę CCA używaną do przyłączenia amino kwas). Ogólnie rzecz biorąc, takie 3'-końcowe struktury podobne do tRNA są określane jako „znaczniki genomowe”. Rdzeń akceptorowy może zawierać pary zasad innych niż Watson-Crick.
- Ogon CCA jest sekwencją cytozyna -cytozyna- adenina na końcu 3' cząsteczki tRNA. Aminokwas załadowany na tRNA przez syntetazy aminoacylo-tRNA , tworząc aminoacylo-tRNA , jest kowalencyjnie związany z grupą 3'-hydroksylową na ogonie CCA. Ta sekwencja jest ważna dla rozpoznawania tRNA przez enzymy i krytyczna w translacji. U prokariotów sekwencja CCA jest transkrybowana w niektórych sekwencjach tRNA. W większości prokariotycznych tRNA i eukariotycznych tRNA sekwencja CCA jest dodawana podczas przetwarzania i dlatego nie pojawia się w genie tRNA.
- Pętla D to łodyga o długości od 4 do 6 pz zakończona pętlą, która często zawiera dihydrourydynę .
- Pętla antykodonowa to łodyga o długości 5 pz, której pętla zawiera antykodon . Pierwszorzędowa struktura tRNA 5'-do-3' zawiera antykodon, ale w odwrotnej kolejności, ponieważ kierunkowość 3'-do-5' jest wymagana do odczytania mRNA od 5'-do-3'.
- Pętla ΨU została tak nazwana ze względu na charakterystyczną obecność w pętli niezwykłej zasady ΨU, gdzie Ψ to pseudourydyna , zmodyfikowana urydyna . Zmodyfikowana zasada często znajduje się w sekwencji 5'-TΨUCG-3'.
- Pętla zmienna znajduje się pomiędzy pętlą antykodonową a pętlą ΨU i, jak sama nazwa wskazuje, ma różną wielkość od 3 do 21 zasad.
antykodon
Antykodon to jednostka trzech nukleotydów odpowiadająca trzem zasadom kodonu mRNA . Każdy tRNA ma odrębną sekwencję trypletową antykodonu, która może tworzyć 3 komplementarne pary zasad do jednego lub więcej kodonów aminokwasu. Niektóre antykodony łączą się w pary z więcej niż jednym kodonem z powodu parowania zasad oscylacyjnych . Często pierwszym nukleotydem antykodonu jest nukleotyd, którego nie ma na mRNA: inozyna , która może tworzyć wiązania wodorowe z więcej niż jedną zasadą w odpowiedniej pozycji kodonu. W kodzie genetycznym , często zdarza się, że pojedynczy aminokwas jest określony przez wszystkie cztery możliwości na trzeciej pozycji lub przynajmniej przez obie pirymidyny i puryny ; na przykład aminokwas glicyna jest kodowany przez sekwencje kodonów GGU, GGC, GGA i GGG. Inne zmodyfikowane nukleotydy mogą również pojawiać się w pierwszej pozycji antykodonu - czasami nazywanej „pozycją wahania” - powodując subtelne zmiany w kodzie genetycznym, jak na przykład w mitochondriach . Na komórkę potrzeba 61 typów tRNA, aby zapewnić zgodność jeden do jednego między cząsteczkami tRNA a kodonami określającymi aminokwasy, ponieważ standardowy kod genetyczny ma 61 sensownych kodonów. Jednak wiele komórek ma mniej niż 61 typów tRNA, ponieważ wahliwa podstawa jest zdolna do wiązania się z kilkoma, choć niekoniecznie wszystkimi, kodonami, które określają konkretny aminokwas. Co najmniej 31 tRNA jest potrzebnych do jednoznacznej translacji wszystkich 61 kodonów sensownych.
Aminoacylacja
Aminoacylacja to proces dodawania grupy aminoacylowej do związku. Łączy kowalencyjnie aminokwas z końcem 3' CCA cząsteczki tRNA. Każdy tRNA jest aminoacylowany (lub ładowany ) określonym aminokwasem przez syntetazę aminoacylo-tRNA . Zwykle dla każdego aminokwasu jest pojedyncza syntetaza aminoacylo-tRNA, pomimo faktu, że może istnieć więcej niż jeden tRNA i więcej niż jeden antykodon dla aminokwasu. W rozpoznawaniu odpowiedniego tRNA przez syntetazy nie pośredniczy wyłącznie antykodon, a rdzeń akceptorowy często odgrywa znaczącą rolę. Reakcja:
W niektórych organizmach może brakować jednej lub więcej syntetaz aminofosforanowo-tRNA. Prowadzi to do ładowania tRNA przez chemicznie spokrewniony aminokwas, a przy użyciu enzymu lub enzymów tRNA jest modyfikowany, aby był prawidłowo naładowany. Na przykład Helicobacter pylori nie ma syntetazy glutaminylo-tRNA. Zatem syntetaza glutaminianu tRNA ładuje tRNA-glutaminę (tRNA-Gln) glutaminianem . Następnie amidotransferaza przekształca kwaśny łańcuch boczny glutaminianu w amid, tworząc prawidłowo naładowany gln-tRNA-Gln.
Ingerencja w aminoacylację może być przydatna jako podejście do leczenia niektórych chorób: komórki nowotworowe mogą być stosunkowo podatne na zaburzoną aminoacylację w porównaniu ze zdrowymi komórkami. Synteza białek związana z rakiem i biologią wirusów jest często bardzo zależna od określonych cząsteczek tRNA. Na przykład w przypadku raka wątroby ładowanie tRNA-Lys-CUU lizyną podtrzymuje wzrost komórek raka wątroby i przerzuty, podczas gdy zdrowe komórki mają znacznie mniejszą zależność od tego tRNA w celu wspierania fizjologii komórkowej. Podobnie, wirus zapalenia wątroby typu E wymaga krajobrazu tRNA, który zasadniczo różni się od krajobrazu związanego z niezakażonymi komórkami. Dlatego hamowanie aminoacylacji określonych rodzajów tRNA jest uważane za obiecującą nową drogę racjonalnego leczenia wielu chorób.
Wiązanie z rybosomem
Rybosom ma trzy miejsca wiązania cząsteczek tRNA, które rozciągają się między dwiema podjednostkami rybosomu : miejsca A (aminoacylowe) , P (peptydylowe) i E ( wyjściowe) . Ponadto rybosom ma dwa inne miejsca wiązania tRNA, które są wykorzystywane podczas mRNA lub podczas inicjacji syntezy białek . Są to miejsce T (nazywane współczynnikiem wydłużenia Tu ) i miejsce I (inicjacja). Zgodnie z konwencją miejsca wiązania tRNA są oznaczone miejscem na małej podjednostce rybosomu wymienione jako pierwsze, a miejsce na dużej podjednostce rybosomu wymienione jako drugie. Na przykład witryna A jest często zapisywana jako A/A, witryna P jako P/P, a witryna E jako E/E. Białka wiążące, takie jak L27, L2, L14, L15, L16 w miejscach A i P, zostały określone przez znakowanie powinowactwa przez AP Czernilofsky i in. ( Proc. Natl. Acad. Sci, USA , s. 230-234, 1974).
Po zakończeniu inicjacji translacji pierwszy aminoacylo-tRNA znajduje się w miejscu P/P i jest gotowy do opisanego poniżej cyklu wydłużania. Podczas elongacji translacji tRNA najpierw wiąże się z rybosomem jako część kompleksu z czynnikiem elongacyjnym Tu ( EF-Tu ) lub jego eukariotycznym ( eEF-1 ) lub archeologicznym odpowiednikiem. To początkowe miejsce wiązania tRNA nazywa się miejscem A/T. W miejscu A/T połowa miejsca A znajduje się w małej podjednostce rybosomu , w której znajduje się miejsce dekodowania mRNA. Miejsce dekodowania mRNA to miejsce, w którym kodon mRNA jest odczytywany podczas tłumaczenia. Połowa miejsca T znajduje się głównie na dużej podjednostce rybosomu , gdzie EF-Tu lub eEF-1 oddziałuje z rybosomem. Po zakończeniu dekodowania mRNA, aminoacylo-tRNA jest wiązane w miejscu A/A i jest gotowe do utworzenia kolejnego wiązania peptydowego z przyłączonym do niego aminokwasem. Peptydylo-tRNA, które przenosi rosnący polipeptyd do aminoacylo-tRNA związanego w miejscu A/A, jest związane w miejscu P/P. Po utworzeniu wiązania peptydowego tRNA w miejscu P/P jest acylowane lub ma wolny koniec 3' , a tRNA w miejscu A/A dysocjuje rosnący łańcuch polipeptydowy. Aby umożliwić następny cykl wydłużania, tRNA przemieszczają się następnie przez hybrydowe miejsca wiązania A/P i P/E, zanim zakończą cykl i zamieszkają w miejscach P/P i E/E. Gdy tRNA A/A i P/P przesunęły się do miejsc P/P i E/E, mRNA również przesunął się o jeden kodon, a miejsce A/T jest puste, gotowe do następnej rundy dekodowania mRNA. Następnie tRNA związany w miejscu E/E opuszcza rybosom.
Miejsce P/I jest właściwie pierwszym, które wiąże się z aminoacylo-tRNA, które jest dostarczane przez czynnik inicjujący zwany IF2 w bakteriach. Jednak istnienie miejsca P/I w rybosomach eukariotycznych lub archeologicznych nie zostało jeszcze potwierdzone. Białko L27 w miejscu P zostało określone przez znakowanie powinowactwa przez E. Collatza i AP Czernilofsky'ego ( FEBS Lett. , tom 63, str. 283-286, 1976).
geny tRNA
Organizmy różnią się liczbą genów tRNA w swoim genomie . Na przykład robak nicieni C. elegans , organizm modelowy powszechnie używany w badaniach genetycznych , ma 29 647 genów w swoim genomie jądrowym , z których 620 koduje tRNA. Pączkujące drożdże Saccharomyces cerevisiae mają w swoim genomie 275 genów tRNA. Liczba genów tRNA w genomie może się znacznie różnić, przy czym gatunki bakterii z grup takich jak Fusobacteria i Tenericutes mają około 30 genów w genomie, podczas gdy złożone genomy eukariotyczne, takie jak danio pręgowany ( Danio rerio ) może zawierać ponad 10 tysięcy genów tRNA.
W ludzkim genomie, który według szacunków ze stycznia 2013 roku zawiera łącznie około 20 848 genów kodujących białka, znajduje się 497 genów jądrowych kodujących cytoplazmatyczne cząsteczki tRNA i 324 pseudogeny pochodzące z tRNA — geny tRNA uważane za niefunkcjonalne ( chociaż pseudogeny Wykazano, że tRNA biorą udział w oporności bakterii na antybiotyki). Podobnie jak w przypadku wszystkich eukariotów, u ludzi występują 22 mitochondrialne geny tRNA [ potrzebne pełne źródło ] . Mutacje w niektórych z tych genów były związane z poważnymi chorobami, takimi jak zespół MELAS . Zidentyfikowano również regiony w chromosomach jądrowych, bardzo podobne w sekwencji do mitochondrialnych genów tRNA (podobne do tRNA). Te podobne do tRNA są również uważane za część jądrowego mitochondrialnego DNA (geny przenoszone z mitochondriów do jądra). Zjawisko wielu jądrowych kopii mitochondrialnego tRNA (podobnych do tRNA) zaobserwowano w wielu organizmach wyższych, od człowieka po oposa, co sugeruje możliwość, że sobowtóry są funkcjonalne.
Cytoplazmatyczne geny tRNA można podzielić na 49 rodzin zgodnie z ich cechami antykodonowymi. Geny te znajdują się na wszystkich chromosomach, z wyjątkiem chromosomu 22 i chromosomu Y. Obserwuje się wysokie skupienie na 6p (140 genów tRNA), jak również na 1 chromosomie.
HGNC we współpracy z Genomic tRNA Database ( GtRNAdb ) i ekspertami w tej dziedzinie zatwierdziła unikalne nazwy ludzkich genów, które kodują tRNA .
Ewolucja
Górna połowa tRNA (składająca się z ramienia T i łodygi akceptorowej z 5'-końcową grupą fosforanową i 3'-końcową grupą CCA) i dolna połowa (składająca się z ramienia D i ramienia antykodonowego) są niezależnymi jednostkami w strukturze jak i w funkcji. Górna połowa mogła wyewoluować jako pierwsza, w tym 3-końcowy znacznik genomowy, który pierwotnie mógł oznaczać cząsteczki podobne do tRNA do replikacji we wczesnym świecie RNA . Dolna połowa mogła ewoluować później jako ekspansja, np. Gdy synteza białek rozpoczęła się w świecie RNA i przekształciła go w świat rybonukleoprotein ( świat RNP ). Ten proponowany scenariusz nazywa się hipotezą znacznika genomowego. W rzeczywistości agregaty tRNA i tRNA-podobne mają istotny wpływ katalityczny (tj. jako rybozymy ) na replikację do dziś. Role te można uznać za „ molekularne (lub chemiczne) skamieniałości ” świata RNA.
Zawartość genomowego tRNA jest cechą różnicującą genomy wśród biologicznych domen życia: Archaea przedstawiają najprostszą sytuację pod względem zawartości genomowego tRNA z jednolitą liczbą kopii genów, Bakterie mają sytuację pośrednią, a Eukarya przedstawiają najbardziej złożoną sytuację. Eukarya mają nie tylko większą zawartość genów tRNA niż pozostałe dwa królestwa, ale także duże zróżnicowanie liczby kopii genów wśród różnych izoakceptorów, a ta złożoność wydaje się wynikać z duplikacji genów tRNA i zmian w specyficzności antykodonowej [ potrzebne źródło ] .
Ewolucja liczby kopii genu tRNA u różnych gatunków została powiązana z pojawieniem się specyficznych enzymów modyfikujących tRNA (metylotransferazy urydyny u bakterii i deaminaz adenozyny u eukariotów), które zwiększają zdolność dekodowania danego tRNA. Na przykład tRNAAla koduje cztery różne izoakceptory tRNA (AGC, UGC, GGC i CGC). U Eukaryi izoakceptory AGC są niezwykle wzbogacone w liczbę kopii genów w porównaniu z resztą izoakceptorów, co zostało skorelowane z modyfikacją A-do-I jego podstawy chybotania. Ten sam trend wykazano dla większości aminokwasów gatunków eukarioli. Rzeczywiście, efekt tych dwóch modyfikacji tRNA jest również widoczny w błąd użycia kodonów . Wydaje się, że geny o wysokiej ekspresji są wzbogacone w kodony, które wykorzystują wyłącznie kodony, które będą dekodowane przez te zmodyfikowane tRNA, co sugeruje możliwą rolę tych kodonów - aw konsekwencji tych modyfikacji tRNA - w wydajności translacji.
Należy zauważyć, że wiele gatunków utraciło określone tRNA podczas ewolucji. Na przykład zarówno ssaki, jak i ptaki nie mają tych samych 14 z możliwych 64 genów tRNA, ale inne formy życia zawierają te tRNA. W celu translacji kodonów, dla których brakuje dokładnie sparowanego tRNA, organizmy uciekają się do strategii zwanej wobblingiem , w której niedokładnie dopasowane pary tRNA/mRNA nadal powodują translację, chociaż strategia ta zwiększa również skłonność do błędów translacji. Powody, dla których geny tRNA zostały utracone podczas ewolucji, pozostają przedmiotem dyskusji, ale mogą dotyczyć poprawy odporności na infekcje wirusowe. Ponieważ trójki nukleotydów mogą prezentować więcej kombinacji niż aminokwasów i związanych z nimi tRNA, w kodzie genetycznym występuje redundancja, a kilka różnych kodonów 3-nukleotydowych może wyrażać ten sam aminokwas. To odchylenie kodonów jest tym, co wymaga optymalizacji kodonów.
fragmenty pochodzące z tRNA
Fragmenty pochodzące z tRNA (lub tRF) to krótkie cząsteczki, które powstają po rozszczepieniu dojrzałego tRNA lub transkryptu prekursora. Zarówno cytoplazmatyczne, jak i mitochondrialne tRNA mogą wytwarzać fragmenty. Uważa się, że istnieją co najmniej cztery typy strukturalne tRF pochodzące z dojrzałych tRNA, w tym stosunkowo długie połówki tRNA i krótkie 5'-tRF, 3'-tRF i i-tRF. Prekursorowy tRNA można rozszczepić, aby wytworzyć cząsteczki z sekwencji lidera 5' lub sekwencji szlaku 3'. Enzymy rozszczepiające obejmują angiogeninę, Dicer, RNazę Z i RNazę P. Szczególnie w przypadku angiogeniny, tRF mają charakterystycznie niezwykły cykliczny fosforan na swoim końcu 3' i grupę hydroksylową na końcu 5'. Wydaje się, że tRF odgrywają rolę w Interferencja RNA , szczególnie w supresji retrowirusów i retrotranspozonów, które wykorzystują tRNA jako starter do replikacji. Pół-tRNA rozszczepiane przez angiogeninę są również znane jako tiRNA. Biogeneza mniejszych fragmentów, w tym tych, które działają jako piRNA , jest mniej poznana.
tRF mają wiele zależności i ról; takie jak wykazywanie znaczących zmian między płciami, rasami i statusem chorobowym. Funkcjonalnie mogą być ładowane na Ago i działać poprzez szlaki RNAi, uczestniczyć w tworzeniu granulek stresowych, wypierać mRNA z białek wiążących RNA lub hamować translację. Na poziomie systemu lub organizmu cztery typy tRF mają zróżnicowane spektrum działań. Funkcjonalnie tRF są związane z infekcją wirusową, rakiem, proliferacją komórek, a także z epigenetyczną międzypokoleniową regulacją metabolizmu.
tRF nie są ograniczone do ludzi i wykazano, że istnieją w wielu organizmach.
, którzy chcą dowiedzieć się więcej o tRF, dostępne są dwa narzędzia online: platforma do interaktywnej eksploracji mitochondrialnych i jądrowych fragmentów t RNA ( MINTbase ) oraz relacyjna baza danych fragmentów związanych z transferem RNA ( tRFdb ). MINTbase zapewnia również schemat nazewnictwa dla nazewnictwa tRF zwanych tablicami rejestracyjnymi tRF (lub kodami MINT), który jest niezależny od genomu; schemat kompresuje sekwencję RNA do krótszego łańcucha.
Zaprojektowane tRNA
Sztuczne wydłużające supresorowe tRNA są używane do włączania nienaturalnych aminokwasów w kodonach nonsensownych umieszczonych w sekwencji kodującej genu. Zaprojektowane inicjatorowe tRNA (tRNA fMet2 z antykodonem CUA kodowanym przez gen metY ) zostały użyte do zainicjowania translacji w bursztynowym kodonie stop UAG. Ten typ zmodyfikowanego tRNA jest nazywany nonsensownym supresorowym tRNA, ponieważ tłumi sygnał zatrzymania translacji, który normalnie występuje w kodonach UAG. Bursztynowy inicjator tRNA wstawia metioninę i glutaminę w kodony UAG poprzedzone silnym Sekwencja Shine-Dalgarno . Badanie tRNA bursztynowego inicjatora wykazało, że było ono ortogonalne w stosunku do zwykłego kodonu startowego AUG, nie wykazując żadnych wykrywalnych zdarzeń inicjacji translacji poza celem w genomowo zakodowanym E. coli .
biogeneza tRNA
W komórkach eukariotycznych tRNA są transkrybowane przez polimerazę RNA III jako pre-tRNA w jądrze. Polimeraza RNA III rozpoznaje dwie wysoce konserwatywne sekwencje promotora w dół: wewnątrzgenowy region kontrolny 5 '(5'-ICR, region kontrolny D lub blok A) i 3'-ICR (region kontrolny T lub blok B) wewnątrz tRNA geny. Pierwszy promotor zaczyna się od +8 dojrzałych tRNA, a drugi promotor znajduje się 30–60 nukleotydów poniżej pierwszego promotora. Transkrypcja kończy się po odcinku czterech lub więcej tymidyn .
Pre-tRNA ulegają rozległym modyfikacjom wewnątrz jądra. Niektóre pre-tRNA zawierają introny , które są łączone lub cięte w celu utworzenia funkcjonalnej cząsteczki tRNA; u bakterii ulegają samoskładaniu , podczas gdy u eukariontów i archeonów są usuwane przez endonukleazy splicingowe tRNA . Eukariotyczny pre-tRNA zawiera motyw struktury wybrzuszenie-helisa-wybrzuszenie (BHB), który jest ważny dla rozpoznawania i precyzyjnego składania intronu tRNA przez endonukleazy. Ta pozycja i struktura motywu są zachowane ewolucyjnie. Jednak niektóre organizmy, takie jak glony jednokomórkowe, mają niekanoniczną pozycję motywu BHB, jak również 5'- i 3'-końce złożonej sekwencji intronu. Sekwencja 5' jest usuwana przez RNazę P , podczas gdy koniec 3' jest usuwany przez enzym tRNazę Z. Godnym uwagi wyjątkiem jest archeon Nanoarchaeum equitans , który nie posiada enzymu RNazy P i ma promotor umieszczony w taki sposób, że transkrypcja rozpoczyna się na końcu 5' dojrzałego tRNA. Ogon 3 'CCA bez matrycy jest dodawany przez transferazę nukleotydylową . Zanim tRNA zostaną wyeksportowane do cytoplazmy przez Los1/ Xpo-t , tRNA są aminoacylowane . Kolejność zdarzeń przetwarzania nie jest zachowywana. Na przykład u drożdży splicing nie zachodzi w jądrze, ale po cytoplazmatycznej stronie błon mitochondrialnych .
Niemniej jednak w marcu 2021 roku naukowcy przedstawili dowody sugerujące, że wstępna forma transferowego RNA mogła być cząsteczką replikatora na bardzo wczesnym etapie rozwoju życia, czyli abiogenezie .
Historia
Istnienie tRNA zostało po raz pierwszy postawione przez Francisa Cricka jako „ hipoteza adaptera ” oparta na założeniu, że musi istnieć cząsteczka adaptera zdolna do pośredniczenia w translacji alfabetu RNA na alfabet białkowy. Paul C. Zamecnik i Mahlon Hoagland odkryli tRNA Znaczące badania nad strukturą zostały przeprowadzone na początku lat 60 . grupa w King’s College w Londynie . W 1965 roku Robert W. Holley z Cornell University opisał strukturę pierwotną i zasugerował trzy struktury drugorzędowe. tRNA został po raz pierwszy skrystalizowany w Madison w stanie Wisconsin przez Roberta M. Bocka. Struktura koniczyny została potwierdzona w kilku innych badaniach w następnych latach i ostatecznie potwierdzona za pomocą krystalografii rentgenowskiej w 1974 roku. Dwie niezależne grupy, Kim Sung-Hou pracujący pod kierunkiem Alexandra Richa i brytyjska grupa kierowana przez Aarona Kluga , opublikował te same wyniki krystalografii w ciągu roku.