Pierwotny transkrypcja

Pierwotny transkrypt to jednoniciowy kwas rybonukleinowy ( RNA ) syntetyzowany w drodze transkrypcji DNA i przetwarzany w celu uzyskania różnych dojrzałych produktów RNA , takich jak mRNA , tRNA i rRNA . Pierwotne transkrypty określone jako mRNA są modyfikowane w ramach przygotowań do translacji . Na przykład prekursorowy mRNA ( pre-mRNA ) to rodzaj pierwotnego transkryptu, który po przetworzeniu staje się informacyjnym RNA (mRNA) .
Pre-mRNA jest syntetyzowany z matrycy DNA w jądrze komórkowym na drodze transkrypcji . Pre-mRNA obejmuje większość heterogennego jądrowego RNA ( hnRNA ). Po całkowitym przetworzeniu pre-mRNA określa się go mianem „ dojrzałego informacyjnego RNA ” lub po prostu „ wiadomościowego RNA ”. Termin hnRNA jest często używany jako synonim pre-mRNA, chociaż w ścisłym tego słowa znaczeniu hnRNA może obejmować jądrowe transkrypty RNA, które nie kończą jako cytoplazmatyczny mRNA.
Istnieje kilka etapów przyczyniających się do produkcji pierwotnych transkryptów. Wszystkie te etapy obejmują szereg interakcji w celu zainicjowania i zakończenia transkrypcji DNA w jądrze eukariontów . Pewne czynniki odgrywają kluczową rolę w aktywacji i hamowaniu transkrypcji, gdzie regulują produkcję pierwotnego transkryptu. Transkrypcja tworzy pierwotne transkrypty, które są dalej modyfikowane przez kilka procesów. Procesy te obejmują czapeczkę 5' , poliadenylację 3' i splicing alternatywny . W szczególności alternatywny splicing bezpośrednio przyczynia się do różnorodności mRNA występującego w komórkach. Modyfikacje pierwotnych transkryptów były dalej badane w badaniach poszukujących większej wiedzy na temat roli i znaczenia tych transkryptów. Badania eksperymentalne oparte na zmianach molekularnych w pierwotnych transkryptach oraz procesach przed i po transkrypcji doprowadziły do lepszego zrozumienia chorób związanych z pierwotnymi transkryptami.
Produkcja
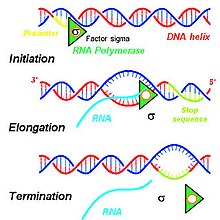
Etapy przyczyniające się do produkcji pierwotnych transkryptów obejmują szereg interakcji molekularnych, które inicjują transkrypcję DNA w jądrze komórkowym. W zależności od potrzeb danej komórki, niektóre sekwencje DNA są transkrybowane w celu wytworzenia różnych produktów RNA, które mają zostać przetłumaczone na funkcjonalne białka do użytku komórkowego. Aby zainicjować proces transkrypcji w jądrze komórkowym, podwójne helisy DNA są rozwijane i wiązania wodorowe łączące zgodne kwasy nukleinowe DNA są rozrywane, tworząc dwie niepołączone pojedyncze nici DNA. Jedna nić matrycy DNA jest używana do transkrypcji jednoniciowego mRNA pierwotnego transkryptu. Ta nić DNA jest wiązana przez polimerazę RNA w regionie promotora DNA.
U eukariontów trzy rodzaje RNA — rRNA , tRNA i mRNA — są wytwarzane w oparciu o aktywność trzech różnych polimeraz RNA, podczas gdy u prokariotów istnieje tylko jedna polimeraza RNA, która tworzy wszystkie rodzaje cząsteczek RNA. Polimeraza RNA II eukariontów dokonuje transkrypcji pierwotnego transkryptu, transkryptu przeznaczonego do przetworzenia w mRNA, z antysensownej matrycy DNA w kierunku od 5' do 3', a ten nowo zsyntetyzowany pierwotny transkrypt jest komplementarny do antysensownej nici DNA. Polimeraza RNA II konstruuje pierwotny transkrypt przy użyciu zestawu czterech specyficznych reszty monofosforanu rybonukleozydu ( monofosforan adenozyny (AMP), monofosforan cytydyny (CMP), monofosforan guanozyny (GMP) i monofosforan urydyny (UMP)), które są dodawane w sposób ciągły do grupy hydroksylowej 3' na końcu 3' rosnącego mRNA.
Badania pierwotnych transkryptów wytwarzanych przez polimerazę RNA II ujawniają, że przeciętny pierwotny transkrypt ma długość 7 000 nukleotydów , a niektóre dorastają do 20 000 nukleotydów. Włączenie zarówno eksonu , jak i intronu do pierwotnych transkryptów wyjaśnia różnicę wielkości między większymi pierwotnymi transkryptami a mniejszym, dojrzałym mRNA gotowym do translacji na białko.
Rozporządzenie
Szereg czynników przyczynia się do aktywacji i hamowania transkrypcji, a zatem reguluje produkcję pierwotnych transkryptów z danej matrycy DNA.
Aktywacja aktywności polimerazy RNA w celu wytworzenia pierwotnych transkryptów jest często kontrolowana przez sekwencje DNA zwane wzmacniaczami . Czynniki transkrypcyjne , białka, które wiążą się z elementami DNA w celu aktywacji lub hamowania transkrypcji, wiążą się ze wzmacniaczami i rekrutują enzymy, które zmieniają nukleosom składników, powodując, że DNA jest mniej lub bardziej dostępne dla polimerazy RNA. Unikalne kombinacje aktywujących lub hamujących czynników transkrypcyjnych, które wiążą się z regionami wzmacniacza DNA, określają, czy gen, z którym wzmacniacz oddziałuje, jest aktywowany do transkrypcji, czy nie. Aktywacja transkrypcji zależy od tego, czy kompleks wydłużania transkrypcji, który sam składa się z różnych czynników transkrypcyjnych, może indukować polimerazę RNA do dysocjacji od kompleksu Mediator, który łączy region wzmacniający z promotorem.
Hamowanie aktywności polimerazy RNA może być również regulowane przez sekwencje DNA zwane wyciszaczami . Podobnie jak wzmacniacze, tłumiki mogą znajdować się w miejscach położonych dalej w górę lub w dół od genów, które regulują. Te sekwencje DNA wiążą się z czynnikami, które przyczyniają się do destabilizacji kompleksu inicjacyjnego wymaganego do aktywacji polimerazy RNA, a zatem hamują transkrypcję.
histonów przez czynniki transkrypcyjne jest kolejnym kluczowym czynnikiem regulującym transkrypcję przez polimerazę RNA. Ogólnie rzecz biorąc, czynniki prowadzące do acetylacji histonów aktywują transkrypcję, podczas gdy czynniki prowadzące do deacetylacji histonów hamować transkrypcję. Acetylacja histonów indukuje odpychanie między ujemnymi składnikami w nukleosomach, umożliwiając dostęp polimerazy RNA. Deacetylacja histonów stabilizuje ciasno zwinięte nukleosomy, hamując dostęp polimerazy RNA. Oprócz wzorców acetylacji histonów, wzorce metylacji w regionach promotorowych DNA mogą regulować dostęp polimerazy RNA do danej matrycy. Polimeraza RNA często nie jest zdolna do syntezy pierwotnego transkryptu, jeśli region promotora docelowego genu zawiera specyficzne metylowane cytozyny – reszty, które utrudniają wiązanie czynników aktywujących transkrypcję i rekrutują inne enzymy do stabilizacji ściśle związanej struktury nukleosomu, wykluczając dostęp do polimerazy RNA i zapobiegając tworzenie transkryptów pierwotnych.
Pętle R
Podczas transkrypcji powstają pętle R. Pętla R to trójniciowa struktura kwasu nukleinowego zawierająca region hybrydowy DNA-RNA i związany z nim jednoniciowy DNA bez matrycy. Aktywnie transkrybowane regiony DNA często tworzą pętle R, które są podatne na uszkodzenia DNA . Introny zmniejszają tworzenie pętli R i uszkodzenia DNA w genach drożdży o wysokiej ekspresji.
przetwarzanie RNA
Transkrypcja, wysoce regulowana faza ekspresji genów, wytwarza pierwotne transkrypty. Jednak transkrypcja to tylko pierwszy krok, po którym powinno nastąpić wiele modyfikacji, które dają funkcjonalne formy RNA. Innymi słowy, nowo zsyntetyzowane pierwotne transkrypty są modyfikowane na kilka sposobów w celu przekształcenia ich w dojrzałe, funkcjonalne formy w celu wytworzenia różnych białek i RNA, takich jak mRNA, tRNA i rRNA.
Przetwarzanie
Podstawowy proces pierwotnej modyfikacji transkryptu jest podobny dla tRNA i rRNA zarówno w komórkach eukariotycznych, jak i prokariotycznych. Z drugiej strony pierwotne przetwarzanie transkryptu różni się w mRNA komórek prokariotycznych i eukariotycznych. Na przykład niektóre mRNA bakterii prokariotycznych służą jako matryce do syntezy białek w tym samym czasie, gdy są wytwarzane w drodze transkrypcji. Alternatywnie, pre-mRNA komórek eukariotycznych przechodzi szeroki zakres modyfikacji przed ich transportem z jądra do cytoplazmy, gdzie ich dojrzałe formy ulegają translacji. Modyfikacje te są odpowiedzialne za różne typy zakodowanych wiadomości, które prowadzą do tłumaczenia różnych typów produktów. Ponadto pierwotne przetwarzanie transkryptu zapewnia kontrolę ekspresji genów, a także mechanizm regulacyjny szybkości degradacji mRNA. Obróbka pre-mRNA w komórkach eukariotycznych obejmuje Capping 5' , poliadenylacja 3' i splicing alternatywny .
5' zakrycie
Krótko po zainicjowaniu transkrypcji u eukariotów koniec 5' pre-mRNA jest modyfikowany przez dodanie czapeczki 7-metyloguanozynowej , znanej również jako czapeczka 5'. Modyfikację czapeczki na końcu 5' inicjuje się przez dodanie GTP do końcowego 5' nukleotydu pre-mRNA w odwrotnej orientacji, a następnie dodanie grup metylowych do reszty G. Czapeczka 5' jest niezbędna do produkcji funkcjonalnych mRNA, ponieważ czapeczka 5' jest odpowiedzialna za dopasowanie mRNA do rybosomu podczas translacji.
Poliadenylacja
dodawana jest struktura zwana ogonem poli-A . Sygnały poliadenylacji, które obejmują kilka elementów sekwencji RNA, są wykrywane przez grupę białek sygnalizujących dodanie ogona poli-A (o długości około 200 nukleotydów). Reakcja poliadenylacji dostarcza sygnał do końca transkrypcji, a ta reakcja kończy się około kilkuset nukleotydów poniżej lokalizacji ogona poli-A.
Splicing alternatywny
Eukariotyczne pre-mRNA mają swoje introny wycięte przez spliceosomy zbudowane z małych jądrowych rybonukleoprotein .
W złożonych komórkach eukariotycznych jeden pierwotny transkrypt jest w stanie przygotować duże ilości dojrzałych mRNA dzięki alternatywnemu splicingowi. Splicing alternatywny jest regulowany w taki sposób, że każdy dojrzały mRNA może kodować wiele białek.
Wpływ alternatywnego splicingu na ekspresję genów można zaobserwować u złożonych eukariotów, które mają ustaloną liczbę genów w swoim genomie, ale wytwarzają znacznie większą liczbę różnych produktów genów. Większość eukariotycznych transkryptów pre-mRNA zawiera wiele intronów i eksonów. Różne możliwe kombinacje miejsc splicingowych 5' i 3' w pre-mRNA mogą prowadzić do różnych cięć i kombinacji egzonów, podczas gdy introny są eliminowane z dojrzałego mRNA. W ten sposób generowane są różne rodzaje dojrzałych mRNA. Splicing alternatywny zachodzi w dużym kompleksie białkowym zwanym spliceosomem . Alternatywny splicing ma kluczowe znaczenie dla specyficznej tkankowo i rozwojowej regulacji ekspresji genów. Na splicing alternatywny mogą wpływać różne czynniki, w tym mutacje, takie jak translokacja chromosomalna .
U prokariotów splicing odbywa się przez rozszczepienie autokatalityczne lub rozszczepienie endolityczne. Cięcia autokatalityczne, w których nie biorą udziału żadne białka, są zwykle zarezerwowane dla odcinków kodujących rRNA, podczas gdy cięcie endolityczne odpowiada prekursorom tRNA.
Eksperymenty
Badanie przeprowadzone przez Cindy L. Wills i Bruce'a J. Dolnicka z Wydziału Terapii Eksperymentalnej w Roswell Park Comprehensive Cancer Center (wówczas znanego jako Roswell Park Memorial Institute) w Buffalo w stanie Nowy Jork oraz z programu Cell and Molecular Biology na University of Wisconsin w Madison, Wisconsin został stworzony, aby zrozumieć procesy komórkowe obejmujące pierwotne transkrypty. Naukowcy chcieli zrozumieć, czy 5- fluorouracyl (FUra), lek znany ze stosowania w leczeniu raka, hamuje lub wyłącza reduktazę dihydrofolianową (DHFR) obróbka pre-mRNA i/lub stabilność jądrowego mRNA w komórkach KB opornych na metotreksat . Długotrwała ekspozycja na FUra nie miała wpływu na poziom pre-mRNA DHFR zawierającego pewne introny, czyli odcinki pre-mRNA, które są zwykle wycinane z sekwencji w ramach przetwarzania. Jednak poziomy całkowitego mRNA DHFR zmniejszyły się dwukrotnie w komórkach eksponowanych na 1,0 μM FURa. Nie było znaczących zmian w okresie półtrwania , co odnosi się do czasu potrzebnego do rozpadu 50% mRNA całkowitego mRNA lub pre-mRNA DHFR obserwowanego w komórkach eksponowanych na FUra. Eksperymenty ze znakowaniem jądrowym/cytoplazmatycznym RNA wykazały, że szybkość zmiany jądrowego RNA DHFR na mRNA cytoplazmatycznego DHFR zmniejszyła się w komórkach traktowanych FUra. Wyniki te dostarczają dalszych dowodów na to, że FUra może pomóc w przetwarzaniu prekursorów mRNA i / lub wpływać na stabilność mRNA jądrowego DHFR.
Judith Lengyel i Sheldon Penman z Wydziału Biologii Massachusetts Institute of Technology (MIT) w Cambridge w stanie Massachusetts napisali artykuł o jednym typie pierwotnego transkryptu zaangażowanego w geny dwóch muchówek , czyli owadów, które mają dwa skrzydła: Drosophila i Aedes . W artykule opisano, w jaki sposób naukowcy przyjrzeli się hnRNA, czyli pre-mRNA, pierwotnym transkryptom u dwóch rodzajów owadów. Porównano wielkość transkryptów hnRNA i frakcję hnRNA, która jest przekształcana w mRNA w liniach komórkowych lub grupach komórek pochodzących z pojedynczej komórki Drosophila melanogaster i Aedes albopictus . Oba owady są muchówkami, ale Aedes ma większy genom niż Drosophila . Oznacza to, że Aedes ma więcej DNA, co oznacza więcej genów. Linia Aedes wytwarza większe hnRNA niż Drosophila linii komórkowej, mimo że obie linie komórkowe rosły w podobnych warunkach i wytwarzały dojrzałe lub przetworzone mRNA o tej samej wielkości i złożoności sekwencji. Dane te sugerują, że rozmiar hnRNA wzrasta wraz ze wzrostem rozmiaru genomu, co oczywiście pokazuje Aedes.
Ivo Melcak, Stepanka Melcakova, Vojtech Kopsky, Jaromıra Vecerova i Ivan Raska z Zakładu Biologii Komórki Instytutu Medycyny Doświadczalnej Czeskiej Akademii Nauk w Pradze badali wpływ plamek jądrowych na pre- mRNA . Plamki jądrowe (plamki) są częścią jąder komórkowych i są wzbogacone czynnikami splicingowymi znanymi z udziału w przetwarzaniu mRNA. Wykazano, że plamki jądrowe służą sąsiednim aktywnym genom jako miejsca przechowywania tych czynników splicingowych. W tym badaniu naukowcy wykazali, że w komórkach HeLa pochodzących z komórek osoby chorej na raka szyjki macicy, które okazały się przydatne w eksperymentach, pierwsza grupa spliceosomy na pre-mRNA pochodzą z tych plamek. Naukowcy wykorzystali mikroiniekcje akceptujących spliceosom i zmutowanych adenowirusowych pre-mRNA z różnicowym wiązaniem czynnika splicingowego, aby utworzyć różne grupy, a następnie śledzili miejsca, w których były one silnie obecne. Pre-mRNA akceptujące spliceosomy były szybko kierowane do plamek, ale stwierdzono, że celowanie jest zależne od temperatury. Szlak polipirymidynowy sekwencje w mRNA promują budowę grup spliceosomów i są wymagane do kierowania, ale same w sobie nie były wystarczające. Sekwencje flankujące w dół były szczególnie ważne dla celowania w zmutowane pre-mRNA w plamkach. W eksperymentach pomocniczych śledzono zachowanie plamek po mikroiniekcji antysensownych dezoksyoligorybonukleotydów (sekwencje komplementarne DNA i/lub RNA do określonej sekwencji) oraz, w tym przypadku, specyficzne sekwencje snRNA . snRNA są również znane z tego, że pomagają w przetwarzaniu pre-mRNA. W tych warunkach na endogennych pre-mRNA tworzyły się grupy spliceosomów. Badacze doszli do wniosku, że wewnątrz plamek tworzą się grupy spliceosomów na wstrzykniętym pre-mRNA. Celowanie pre-mRNA i gromadzenie się w plamkach jest wynikiem ładowania czynników splicingowych do pre-mRNA, a grupy spliceosomów dały początek zaobserwowanemu wzorowi plamistości.
Choroby pokrewne
Badania doprowadziły również do poszerzenia wiedzy na temat niektórych chorób związanych ze zmianami w pierwotnych transkryptach. Jedno badanie obejmowało receptory estrogenowe i splicing różnicowy. Artykuł zatytułowany „Alternatywny splicing pierwotnego transkryptu ludzkiego receptora estrogenowego: mechanizmy pomijania eksonów” autorstwa Paoli Ferro, Alessandry Forlani, Marco Muselli i Ulricha Pfeffera z laboratorium onkologii molekularnej w National Cancer Research Institute w Genui we Włoszech, wyjaśnia że 1785 nukleotydów regionu w DNA, który koduje receptor estrogenowy alfa (ER-alfa) jest rozmieszczonych w regionie, który zawiera ponad 300 000 nukleotydów w pierwotnym transkrypcie. Składanie tego pre-mRNA często prowadzi do wariantów lub różnych rodzajów mRNA pozbawionych jednego lub więcej egzonów lub regionów niezbędnych do kodowania białek. Te warianty zostały powiązane z raka piersi . W cyklu życiowym retrowirusów , prowirusowy DNA jest włączany do transkrypcji DNA zakażonej komórki. Ponieważ retrowirusy muszą zmienić swój pre-mRNA w DNA, aby ten DNA mógł zostać zintegrowany z DNA gospodarza, na który ma wpływ, utworzenie tej matrycy DNA jest niezbędnym krokiem do replikacji retrowirusa. Typ komórki, zróżnicowanie lub zmieniony stan komórki oraz stan fizjologiczny komórki powodują istotną zmianę w dostępności i aktywności niektórych czynników niezbędnych do transkrypcji. Te zmienne tworzą szeroki zakres ekspresji genów wirusowych. Na przykład komórki hodowli tkankowej aktywnie wytwarzające zakaźne wiriony ptasie lub mysie białaczkowe (ASLV lub MLV) zawierają tak wysoki poziom wirusowego RNA, że 5–10% mRNA w komórce może być pochodzenia wirusowego. Pokazuje to, że pierwotne transkrypty wytwarzane przez te retrowirusy nie zawsze podążają normalną ścieżką do produkcji białka i przekształcają się z powrotem w DNA w celu namnażania i ekspansji.