Analog kwasu nukleinowego
Analogi kwasów nukleinowych to związki, które są analogiczne (podobne strukturalnie) do naturalnie występujących RNA i DNA , stosowane w medycynie i badaniach biologii molekularnej. Kwasy nukleinowe to łańcuchy nukleotydów, które składają się z trzech części: szkieletu fosforanowego , cukru pentozowego, rybozy lub dezoksyrybozy oraz jednej z czterech zasad nukleinowych . Analog może mieć dowolne z nich zmienione. Zazwyczaj analogowe zasady nukleinowe nadają, między innymi, różne właściwości parowania zasad i układania zasad w stosy. Przykłady obejmują uniwersalne zasady, które mogą łączyć się ze wszystkimi czterema kanonicznymi zasadami, oraz analogi szkieletu fosforanowo-cukrowego, takie jak PNA , które wpływają na właściwości łańcucha (PNA może nawet tworzyć potrójną helisę ). Analogi kwasów nukleinowych są również nazywane kwasami ksenonukleinowymi i stanowią jeden z głównych filarów ksenobiologii , projektowania nowych form życia w naturze w oparciu o alternatywne biochemie.
Sztuczne kwasy nukleinowe obejmują kwas peptydonukleinowy (PNA), morfolinowy i zablokowany kwas nukleinowy (LNA), jak również glikolowy kwas nukleinowy (GNA), kwas nukleinowy treozy (TNA) i heksitolowy kwas nukleinowy (HNA). Każdy z nich różni się od naturalnie występującego DNA lub RNA zmianami w szkielecie cząsteczki.
W maju 2014 r. Naukowcy ogłosili, że z powodzeniem wprowadzili dwa nowe sztuczne nukleotydy do bakteryjnego DNA, a poprzez włączenie pojedynczych sztucznych nukleotydów do pożywki hodowlanej byli w stanie przejść przez bakterie 24 razy; nie stworzyli mRNA ani białek zdolnych do wykorzystania sztucznych nukleotydów. Sztuczne nukleotydy zawierały 2 skondensowane pierścienie aromatyczne.
Medycyna
Kilka analogów nukleozydów stosuje się jako środki przeciwwirusowe lub przeciwnowotworowe. Polimeraza wirusowa łączy te związki z niekanonicznymi zasadami. Związki te są aktywowane w komórkach poprzez przekształcenie w nukleotydy, są podawane jako nukleozydy, ponieważ naładowane nukleotydy nie mogą łatwo przechodzić przez błony komórkowe.
Biologia molekularna
Analogi kwasów nukleinowych są wykorzystywane w biologii molekularnej do kilku celów: Badanie możliwych scenariuszy powstania życia: Testując różne analogi, naukowcy próbują odpowiedzieć na pytanie, czy wykorzystanie DNA i RNA w życiu zostało wybrane w czasie ze względu na jego zalety, lub jeśli zostali wybrani przypadkowo; Jako narzędzie do wykrywania określonych sekwencji: XNA może być używany do znakowania i identyfikowania szerokiego zakresu składników DNA i RNA z wysoką specyficznością i dokładnością; Jako enzym działający na substraty DNA, RNA i XNA - wykazano, że XNA ma zdolność cięcia i ligacji DNA, RNA i inne cząsteczki XNA podobne do działania rybozymów RNA ; Jako narzędzie o odporności na hydrolizę RNA ; Badanie mechanizmów działania enzymu; Badanie cech strukturalnych kwasów nukleinowych.
Analogi kręgosłupa
Odporne na hydrolizę analogi RNA
Aby przezwyciężyć fakt, że grupa hydroksylowa 2' rybozy reaguje z grupą hydroksylową 3' związaną z fosforanem (RNA jest zbyt niestabilny, aby można go było niezawodnie wykorzystać lub zsyntetyzować), stosuje się analog rybozy. Najczęstszymi analogami RNA są 2'-O-metylo-podstawiony RNA, zablokowany kwas nukleinowy (LNA) lub zmostkowany kwas nukleinowy (BNA), morfolino i peptydowy kwas nukleinowy ( PNA ). Chociaż te oligonukleotydy mają inny cukier szkieletowy lub, w przypadku PNA, resztę aminokwasową zamiast fosforanu rybozy, nadal wiążą się z RNA lub DNA zgodnie z parowaniem Watsona i Cricka, ale są odporne na działanie nukleazy. Nie można ich zsyntetyzować enzymatycznie i można je otrzymać jedynie syntetycznie, stosując strategię amidofosforanową lub, w przypadku PNA, metody syntezy peptydów .
Inne godne uwagi analogi używane jako narzędzia
sekwencjonowania stosuje się dideoksynukleotydy . Te trifosforany nukleozydów zawierają niekanoniczny cukier, dideoksyrybozę, który nie ma grupy hydroksylowej 3' normalnie obecnej w DNA i dlatego nie może wiązać się z następną zasadą. Brak grupy hydroksylowej 3' kończy reakcję łańcuchową, ponieważ polimerazy DNA mylą ją z regularnym dezoksyrybonukleotydem. Innym analogiem kończącym łańcuch, który nie ma 3'-hydroksylu i naśladuje adenozynę , jest kordycepina . Kordycepina to lek przeciwnowotworowy, którego celem jest RNA replikacja. Innym analogiem w sekwencjonowaniu jest analog zasady nukleinowej, 7-deaza-GTP i jest używany do sekwencjonowania regionów bogatych w CG, zamiast tego 7-deaza-ATP jest nazywany tubercydyną, antybiotykiem.
Prekursory świata RNA
RNA może być zbyt złożony, aby być pierwszym kwasem nukleinowym, więc przed światem RNA zaproponowano kilka prostszych kwasów nukleinowych różniących się szkieletem, takich jak TNA , GNA i PNA , jako kandydatów na pierwsze kwasy nukleinowe.
Podstawowe analogi
Struktura zasad jądrowych i nomenklatura
Naturalnie występujące zasady można podzielić na dwie klasy w zależności od ich budowy:
- pirymidyny są sześcioczłonowymi heterocyklami z atomami azotu w pozycjach 1 i 3.
- puryny są bicykliczne i składają się z pirymidyny skondensowanej z pierścieniem imidazolowym.
Sztuczne nukleotydy ( nienaturalne pary zasad (UBP) nazwane d5SICS UBP i dNaM UBP ) zostały wstawione do bakteryjnego DNA, ale te geny nie tworzyły szablonu mRNA ani nie indukowały syntezy białek. Sztuczne nukleotydy zawierały dwa skondensowane pierścienie aromatyczne, które tworzyły kompleks (d5SICS-dNaM) naśladujący naturalną parę zasad (dG-dC).
Mutageny
Jednym z najpowszechniejszych analogów zasad jest 5-bromouracyl (5BU), nieprawidłowa zasada występująca w mutagennym analogu nukleotydu BrdU. Kiedy nukleotyd zawierający 5-bromouracyl jest włączony do DNA, najprawdopodobniej sparuje się z adeniną; jednak może spontanicznie przejść do innego izomeru , który łączy się w pary z inną zasadą nukleinową , guaniną . Jeśli zdarzy się to podczas replikacji DNA, guanina zostanie wstawiona jako przeciwny analog zasady, a podczas następnej replikacji DNA ta guanina połączy się z cytozyną. Powoduje to zmianę w jednej parze zasad DNA, w szczególności a mutacja przejściowa .
Ponadto HNO2 lub kwas azotawy jest silnym mutagenem, który działa na replikujące się i niereplikujące DNA. Może powodować deaminację grup aminowych adeniny, guaniny i cytozyny. Adenina jest deaminowana do hipoksantyny , która tworzy pary zasad z cytozyną zamiast tyminy. Cytozyna jest deaminowana do uracylu, który łączy się w pary z adeniną zamiast guaniną. Deaminacja guaniny nie jest mutagenna. Mutacje wywołane kwasem azotawym są również indukowane do mutacji z powrotem do typu dzikiego przy użyciu kwasu azotawego.
fluorofory
Zwykle fluorofory (takie jak rodamina lub fluoresceina ) są połączone z pierścieniem połączonym z cukrem (w para) za pomocą elastycznego ramienia, przypuszczalnie wystającego z głównego rowka helisy. Ze względu na niską przetwarzalność nukleotydów połączonych z masowymi adduktami, takimi jak florofory, przez polimerazy taq, sekwencja jest zwykle kopiowana przy użyciu nukleotydu z ramieniem, a następnie sprzęgana z reaktywnym fluoroforem (znakowanie pośrednie):
- reaktywny z aminą: nukleotyd aminoallilowy zawiera pierwszorzędową grupę aminową na łączniku, który reaguje z barwnikiem reagującym z aminami, takim jak barwniki cyjaninowe lub Alexa Fluor , które zawierają reaktywną grupę opuszczającą, taką jak ester sukcynoimidylowy (NHS). (nie ma to wpływu na grupy aminowe tworzące pary zasad).
- tiol reaktywny: nukleotydy zawierające tiol reagują z fluoroforem połączonym z reaktywną grupą opuszczającą, taką jak maleimid.
- biotyną opierają się na tej samej zasadzie znakowania pośredniego (+ fluorescencyjna streptawidyna) i są stosowane w czipach DNA Affymetrix .
Fluorofory znajdują różnorodne zastosowania w medycynie i biochemii.
Fluorescencyjne analogi zasad
Najczęściej stosowany i dostępny w handlu fluorescencyjny analog zasady, 2-aminopuryna (2-AP), ma wysoką wydajność kwantową fluorescencji bez roztworu (0,68), która jest znacznie zmniejszona (około 100 razy, ale w dużym stopniu zależy od sekwencji zasad), gdy wbudowywane w kwasy nukleinowe. Czułość emisji 2-AP do bezpośredniego otoczenia jest wspólna dla innych obiecujących i użytecznych analogów zasad fluorescencyjnych, takich jak 3-MI, 6-MI, 6-MAP, pirolo-dC (również dostępne w handlu), zmodyfikowane i ulepszone pochodne pirolo-dC , zasady modyfikowane furanem i wiele innych (patrz ostatnie recenzje). Ta wrażliwość na mikrośrodowisko została wykorzystana w badaniach m.in. struktury i dynamiki DNA i RNA, dynamiki i kinetyki interakcji DNA-białko oraz transferu elektronów w DNA.
Nowo opracowaną i bardzo interesującą grupą fluorescencyjnych analogów zasad, których wydajność kwantowa fluorescencji jest prawie niewrażliwa na ich bezpośrednie otoczenie, jest rodzina tricyklicznych cytozyn. 1,3-Diaza-2-oksofenotiazyna, tC, ma wydajność kwantową fluorescencji około 0,2 zarówno w przypadku pojedynczej, jak i podwójnej nici, niezależnie od otaczających zasad. Również okso-homolog tC zwany tC O (oba dostępne w handlu), 1,3-diaza-2-oksofenoksazyna, ma wydajność kwantową 0,2 w układach dwuniciowych. Jest jednak nieco wrażliwy na otaczające zasady w pojedynczych niciach (wydajności kwantowe 0,14–0,41). Wysokie i stabilne wydajności kwantowe tych analogów zasad czynią je bardzo jasnymi, a w połączeniu z ich dobrymi właściwościami analogów zasad (pozostawia strukturę DNA i stabilność prawie nienaruszoną), są one szczególnie przydatne w pomiarach anizotropii fluorescencji i FRET, obszarach, w których inne fluorescencyjne analogi zasad są mniej dokładne. Również w tej samej rodzinie analogów cytozyny, analog zasady akceptora FRET, tC nitro , został opracowany. Wraz z tCO jako donorem FRET stanowi to pierwszą opracowaną parę FRET analogu zasady kwasu nukleinowego. Rodzina tC została na przykład wykorzystana w badaniach związanych z mechanizmami wiązania DNA polimerazy i polimeryzacji DNA.
Naturalne bazy niekanoniczne
W komórce występuje kilka niekanonicznych zasad: wyspy CpG w DNA (często są metylowane), wszystkie eukariotyczne mRNA (zakończone metylo-7-guanozyną) i kilka zasad rRNA (są metylowane). Często tRNA są silnie modyfikowane potranskrypcyjnie w celu poprawy ich konformacji lub parowania zasad, w szczególności w/w pobliżu antykodonu: inozyna może tworzyć pary zasad z C, U, a nawet z A, podczas gdy tiourydyna (z A) jest bardziej specyficzna niż uracyl (z puryną). Inne powszechne modyfikacje zasad tRNA to pseudourydyna (od której pochodzi nazwa pętli TΨC), dihydrourydyna (która nie układa się w stos, ponieważ nie jest aromatyczna), queuozyna, wyozyna i tak dalej. Niemniej jednak są to wszystkie modyfikacje normalnych zasad i nie są umieszczane przez polimerazę.
Parowanie podstawowe
Zasady kanoniczne mogą mieć grupę karbonylową lub aminową na atomach węgla otaczających atom azotu najdalej od wiązania glikozydowego, co umożliwia im tworzenie par zasad (parowanie zasad Watsona-Cricka) poprzez wiązania wodorowe (amina z ketonem, puryna z pirymidyną) . Adenina i 2-aminoadenina mają jedną lub dwie grupy aminowe, podczas gdy tymina ma dwie grupy karbonylowe, a cytozyna i guanina są mieszanymi grupami aminowymi i karbonylowymi (odwróconymi względem siebie).
| Naturalne pary zasad | |
|---|---|

|

|
| Para zasad GC: karbonyl purynowy / amina tworzy trzy międzycząsteczkowe wiązania wodorowe z aminą pirymidynową / karbonylem | Para zasad AT: amina purynowa/- tworzy dwa międzycząsteczkowe wiązania wodorowe z karbonylem/karbonylem pirymidyny |
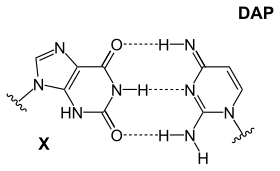
Dokładny powód, dla którego istnieją tylko cztery nukleotydy, jest przedmiotem dyskusji, ale istnieje kilka niewykorzystanych możliwości. Co więcej, adenina nie jest najbardziej stabilnym wyborem do parowania zasad: w Cyanophage S-2L diaminopuryna (DAP) jest używana zamiast adeniny ( unikanie gospodarza ). Diaminopuryna doskonale łączy się z tyminą, ponieważ jest identyczna z adeniną, ale ma grupę aminową w pozycji 2, tworząc 3 wewnątrzcząsteczkowe wiązania wodorowe, eliminując główną różnicę między dwoma typami par zasad (słaby: AT i silny: CG). Ta poprawiona stabilność wpływa na interakcje wiązania białek, które opierają się na tych różnicach. Inne kombinacje obejmują,
- izoguanina i izocytozyna, które mają odwrócone aminy i ketony w porównaniu ze standardową guaniną i cytozyną (prawdopodobnie nie używane, ponieważ tautomery są problematyczne dla parowania zasad, ale isoC i isoG można prawidłowo amplifikować za pomocą PCR, nawet w obecności 4 kanonicznych zasad)
- diaminopirymidyna i ksantyna, które wiążą się jak 2-aminoadenina i tymina, ale z odwróconymi strukturami (nie używane, ponieważ ksantyna jest produktem deaminacji)
| Niewykorzystane układy par podstawowych | ||
|---|---|---|

|
|

|
| Zasada DAP-T: amina/amina purynowa tworzy trzy międzycząsteczkowe wiązania wodorowe z ketonem/ketonem pirymidynowym | Zasada X-DAP: keton/keton purynowy tworzy trzy międzycząsteczkowe wiązania wodorowe z aminą/aminą pirymidyny | Zasada iG-iC: amina/keton purynowy tworzy trzy międzycząsteczkowe wiązania wodorowe z ketonem/aminą pirymidynową |
Jednak prawidłowa struktura DNA może powstać nawet wtedy, gdy zasady nie są sparowane poprzez wiązania wodorowe; to znaczy pary zasad dzięki hydrofobowości, jak wykazały badania przy użyciu izosterów DNA (analogów o tej samej liczbie atomów), takich jak analog tyminy 2,4-difluorotoluen (F) lub analog adeniny 4-metylobenzimidazol (Z). Alternatywną parą hydrofobową może być izochinolina, a pirolo[2,3-b]pirydyna
Inne godne uwagi pary podstawowe:
- Wytworzono również kilka zasad fluorescencyjnych, takich jak para zasad 2-amino-6- (2-tienylo) purynowych i pirolo-2-karboaldehydowych.
- Zasady koordynowane metalem, takie jak parowanie między pirydyno-2,6-dikarboksylanem (ligand trójkleszczowy) i pirydyną (ligand jednokleszczowy) poprzez kwadratową koordynację planarną z centralnym jonem miedzi.
- Zasady uniwersalne mogą łączyć się w pary bez wyjątku z dowolną inną zasadą, ale generalnie znacznie obniżają temperaturę topnienia sekwencji; przykłady obejmują pochodne 2'-deoksyinozyny (deoksynukleotyd hipoksantyny), analogi nitroazolu i hydrofobowe aromatyczne zasady niewiążące wiązań wodorowych (silne efekty układania). Są one używane jako dowód koncepcji i generalnie nie są wykorzystywane w zdegenerowanych starterach (które są mieszaniną starterów).
- Liczba możliwych par zasad jest podwojona, gdy bierze się pod uwagę xDNA . xDNA zawiera rozszerzone zasady, w których dodano pierścień benzenowy, który może łączyć się z kanonicznymi zasadami, dając cztery możliwe pary zasad (8 zasad:xA-T,xT-A,xC-G,xG-C, 16 zasad w przypadku wykorzystania niewykorzystanych układów). Inną postacią zasad z dodanym benzenem jest yDNA, w którym zasada jest poszerzona przez benzen.
| Nowe pary zasad o specjalnych właściwościach | ||
|---|---|---|

|

|

|
| Zasada A FZ: metylobenzimidazol nie tworzy międzycząsteczkowych wiązań wodorowych z toluenem F/F | Zasada S-Pa: puryna tienylo/amina tworzy trzy międzycząsteczkowe wiązania wodorowe z pirolem -/karbaldehydem | Baza xA-T: takie samo wiązanie jak AT |
Metalowe pary zasad
W parowaniu zasad metali wiązania wodorowe Watsona-Cricka są zastępowane przez oddziaływanie między jonem metalu a nukleozydami działającymi jako ligandy. Możliwe geometrie metalu, które pozwoliłyby na utworzenie dupleksu z dwoma dwukleszczowymi nukleozydami wokół centralnego atomu metalu, to: tetraedryczny , dwunastościenny i kwadratowy płaski . Kompleksowanie metali z DNA może zachodzić poprzez tworzenie niekanonicznych par zasad z naturalnych nukleozasad z udziałem jonów metali, a także poprzez wymianę atomów wodoru wchodzących w skład par zasad Watsona-Cricka na jony metali. Wykazano, że wprowadzenie jonów metali do dupleksu DNA ma potencjalne właściwości magnetyczne, przewodzące, a także zwiększoną stabilność.
Wykazano, że między naturalnymi zasadami nukleinowymi zachodzi tworzenie kompleksów metali . Dobrze udokumentowanym przykładem jest tworzenie T-Hg-T, które obejmuje dwie zdeprotonowane tyminy , które są połączone przez Hg 2+ i tworzą połączoną parę metal-zasada. Ten motyw nie mieści ułożonych w stos Hg 2+ w dupleksie z powodu procesu tworzenia szpilki do włosów wewnątrz pasma, który jest preferowany w porównaniu z tworzeniem dupleksu. Dwie tyminy naprzeciw siebie w dupleksie nie tworzą pary zasad Watsona-Cricka w dupleksie; jest to przykład, w którym niedopasowanie par zasad Watsona-Cricka jest stabilizowane przez utworzenie pary metal-zasada. Innym przykładem kompleksowania metalu z naturalnymi zasadami nukleinowymi jest tworzenie A-Zn-T i G-Zn-C przy wysokim pH; Co +2 i Ni +2 również tworzą te kompleksy. Są to pary zasad Watsona-Cricka, w których dwuwartościowy kation jest skoordynowany z zasadami nukleotydowymi. Dokładne wiązanie jest przedmiotem dyskusji.
Opracowano wiele różnych sztucznych zasad nukleinowych do stosowania jako pary zasad metali. Te zmodyfikowane zasady nukleinowe wykazują regulowane właściwości elektroniczne, rozmiary i powinowactwa wiązania, które można zoptymalizować dla określonego metalu. Na przykład wykazano, że nukleozyd zmodyfikowany pirydyno-2,6-dikarboksylanem ściśle wiąże się z Cu 2+ , podczas gdy inne jony dwuwartościowe są tylko luźno związane. Charakter trójzębny przyczynia się do tej selektywności. Czwarte miejsce koordynacyjne na miedzi jest nasycone przeciwstawnie ułożoną zasadą nukleinową pirydyny. Asymetryczny system parowania metalowych baz jest prostopadły do par zasad Watsona-Cricka. Innym przykładem sztucznej nukleozasady są hydroksypirydonowe nukleozasady, które są zdolne do wiązania Cu 2+ wewnątrz dupleksu DNA. Pięć kolejnych par zasad miedź-hydroksypirydon włączono do podwójnej nici, która była otoczona tylko jedną naturalną zasadą nukleotydową na obu końcach. Dane EPR wykazały, że odległość między centrami miedzi oszacowano na 3,7 ± 0,1 Å, podczas gdy naturalny dupleks DNA typu B jest tylko nieznacznie większy (3,4 Å). Apel o układanie jonów metali wewnątrz dupleksu DNA jest nadzieją na uzyskanie nanoskopowych samoorganizujących się drutów metalowych, chociaż nie zostało to jeszcze zrealizowane.
Nienaturalna para zasad (UBP)
Nienaturalna para zasad (UBP) to zaprojektowana podjednostka (lub nukleozasada ) DNA , która jest tworzona w laboratorium i nie występuje w naturze. W 2012 roku grupa amerykańskich naukowców kierowana przez Floyda Romesberga, biologa chemicznego z Scripps Research Institute w San Diego w Kalifornii, opublikowała, że jego zespół zaprojektował nienaturalną parę zasad (UBP). Dwa nowe sztuczne nukleotydy lub nienaturalna para zasad (UBP) zostały nazwane d5SICS i dNaM . Mówiąc bardziej technicznie, te sztuczne nukleotydy mają właściwości hydrofobowe zasady nukleinowe , zawierają dwa skondensowane pierścienie aromatyczne , które tworzą kompleks (d5SICS – dNaM) lub parę zasad w DNA. W 2014 roku ten sam zespół z Instytutu Badawczego Scripps poinformował, że zsyntetyzował odcinek kolistego DNA znanego jako plazmid zawierający naturalne pary zasad TA i CG wraz z najlepiej działającym laboratorium UBP Romesberg, które zaprojektowało, i wstawił go do komórek wspólnego bakteria E. coli które z powodzeniem replikowały nienaturalne pary zasad przez wiele pokoleń. Jest to pierwszy znany przykład żywego organizmu przekazującego rozszerzony kod genetyczny kolejnym pokoleniom. Osiągnięto to częściowo dzięki dodaniu wspomagającego genu alg, który wykazuje ekspresję trifosforanów nukleotydów , który skutecznie importuje trifosforany zarówno d5SICSTP, jak i dNaMTP do bakterii E. coli . Następnie wykorzystują je naturalne szlaki replikacji bakterii do dokładnej replikacji plazmidu zawierającego d5SICS–dNaM.
Pomyślne włączenie trzeciej pary zasad jest znaczącym przełomem w kierunku znacznego zwiększenia liczby aminokwasów , które mogą być kodowane przez DNA, z istniejących 20 aminokwasów do teoretycznie możliwych 172, zwiększając w ten sposób potencjał żywych organizmów do produkować nowe białka . Wcześniej sztuczne łańcuchy DNA niczego nie kodowały, ale naukowcy spekulowali, że można je zaprojektować do produkcji nowych białek, które mogłyby mieć zastosowania przemysłowe lub farmaceutyczne. Transkrypcja DNA zawierającego nienaturalne pary zasad i translacja odpowiedniego mRNA zostały faktycznie osiągnięte niedawno. W listopadzie 2017 r. ten sam zespół z Scripps Research Institute , który jako pierwszy wprowadził dwie dodatkowe zasady nukleinowe do bakteryjnego DNA, poinformował, że skonstruował półsyntetyczną bakterię E. coli zdolną do wytwarzania białek przy użyciu takiego DNA. Jego DNA zawierało sześć różnych zasad nukleinowych : cztery kanoniczne i dwa sztucznie dodane, dNaM i dTPT3 (te dwa tworzą parę). Ponadto bakteria ta miała dwie odpowiednie dodatkowe zasady RNA zawarte w dwóch nowych kodonach, dodatkowe tRNA rozpoznające te nowe kodony (te tRNA zawierały również dwie nowe zasady RNA w swoich antykodonach) oraz dodatkowe aminokwasy, dzięki czemu bakterie były w stanie syntetyzować „nienaturalne” białka .
Kolejna demonstracja UBP została przeprowadzona przez grupę Ichiro Hirao w instytucie RIKEN w Japonii. W 2002 roku opracowali nienaturalną parę zasad między 2-amino-8-(2-tienylo)puryną (s) i pirydyno-2-onem (y), która działa in vitro w transkrypcji i translacji, w celu specyficznego dla miejsca włączenia aminokwasy niestandardowe w białka. W 2006 roku stworzyli 7-(2-tienylo)imidazo[4,5-b]pirydynę (Ds) i pirolo-2-karbaldehyd (Pa) jako trzecią parę zasad do replikacji i transkrypcji. Następnie odkryto Ds i 4-[3-(6-aminoheksanamido)-1-propynylo]-2-nitropirol (Px) jako parę o wysokiej wierności w amplifikacji PCR. W 2013 roku zastosowali parę Ds-Px do generowania aptamerów DNA przez in vitro (SELEX) i wykazano, że ekspansja alfabetu genetycznego znacznie zwiększa powinowactwo aptameru DNA do białek docelowych.
Układ ortogonalny
Zaproponowano i zbadano, zarówno teoretycznie, jak i eksperymentalnie, możliwość zaimplementowania systemu ortogonalnego wewnątrz komórek, niezależnego od komórkowego materiału genetycznego, w celu stworzenia całkowicie bezpiecznego systemu, z możliwym wzrostem potencjałów kodowania. Kilka grup skupiło się na różnych aspektach:
- nowe szkielety i pary zasad, jak omówiono powyżej
- Sztuczna replikacja/transkrypcja XNA ( kwasu ksenonukleinowego ) zaczynająca się generalnie od polimerazy RNA T7
- rybosomy ( sekwencje 16S ze zmienioną sekwencją anty Shine-Dalgarno umożliwiające translację tylko ortogonalnego mRNA z dopasowaną zmienioną sekwencją Shine-Dalgarno)
- nowe tRNA kodujące nienaturalne aminokwasy. Zobacz Rozszerzony kod genetyczny
Zobacz też
- Biotyna
- Ciemny wygaszacz
- Dezoksyrybozym
- Rozszerzony kod genetyczny
- Fluorofor
- Genetyka
- Biologia molekularna
- Kwas nukleinowy
- zasad jądrowych
- Nukleozyd
- Nukleotyd
- Synteza oligonukleotydów
- Rybozym
- Biologia syntetyczna
- Ksenobiologia
- xDNA
- DNA Hachimoji
- Sztucznie rozszerzony system informacji genetycznej (AEGIS)
- Kwas nukleinowy ksenonukleinowy