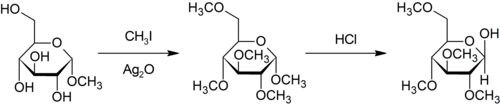Metylacja
W naukach chemicznych metylacja oznacza dodanie grupy metylowej do substratu lub zastąpienie atomu (lub grupy) grupą metylową. Metylacja jest formą alkilowania , w której grupa metylowa zastępuje atom wodoru . Terminy te są powszechnie używane w chemii , biochemii , gleboznawstwie i naukach biologicznych .
W systemach biologicznych metylacja jest katalizowana przez enzymy ; taka metylacja może być zaangażowana w modyfikację metali ciężkich , regulację ekspresji genów , regulację funkcji białek i obróbkę RNA . Metylacja in vitro próbek tkanek jest również jedną z metod zmniejszania niektórych artefaktów barwienia histologicznego . Odwrotnością metylacji jest demetylacja .
w biologii
W systemach biologicznych metylację przeprowadzają enzymy. Metylacja może modyfikować metale ciężkie, regulować ekspresję genów, przetwarzanie RNA i funkcje białek. Został on uznany za kluczowy proces leżący u podstaw epigenetyki .
Metanogeneza
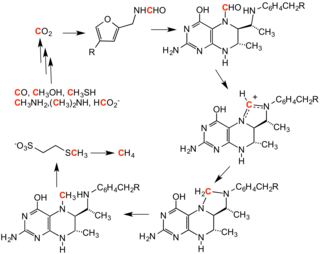
Metanogeneza , proces wytwarzania metanu z CO 2 , obejmuje szereg reakcji metylacji. Reakcje te są przeprowadzane przez zestaw enzymów występujących w rodzinie drobnoustrojów beztlenowych.
W odwrotnej metanogenezie metan służy jako czynnik metylujący. [ potrzebne źródło ]
O-metylotransferazy
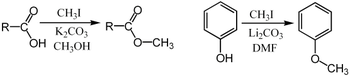
Szeroka gama fenoli ulega O-metylacji, dając pochodne anizolu . Proces ten, katalizowany przez enzymy , takie jak O-metylotransferaza kawoilo-CoA , jest kluczową reakcją w biosyntezie lignoli , prekursorów ligniny , głównego składnika strukturalnego roślin.
Rośliny wytwarzają flawonoidy i izoflawony z metylacją grup hydroksylowych, czyli wiązań metoksy . Ta 5-O-metylacja wpływa na rozpuszczalność flawonoidów w wodzie. Przykładami są 5-O-metylogenisteina , 5-O-metylomirycetyna lub 5-O-metylokwercetyna , znana również jako azalatyna.
Białka
Wraz z ubikwityną i fosforylacją metylacja jest głównym procesem biochemicznym modyfikującym funkcję białek. Najbardziej rozpowszechnione metylacje białek wpływają na reszty argininy i lizyny w określonych histonach. Inaczej histydyna, glutaminian, asparagina, cysteina są podatne na metylację. Niektóre z tych produktów obejmują S -metylocysteinę , dwa izomery N -metylohistydyny i dwa izomery N -metyloargininy.
Syntaza metioniny

Syntaza metioniny regeneruje metioninę (Met) z homocysteiny (Hcy). Ogólna reakcja przekształca 5-metylotetrahydrofolian (N 5 -MeTHF) w tetrahydrofolian (THF), jednocześnie przenosząc grupę metylową do Hcy, tworząc Met. Synteza metioniny może być zależna od kobalaminy i niezależna od kobalaminy: rośliny mają jedno i drugie, zwierzęta zależą od formy zależnej od metylokobalaminy.
W formach enzymu zależnych od metylokobalaminy reakcja przebiega dwuetapowo w reakcji ping-ponga. Enzym jest początkowo wprowadzany w stan reaktywny przez przeniesienie grupy metylowej z N 5 -MeTHF do Co(I) w kobalaminie związanej z enzymem (Cob), tworząc metylokobalaminę (Me-Cob), która teraz zawiera Me-Co (III) i aktywacja enzymu. Następnie Hcy, który skoordynował się z cynkiem związanym z enzymem , tworząc reaktywny tiolan, reaguje z Me-Cob. Aktywowana grupa metylowa jest przenoszona z Me-Cob do tiolanu Hcy, który regeneruje Co(I) w Cob, a Met jest uwalniany z enzymu.
Metale ciężkie: arsen, rtęć, kadm
Biometylacja to droga przekształcania niektórych ciężkich pierwiastków w bardziej mobilne lub śmiercionośne pochodne, które mogą dostać się do łańcucha pokarmowego . Biometylacja związków arsenu rozpoczyna się od powstania metanoarsonianów . Tak więc trójwartościowe nieorganiczne związki arsenu są metylowane z wytworzeniem metanoarsonianu. Donorem grupy metylowej jest S-adenozylometionina . Metanoarsoniany są prekursorami dimetyloarsonianów, ponownie w cyklu redukcji (do kwasu metyloarsonowego), po którym następuje druga metylacja. Powiązane ścieżki mają zastosowanie do biosynteza metylortęci . _
Metylacja epigenetyczna
Metylacja DNA/RNA
Metylacja DNA u kręgowców zazwyczaj występuje w miejscach CpG (miejscach cytozyna-fosforan-guanina – to znaczy, gdzie bezpośrednio po cytozynie następuje guanina w sekwencji DNA). Ta metylacja powoduje konwersję cytozyny do 5-metylocytozyny . Tworzenie Me-CpG jest katalizowane przez enzym metylotransferazę DNA . U ssaków metylacja DNA jest powszechna w komórkach organizmu, a metylacja miejsc CpG wydaje się być domyślna. Ludzkie DNA ma około 80-90% zmetylowanych miejsc CpG, ale są pewne obszary, znane jako Wyspy CpG , które są bogate w CG (wysoka zawartość cytozyny i guaniny, składają się z około 65% reszt CG ), z których żadna nie jest metylowana. Są one związane z promotorami 56% genów ssaków, w tym wszystkich wszechobecnych genów . Jeden do dwóch procent ludzkiego genomu to klastry CpG i istnieje odwrotna zależność między metylacją CpG a aktywnością transkrypcyjną. Metylacja przyczyniająca się do dziedziczenia epigenetycznego może zachodzić poprzez metylację DNA lub metylację białek. Niewłaściwa metylacja ludzkich genów może prowadzić do rozwoju chorób, w tym raka. Podobnie metylacja RNA występuje w różnych gatunkach RNA, mianowicie. tRNA , rRNA , mRNA , tmRNA , snRNA , snoRNA , miRNA i wirusowego RNA. Różne strategie katalityczne są stosowane do metylacji RNA przez różne RNA-metylotransferazy. Uważa się, że metylacja RNA istniała przed metylacją DNA we wczesnych formach życia ewoluujących na Ziemi.
N6-metyladenozyna (m6A) jest najpowszechniejszą i najbardziej rozpowszechnioną modyfikacją metylacyjną w cząsteczkach RNA (mRNA) obecnych u eukariontów. 5-metylocytozyna (5-mC) również powszechnie występuje w różnych cząsteczkach RNA. Ostatnie dane zdecydowanie sugerują, że metylacja m6A i 5-mC RNA wpływa na regulację różnych procesów biologicznych, takich jak stabilność RNA i translacja mRNA, oraz że nieprawidłowa metylacja RNA przyczynia się do etiologii chorób człowieka.
Metylacja białek
Metylacja białka zwykle zachodzi na resztach aminokwasowych argininy lub lizyny w sekwencji białka. Arginina może być metylowana raz (monometylowana arginina) lub dwukrotnie, obiema grupami metylowymi na jednym końcowym azocie ( asymetryczna dimetyloarginina ) lub jedną na obu atomach azotu (symetryczna dimetyloarginina), przez białkowe metylotransferazy argininowe (PRMT). Lizyna może być metylowana raz, dwa lub trzy razy przez metylotransferazy lizyny. Metylacja białek była najczęściej badana w histonach . Przenoszenie metylu grupy od S-adenozylometioniny do histonów jest katalizowana przez enzymy znane jako metylotransferazy histonowe . Histony, które są metylowane na niektórych resztach, mogą działać epigenetycznie , represjonując lub aktywując ekspresję genów. Metylacja białek jest jednym z rodzajów modyfikacji potranslacyjnych .
Ewolucja
Metabolizm metylu jest bardzo stary i można go znaleźć we wszystkich organizmach na ziemi, od bakterii po ludzi, co wskazuje na znaczenie metabolizmu metylu dla fizjologii. Rzeczywiście, farmakologiczne hamowanie globalnej metylacji u różnych gatunków, od ludzi, myszy, ryb, much, glisty, roślin, alg i sinic, ma taki sam wpływ na ich rytmy biologiczne, wykazując konserwatywne fizjologiczne role metylacji podczas ewolucji.
w chemii
Termin metylowanie w chemii organicznej odnosi się do procesu alkilowania stosowanego do opisania dostarczania grupy CH3 .
Metylacja elektrofilowa
Metylacje są zwykle przeprowadzane przy użyciu elektrofilowych źródeł metylu, takich jak jodometan , siarczan dimetylu , węglan dimetylu lub chlorek tetrametyloamoniowy . Mniej powszechne, ale silniejsze (i bardziej niebezpieczne) odczynniki metylujące obejmują triflat metylu , diazometan i fluorosulfonian metylu ( magiczny metyl ). Wszystkie te odczynniki reagują poprzez podstawienia nukleofilowe SN2 . Na przykład karboksylan można metylować tlenem z wytworzeniem estru metylowego ; sól alkoholanu RO - może być podobnie metylowana ROCH z wytworzeniem eteru 3 ; lub enolan ketonu może być metylowany na węglu w celu wytworzenia nowego ketonu .
Metylacja Purdiego jest specyficzna dla metylacji węglowodanów przy użyciu tlenu przy użyciu jodometanu i tlenku srebra .
Metylacja Eschweilera-Clarke'a
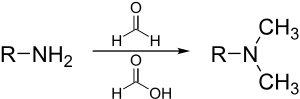
Eschweilera -Clarke'a jest metodą metylacji amin . Ta metoda pozwala uniknąć ryzyka czwartorzędowania , które występuje, gdy aminy są metylowane halogenkami metylu.
Diazometan i trimetylosililodiazometan
Diazometan i bezpieczniejszy analog trimetylosililodiazometanu metylanu , kwasów karboksylowych, fenoli, a nawet alkoholi:
Sposób ma tę zaletę, że produkty uboczne można łatwo usunąć z mieszaniny produktów.
Metylacja nukleofilowa
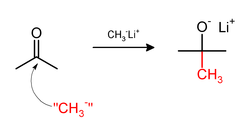
Metylacja czasami wymaga użycia nukleofilowych odczynników metylowych. CH3MgX Silnie nukleofilowe środki metylujące obejmują metylolit ( CH3Li bromek , ) lub odczynniki Grignarda takie jak metylomagnezu ( ). Na przykład CH 3 Li doda grupy metylowe do grupy karbonylowej (C=O) ketonów i aldehydów.:
Łagodniejsze środki metylujące obejmują tetrametylocynę , dimetylocynk i trimetyloglin .
Zobacz też
Tematy z biologii
- Sekwencjonowanie wodorosiarczynowe - metoda biochemiczna stosowana do określania obecności lub braku grup metylowych w sekwencji DNA
- Baza danych metylacji DNA MethDB
- Termoforeza w mikroskali – biofizyczna metoda określania stanu metylacji DNA
Tematyka chemii organicznej
- alkilowanie
- metoksy
- Metylenowanie tytanu i cynku
- odczynnik Petasisa
- odczynnik Nysteda
- Reakcja Wittiga
- odczynnik Tebbego
Linki zewnętrzne
- deltaMasses Wykrywanie metylacji po spektrometrii mas