kompleks SCF
Skp, Cullin, F-box zawierający kompleks (lub kompleks SCF ) to wielobiałkowy kompleks ligazy ubikwityny E3 , który katalizuje ubikwitynację białek przeznaczonych do degradacji proteasomów 26S . Wraz z kompleksem promującym anafazę SCF odgrywa ważną rolę w ubikwitynacji białek zaangażowanych w cykl komórkowy. Kompleks SCF oznacza również różne inne białka komórkowe do zniszczenia.
Podstawowe komponenty
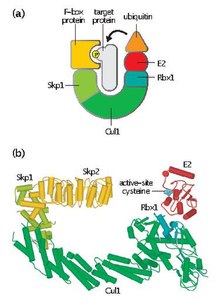
SCF zawiera zmienne białko F-box i trzy podstawowe podjednostki:
- Białko F-box (FBP) – FBP przyczynia się do specyficzności substratowej kompleksu SCF poprzez najpierw agregację do białek docelowych niezależnie od kompleksu. Każdy FBP (np. Skp2) może rozpoznawać kilka różnych substratów w sposób zależny od modyfikacji potranslacyjnych, takich jak fosforylacja lub glikozylacja. FBP następnie wiąże się ze Skp1 kompleksu SCF przy użyciu motywu F-box, zbliżając docelowe białko do funkcjonalnego enzymu sprzęgającego ubikwitynę E2. FBP jest również niezbędny w regulacji aktywności SCF w trakcie cyklu komórkowego. Uważa się, że poziomy SCF pozostają stałe przez cały cykl komórkowy. Zamiast tego powinowactwo FBP do substratów białkowych jest regulowane przez fosforylację docelowych białek za pośrednictwem cykliny-CDK.
- Skp1 – Skp1 jest białkiem adaptorowym, które jest niezbędne do rozpoznawania i wiązania białek F-box.
- Cullin ( CUL1 ) – Cullin tworzy główne rusztowanie strukturalne kompleksu SCF i łączy domenę skp1 z domeną Rbx1. Różne kombinacje Cullina i FBP mogą generować setki typów ligaz ubikwitynowych E3, które celują w różne substraty.
- RBX1 – Rbx1 zawiera małą, wiążącą cynk domenę palcową naprawdę interesującego nowego genu (RING), z którą wiąże się enzym koniugujący ubikwitynę E2. To zdarzenie wiązania umożliwia przeniesienie ubikwityny z E2 na resztę lizyny na docelowym białku.
Odkrycie
Pierwsza wskazówka, która doprowadziła do odkrycia kompleksu SCF, pochodziła z ekranów genetycznych Saccharomyces cerevisiae , znanych również jako pączkujące drożdże. Wrażliwe na temperaturę mutanty cyklu podziału komórkowego (Cdc) - takie jak Cdc4, Cdc34 i Cdc53 - zatrzymane w G1 z niereplikowanym DNA i wieloma wydłużonymi pąkami. Fenotyp przypisywano niepowodzeniu degradacji Sic1, inhibitora kompleksów S cyklina-CDK. Odkrycia te wskazują, że proteoliza jest ważna w przejściu G1/S.
Następnie badania biochemiczne wykazały, że Cdc34 jest enzymem E2, który fizycznie oddziałuje z kompleksem ligazy ubikwitynowej E3 zawierającym Skp1, Cdc4 i kilka innych białek. Stwierdzono, że znani partnerzy wiążący Skp1 - w szczególności Skp2, cyklina F i Cdc4 - mają wspólny motyw około 40 reszt, który został wymyślony jako motyw F-box. Hipoteza F-box, która nastąpiła po tych odkryciach, sugerowała, że białka F-box rekrutują substraty przeznaczone do degradacji, a Skp1 łączy białko F-box z rdzeniowym kompleksem ubikwitynacji.
Późniejsze badania genetyczne Caenorhabditis elegans przyczyniły się później do wyjaśnienia innych składników kompleksu SCF.
Regulacja cyklu komórkowego
Cykl komórki eukariotycznej jest regulowany poprzez syntezę, degradację, interakcje wiązania, potranslacyjne modyfikacje białek regulatorowych. Spośród tych białek regulatorowych dwie ligazy ubikwitynowe są kluczowe dla przejścia przez punkty kontrolne cyklu komórkowego. Kompleks promujący anafazę (APC) kontroluje przejście metafaza-anafaza, podczas gdy kompleks SCF kontroluje przejścia G1/S i G2/M. W szczególności wykazano, że SCF reguluje podział centrioli od późnej telofazy do przejścia G1/S.
Aktywność SCF jest w dużej mierze regulowana przez modyfikacje potranslacyjne. Na przykład autokatalityczna degradacja FBP za pośrednictwem ubikwityny jest mechanizmem zmniejszania aktywności SCF.
Dobrze scharakteryzowane substraty cyklu komórkowego kompleksów SCF obejmują:
- białka rodziny cyklin: cyklina D, cyklina E
- regulatory transkrypcji: Myc, E2f1, p130
- inhibitory kinazy cyklinozależnej (CKI): p27 Kip1 , p21, Wee1
- białka centriolowe: Cep250, Ninein
Istnieje około siedemdziesięciu ludzkich FBP, z których kilka bierze udział w kontroli cyklu komórkowego jako składnik kompleksów SCF.
Skp2 to FBP, który wiąże CKI, takie jak p27 Kip1 i p21. Skp2 wiąże p27 Kip1 tylko wtedy, gdy spełnione są dwa warunki: p27 Kip1 jest fosforylowany przez E/A/CKD2 i związany z Cks1. W wyniku wiązania Skp2, p27 Kip1 jest ubikwitynowany i ukierunkowany na degradację w późnym G1 i wczesnym S. SCF-Skp2 celuje również w p130 w celu degradacji w sposób zależny od fosforylacji.
Białko zawierające powtórzenia beta-transducyny (βTRCP) to FBP, którego celem jest emi1 - inhibitor APC / C-Cdh1 - i wee1 w celu degradacji podczas wczesnej mitozy. βTRCP rozpoznaje te substraty po ich fosforylacji przez kinazę Polo-podobną 1 lub cyklinę B-CDK1.
Fbw7, który jest ludzkim homologiem cdc4 w drożdżach, jest FBP, którego celem jest degradacja cykliny E, Myc, Notch i c-Jun. Fbw7 jest stabilny przez cały cykl komórkowy i jest zlokalizowany w jądrze ze względu na obecność sekwencji lokalizacji jądrowej (NLS). SCF-Fbw7 celuje w Sic1 - gdy co najmniej sześć z dziewięciu możliwych miejsc jest ufosforylowanych - i Swi5 w celu degradacji. Ponieważ Sic1 zwykle zapobiega przedwczesnemu wejściu w fazę S poprzez hamowanie cykliny B-CDK1, celowanie w Sic1 w celu degradacji sprzyja wejściu w fazę S. Wiadomo, że Fbw7 jest haplo-niewystarczającym genem supresorowym guza zaangażowanym w kilka sporadycznych raków, dla których wystarczy jeden zmutowany allel, aby zakłócić fenotyp typu dzikiego.
Fbxo4 to kolejny supresor guza FBP, który jest zaangażowany w ludzkie raki. SCF-fbxo4 odgrywa rolę w kontroli cyklu komórkowego, kierując cyklinę D1 w celu degradacji.
Cyklina F jest FBP, która jest związana ze stwardnieniem zanikowym bocznym (ALS) i otępieniem czołowo-skroniowym (FTD). Mutacje, które zapobiegają fosforylacji cykliny F, zmieniają aktywność SCF-cykliny F, co prawdopodobnie wpływa na dalsze procesy związane z degeneracją neuronów w ALS i FTD. Zwykle cyklina F celuje w E2f1 w celu degradacji.
Rak
Ostatnio kompleksy SCF stały się atrakcyjnym celem przeciwnowotworowym ze względu na ich regulację w górę w niektórych ludzkich nowotworach i ich biochemicznie odrębne miejsca aktywne. Chociaż wiele z wyżej wymienionych FBP jest związanych z rakiem, cytotoksyczność jest czynnikiem ograniczającym opracowywanie leków.
Antysensowne oligonukleotydy i siRNA ukierunkowane na Skp2 są w trakcie opracowywania leków. Wstępne badania wykazały, że regulacja w dół Skp2 może hamować wzrost czerniaków, komórek raka płuc, komórek raka jamy ustnej i komórek glejaka .
Wykazano, że siRNA ukierunkowane na βTRCP uwrażliwiają komórki raka piersi i komórki raka szyjki macicy na istniejące chemioterapie.
Sygnalizacja hormonów roślinnych
Hormon roślinny auksyna wiąże Tir1 (odpowiedź na inhibitor transportu 1). Tir1 jest białkiem F-box sygnalizującym auksynę (AFB), które działa jako receptor auksyny. Tir1 związany z auksyną stymuluje wiązanie SCF-Tir1 z represorem AUX/IAA. Późniejsza degradacja represora skutkuje aktywacją genów AUX/IAA (tj. reagujących na auksynę).
Hormon roślinny Jasmonate wiąże Coi1, FBP. SCF-Coi1 następnie wiąże czynnik transkrypcyjny JAZ i kieruje go do degradacji. Degradacja czynnika transkrypcyjnego JAZ umożliwia transkrypcję genów reagujących na jasmonian.
- ^ abc młody ; Ou, Rattner, JB (2004), „Centrosom w organizmach wyższych: struktura, skład i duplikacja”, International Review of Cytology , Elsevier, 238 : 119–182, doi : 10.1016/s0074-7696 (04) 38003-4 , ISBN 978-0-12-364642-2 , PMID 15364198
- ^ a b c d e Fischer, Martin; Dang, Chi V.; DeCaprio, James A. (2018), „Kontrola podziału komórek”, Hematologia , Elsevier, s. 176–185, doi : 10.1016/b978-0-323-35762-3.00017-2 , ISBN 978-0-323-35762 -3
- ^ Morgan, David „Degradacja białek w kontroli cyklu komórkowego”, The Cell Cycle; Zasady kontroli 2007
- ^ a b c d Molekularne podstawy raka . Elsevier. 2008. doi : 10.1016/b978-1-4160-3703-3.x5001-7 . ISBN 978-1-4160-3703-3 .
- Bibliografia Linki _ _ _ _ _ _ _ _ _ 045382-8 , pobrano 2019-12-01 zewnętrzne
- ^ a b c Patton, E (1998-06-01). „Kontrola kombinatoryczna w proteolizie zależnej od ubikwityny: nie odrzucaj hipotezy F-box”. Trendy w genetyce . 14 (6): 236–243. doi : 10.1016/s0168-9525(98)01473-5 . ISSN 0168-9525 . PMID 9635407 .
- ^ Schwob, E (1994-10-21). „Inhibitor kinazy cyklinowej typu B p40SIC1 kontroluje przejście G1 do S w S. cerevisiae”. komórka . 79 (2): 233–244. doi : 10.1016/0092-8674(94)90193-7 . ISSN 0092-8674 . PMID 7954792 . S2CID 34939988 .
- ^ ab Willems, Andrew R.; Schwab, Michael; Tyers, Mike (listopad 2004). „Przewodnik autostopowicza po ligazach ubikwityny kullina: SCF i jego krewni” . Biochimica et Biophysica Acta (BBA) - Badania nad komórkami molekularnymi . 1695 (1–3): 133–170. doi : 10.1016/j.bbamcr.2004.09.027 . ISSN 0167-4889 . PMID 15571813 .
- ^ Vodermaier, Hartmut C. (wrzesień 2004). „APC / C i SCF: wzajemna kontrola i cykl komórkowy” . Bieżąca biologia . 14 (18): R787–R796. doi : 10.1016/j.cub.2004.09.020 . ISSN 0960-9822 . PMID 15380093 .
-
^
Lambrus, Bramwell G.; Moyer, Tyler C.; Holandia, Andrew J. (31.08.2017). „Zastosowanie systemu degradacji indukowanej auksyną (AID) do szybkiego wyczerpywania się białek w komórkach ssaków” . doi : 10.1101/182840 .
{{ cite journal }}: Cite journal wymaga|journal=( pomoc ) - ^ Frescas, Dawid; Pagano, Michele (czerwiec 2008). „Deregulacja proteolizy przez białka F-box SKP2 i β-TrCP: przechylanie szali raka” . Nature Recenzje Rak . 8 (6): 438–449. doi : 10.1038/nrc2396 . ISSN 1474-175X . PMC 2711846 . PMID 18500245 .
- Bibliografia _ Steussy, C. Nic; Goebl, Mark G. (13.02.1998). „Niezbędna domena w Cdc34p jest wymagana do wiązania się z kompleksem zawierającym Cdc4p i Cdc53p in Saccharomyces cerevisiae” . Dziennik Chemii Biologicznej . 273 (7): 4040–4045. doi : 10.1074/jbc.273.7.4040 . ISSN 0021-9258 . PMID 9461595 .
- ^ Blondel, M. (15.11.2000). „Degradacja Far1 specyficzna dla jądra jest kontrolowana przez lokalizację białka F-box Cdc4” . Dziennik EMBO . 19 (22): 6085–6097. doi : 10.1093/emboj/19.22.6085 . ISSN 1460-2075 . PMC 305831 . PMID 11080155 .
- Bibliografia _ Ikeda, A.; Koyama, N.; Fukada, J.; Nagao, R. (2008-09-11). „Wyrafinowany system dwuhybrydowy ujawnia, że zależna od SCFCdc4 degradacja Swi5 przyczynia się do mechanizmu regulacyjnego wejścia w fazę S” . Obrady Narodowej Akademii Nauk . 105 (38): 14497–14502. Bibcode : 2008PNAS..10514497K . doi : 10.1073/pnas.0806253105 . ISSN 0027-8424 . PMC 2567208 . PMID 18787112 .
- Bibliografia _ Jones, Jessa B.; Ashfaq, Raheela; Adsay, Volkan; Baker, Suzanne J.; Walentynki, Wirginia; Hempen, Paula M.; Hilgers, Werner; Yeo, Charles J.; Hruban, Ralph H.; Kern, Scott E. (październik 2003). „BRAF i FBXW7 (CDC4, FBW7, AGO, SEL10) Mutacje w odrębnych podzbiorach raka trzustki” . Amerykański Dziennik Patologii . 163 (4): 1255–1260. doi : 10.1016/s0002-9440(10)63485-2 . ISSN 0002-9440 . PMC 1868306 . PMID 14507635 .
- Bibliografia _ Nakagawa, Tadashi; Morohoshi, Akane; Nakagawa, Makiko; Ishida, Noriko; Suzuki, Naoki; Aoki, Masashi; Nakayama, Keiko (30.09.2019). „Patogenne mutacje w genie ALS CCNF powodują błędną lokalizację cykliny F w cytoplazmie i podwyższoną aktywność ATPazy VCP” . Genetyka molekularna człowieka . 28 (20): 3486–3497. doi : 10.1093/hmg/ddz119 . ISSN 0964-6906 . PMID 31577344 .
- ^ b Lee , Albert; Rayner, Stephanie L.; De Luca, Alana; Gwee, Serene SL; Morsch, Marco; Sundaramoorthy, Vinod; Shahheydari, Hamideh; Ragagnin, Audrey; Shi, Bingyang; Yang, Shu; Williams, Kelly L. (październik 2017). „Fosforylacja kinazy kazeinowej II cykliny F w serynie 621 reguluje aktywność ligazy E3 ubikwitylacji Lys48 kompleksu SCF (cyklina F)” . Biologia otwarta . 7 (10): 170058. doi : 10.1098/rsob.170058 . ISSN 2046-2441 . PMC 5666078 . PMID 29021214 .
- ^ Skaar, Jeffrey R.; Pagan, Julia K.; Pagano, Michele (grudzień 2014). „Terapie ukierunkowane na ligazę ubikwitynową SCF” . Nature Recenzje Odkrywanie leków . 13 (12): 889–903. doi : 10.1038/nrd4432 . ISSN 1474-1776 . PMC 4410837 . PMID 25394868 .
- ^ a b c Słońce, L. Jia i Y. (28.02.2011). „SCF E3 ligazy ubikwitynowe jako cele przeciwnowotworowe” . Aktualne cele leków przeciwnowotworowych . 11 (3): 347–356. doi : 10.2174/156800911794519734 . PMC 3323109 . PMID 21247385 .
- ^ Dharmasiri, Nihal; Dharmasiri, Sunethra; Estelle, Mark (maj 2005). „Białko F-box TIR1 jest receptorem auksyny”. Natura . 435 (7041): 441–445. Bibcode : 2005Natur.435..441D . doi : 10.1038/natura03543 . ISSN 0028-0836 . PMID 15917797 . S2CID 4428049 .
- ^ Devoto, Alessandra; Nieto-Rostro, Manuela; Xie, Daoxin; Ellis, Krystyna; Harmston, Rebecca; Patryk, Elaine; Davis, Jackie; Sherratt, Leigh; Coleman, Mark; Turner, John G. (listopad 2002). „COI1 łączy sygnalizację i płodność jasmonianu z kompleksem SCF ubikwityna-ligaza u Arabidopsis”. Dziennik roślin . 32 (4): 457–466. doi : 10.1046/j.1365-313x.2002.01432.x . ISSN 0960-7412 . PMID 12445118 .