Dehydrogenaza sarkozyny
| Identyfikatory | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| dehydrogenazy sarkozyny | |||||||||
| nr WE | 1.5.8.3 | ||||||||
| nr CAS | 37228-65-2 | ||||||||
| Bazy danych | |||||||||
| IntEnz | Widok IntEnz | ||||||||
| BRENDA | Wpis BRENDY | ||||||||
| ExPASy | Widok NiceZyme | ||||||||
| KEGG | Wpis KEGG | ||||||||
| MetaCyc | szlak metaboliczny | ||||||||
| PRYM | profil | ||||||||
| Struktury PDB | RCSB PDB PDBe PDB suma | ||||||||
| Ontologia genów | AmiGO / QuickGO | ||||||||
| |||||||||
W enzymologii dehydrogenaza sarkozyny ( EC 1.5.8.3 ) jest enzymem mitochondrialnym , który katalizuje reakcję chemiczną N-demetylacji sarkozyny z wytworzeniem glicyny . Enzym ten należy do rodziny oksydoreduktaz , w szczególności działających na donorową grupę CH-NH z innymi akceptorami. Systematyczna nazwa tej klasy enzymów to sarkozyna: oksydoreduktaza akceptorowa (demetylująca) . Inne powszechnie używane nazwy to N-demetylaza sarkozyny , dehydrogenaza monometyloglicyny i sarkozyna: (akceptor) oksydoreduktaza (demetylująca) . Dehydrogenaza sarkozyny jest blisko spokrewniona z dehydrogenazą dimetyloglicyny , która katalizuje reakcję demetylacji dimetyloglicyny do sarkozyny . Zarówno dehydrogenaza sarkozyny, jak i dehydrogenaza dimetyloglicyny wykorzystują FAD jako kofaktor. Dehydrogenaza sarkozyny jest połączona przez flawoproteinę przenoszącą elektrony (ETF) do oddechowego łańcucha redoks. Ogólna reakcja chemiczna katalizowana przez dehydrogenazę sarkozynową to:
- sarkozyna + akceptor + H 2 O glicyna + formaldehyd + zredukowany akceptor
Struktura
Nie ma struktury krystalicznej dostępnej dla dehydrogenazy sarkozyny. Dehydrogenaza sarkozyny zawiera kowalencyjnie związaną grupę FAD „połączoną poprzez pozycję 8 alfa pierścienia izoalloksazyny z imidazolem N(3) reszty histydyny”. Enzym, według Freisell Wr. i wsp., zawiera również żelazo niehemowe w stosunku 1 lub 2 żelaza na 300 000 g enzymu oraz 0,5 mola siarki rozpuszczalnej w kwasie, co sugeruje, że przeniesienie elektronu podczas pierwszego etapu reakcji może przebiegać inną drogą niż ta klastrów Fe-S.
Mechanizm

Dehydrogenaza sarkozyny, której substratem jest sarkozyna, jest zgodna z kinetyką Michaelisa-Mentena i ma Km 0,5 mM i Vmax 16 mmol/h/mg białka. Enzym jest hamowany kompetycyjnie przez kwas metoksyoctowy, którego Ki wynosi 0,26 mM
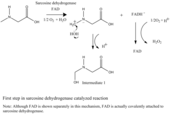
Dokładny mechanizm działania dehydrogenazy sarkozyny nie jest znany. Jednak zgodnie z ogólną reakcją netto omówioną w Honova.E, et al. papier:
- Sarkozyna + H 2 O + O 2 → glicyna + formaldehyd + H 2 O 2
pierwszy etap reakcji może obejmować przeniesienie wodorku z grupy N-metylowej sarkozyny na FAD, umożliwiając H2O atakowanie karbokationu w celu utworzenia związku pośredniego 1 (patrz rysunek 1). Nie ma etapu deaminacji. Zamiast tego demetylacja grupy N-metylowej na sarkozynie zachodzi bezpośrednio. Zredukowany FADH − z pierwszego etapu jest następnie utleniany przez O 2 z wytworzeniem H 2 O 2 .
Demetylacja sarkozyny katalizowana przez dehydrogenazę sarkozyny może przebiegać z lub bez obecności tetrahydrofolianu . Jednak w warunkach beztlenowych i bez tetrahydrofolianu po N-demetylacji sarkozyny powstaje wolny formaldehyd. Reakcja z 1 molem sarkozyny i 1 molem FAD w tych warunkach daje 1 mol glicyny i 1 mol formaldehydu (mechanizm patrz ryc. 2).
W obecności tetrahydrofolianu dehydrogenaza sarkozyny wiąże się z tetrahydrofolianem i przekształca tetrahydrofolian w 5,10-metylenotetrahydrofolian. Tetrahydrofolian służy tutaj jako akceptor węgla 1 podczas procesu demetylacji (mechanizm patrz ryc. 3).
Funkcjonować
Dehydrogenaza sarkozyny jest jednym z enzymów metabolizmu sarkozyny, który katalizuje demetylację sarkozyny do glicyny . Poprzedza ją dehydrogenaza dimetyloglicyny , która przekształca dimetyloglicynę w sarkozynę. Glicyna może być również przekształcona w sarkozynę przez N-metylotransferazę glicyny . Ponieważ glicyna odpowiada za produkcję reakcji katalizowanej przez dehydrogenazę sarkozyny, oprócz metabolizmu sarkozyny, enzym ten jest również pośrednio związany z kreatyną cyklu oddechowego i łańcucha oddechowego w mitochondriach (ścieżka patrz ryc. 4). Mimo to biologiczne znaczenie dehydrogenazy sarkozyny poza metabolizmem sarkozyny nie jest do końca znane. W badaniu dziedzicznej hemochromatozy z użyciem zarówno myszy typu dzikiego, jak i myszy z niedoborem HFE (genu) , karmionych dietą z dodatkiem 2% karbonylożelaza, wykazano, że dehydrogenaza sarkozyny jest obniżona u myszy z niedoborem HFE, ale rola dehydrogenazy sarkozyny w metabolizmie żelaza jest nieznana od przeprowadzony eksperyment.
Znaczenie choroby
Sarkozynemia
Sarkozynemia jest chorobą autosomalną recesywną spowodowaną mutacją genu dehydrogenazy sarkozyny w locus genu 9q33-q34. Prowadzi to do upośledzonego metabolizmu sarkozyny i powoduje gromadzenie się sarkozyny we krwi i moczu, stan zwany sarkozynemią .
Rak prostaty
Oprócz sarkozynemii, dehydrogenaza sarkozyny wydaje się również odgrywać rolę w procesie progresji raka prostaty . Stężenie sarkozyny wraz z uracylem , kinureniną , 3-fosforanem glicerolu , leucyną i proliną wzrasta wraz z postępem raka prostaty . Zatem sarkozyna może być wykorzystana jako potencjalny biomarker do wykrywania raka prostaty i mierzenia postępu choroby. Jak pokazuje artykuł Sreekumar, A. i wsp., usunięcie dehydrogenazy sarkozyny z łagodnych komórek nabłonka prostaty zwiększa stężenie sarkozyny i zwiększa inwazję komórek nowotworowych, podczas gdy usunięcie dehydrogenazy dimetyloglicyny lub N-metylotransferazy glicyny w komórkach raka prostaty zmniejsza inwazję komórek. Pokazuje to, że metabolizm sarkozyny odgrywa kluczową rolę w inwazji i migracji komórek raka prostaty. Badanie Sreekumara sugeruje, że dehydrogenaza sarkozyny i inne enzymy w szlakach metabolizmu sarkozyny mogą być potencjalnymi celami terapeutycznymi raka prostaty. Jednak badanie przeprowadzone przez Jentzmika F. i in. analizując poziom sarkozyny u 92 pacjentów z rakiem prostaty dochodzi do innego wniosku: sarkozyna nie może być stosowana jako wskaźnik i biomarker raka prostaty.
Zobacz też
Dalsza lektura
- FRISELL WR, MACKENZIE CG (1962). „Rozdzielanie i oczyszczanie dehydrogenazy sarkozyny i dehydrogenazy dimetyloglicyny” . J. Biol. chemia . 237 : 94–8. doi : 10.1016/S0021-9258(18)81367-9 . PMID 13895406 .
- HOSKINS DD, MACKENZIE CG (1961). „Wymóg solubilizacji i transferu elektronów flawoprteiny mitochondrialnej dehydrogenazy sarkozyny i dehydrogenazy dimetyloglicyny” . J. Biol. chemia . 236 : 177–83. doi : 10.1016/S0021-9258(18)64450-3 . PMID 13716069 .