fotoliaza
| Kryptochrom / fotoliaza, C-końcowe wiązanie FAD | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
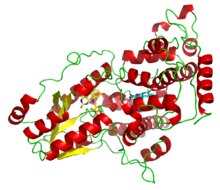 Fotoliaza deazaflawiny z Anacystis nidulans , ilustrująca dwa kofaktory zbierające światło: FADH - (żółty) i 8-HDF (cyjan).
| |||||||||||
| Identyfikatory | |||||||||||
| Symbol | FAD_binding_7 | ||||||||||
| Pfam | PF03441 | ||||||||||
| InterPro | IPR005101 | ||||||||||
| PROZYTA | PDOC00331 | ||||||||||
| SCOP2 | 1qnf / ZAKRES / SUPFAM | ||||||||||
| |||||||||||
| fotoliaza dezoksyrybodipirymidyny (CPD) | |||||||||
|---|---|---|---|---|---|---|---|---|---|
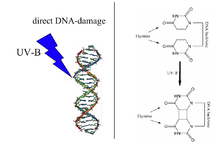 Dimer cyklobutanu tyminy i tyminy wywołany promieniowaniem UV (po prawej) to rodzaj uszkodzenia DNA , które jest naprawiane przez fotoliazę DNA. Uwaga: Powyższy diagram jest błędnie oznaczony jako tymina, ponieważ w strukturach brakuje grup 5-metylowych.
| |||||||||
| Identyfikatory | |||||||||
| nr WE | 4.1.99.3 | ||||||||
| nr CAS | 37290-70-3 | ||||||||
| Bazy danych | |||||||||
| IntEnz | Widok IntEnz | ||||||||
| BRENDA | Wpis BRENDY | ||||||||
| ExPASy | Widok NiceZyme | ||||||||
| KEGG | Wpis KEGG | ||||||||
| MetaCyc | szlak metaboliczny | ||||||||
| PRYM | profil | ||||||||
| Struktury PDB | RCSB PDB PDBe PDB suma | ||||||||
| Ontologia genów | AmiGO / QuickGO | ||||||||
| |||||||||
Fotoliazy ( EC 4.1.99.3 ) to enzymy naprawiające DNA , które naprawiają uszkodzenia spowodowane ekspozycją na światło ultrafioletowe . Enzymy te wymagają światła widzialnego (z fioletowo-niebieskiego końca widma) zarówno do własnej aktywacji, jak i do właściwej naprawy DNA. Mechanizm naprawy DNA z udziałem fotoliaz nazywa się fotoreaktywacją. Przekształcają głównie dimery pirymidynowe w normalną parę zasad pirymidynowych.
Funkcjonować
Fotoliazy wiążą komplementarne nici DNA i rozbijają pewne typy dimerów pirymidynowych , które powstają, gdy para zasad tyminy lub cytozyny na tej samej nici DNA zostaje połączona kowalencyjnie . Długość wiązania tej dimeryzacji jest krótsza niż długość wiązania normalnej struktury B-DNA, która tworzy nieprawidłową matrycę do replikacji i transkrypcji. Bardziej powszechne wiązanie kowalencyjne obejmuje tworzenie cyklobutanu most. Fotoliazy mają duże powinowactwo do tych uszkodzeń i odwracalnie wiążą je i przekształcają z powrotem w pierwotne zasady.
Ewolucja
Fotoliaza jest filogenetycznie starym enzymem, który jest obecny i funkcjonalny u wielu gatunków, od bakterii , przez grzyby , rośliny i zwierzęta . Fotoliaza jest szczególnie ważna w naprawie uszkodzeń roślin wywołanych promieniowaniem UV. Mechanizm fotoliazy nie działa już u ludzi i innych ssaków łożyskowych, które zamiast tego polegają na mniej wydajnym mechanizmie naprawy wycinania nukleotydów , chociaż zachowują wiele kryptochromów .
Fotoliazy są flawoproteinami i zawierają dwa kofaktory zbierające światło . Wiele fotoliaz ma domenę N-końcową , która wiąże drugi kofaktor. Wszystkie fotoliazy zawierają zredukowany dwuelektronowo FADH − ; są one podzielone na dwie główne klasy w oparciu o drugi kofaktor, którym może być pteryny (MTHF) w fotoliazach folianowych lub deazaflawina 8-hydroksy-7,8-didemetylo-5-deazariboflawina (8-HDF) w fotoliazach deazaflawinowych . Chociaż do aktywności katalitycznej wymagany jest tylko FAD, drugi kofaktor znacznie przyspiesza szybkość reakcji w warunkach słabego oświetlenia. Enzym działa na zasadzie transferu elektronów , w którym zredukowana flawina FADH − jest aktywowana przez energię świetlną i działa jako donor elektronów w celu rozbicia dimeru pirymidyny.
Na podstawie podobieństwa sekwencji fotoliazy DNA można podzielić na kilka klas:
|
- Fotoliazy CPD klasy 1 to enzymy, które przetwarzają uszkodzenia dimeru cyklobutanopirymidyny (CPD) z bakterii Gram-ujemnych i Gram-dodatnich, halofilnych archeonów Halobacterium halobium .
- Fotoliazy CPD klasy 2 przetwarzają również zmiany CPD. Występują w roślinach takich jak rzeżucha pospolita Arabidopsis thaliana i ryż .
- Kryptochromy roślin i grzybów są podobne do CPD klasy 1. Są fotoreceptorami światła niebieskiego, które pośredniczą w ekspresji genów indukowanej światłem niebieskim i modulacji rytmów okołodobowych .
- Liazy CPD klasy 3 tworzą siostrzaną grupę kryptochromów roślinnych, które z kolei są grupą siostrzaną CPD klasy 1.
- Grupa Cry-DASH to liazy CPD wysoce specyficzne dla jednoniciowego DNA. Członkowie obejmują Vibrio cholerae , X1Cry z Xenopus laevis i AtCry3 z Arabidopsis thaliana . DASH został początkowo nazwany na cześć Drosophila , Arabidopsis , Synechocystis i Human , czterech taksonów, które początkowo uważano za niosące tę rodzinę liaz. Od tego czasu kategoryzacja uległa zmianie. Część ich nazwy „Cry” wynikała z początkowych założeń, że są to kryptochromy.
- Fotoliazy (6-4)DNA eukariotów tworzą grupę z kryptochromami zwierzęcymi kontrolującymi rytmy dobowe. Występują u różnych gatunków, w tym Drosophila i ludzi. Kryptochromy mają swoje własne szczegółowe grupowanie.
- Bakteryjne liazy 6-4 ( InterPro : IPR007357 ), znane również jako grupa FeS-BCP, tworzą własną grupę zewnętrzną w stosunku do wszystkich fotoliaz.
Gałąź CPD innej niż klasa 2 jest zwykle grupowana w klasie 1 w niektórych systemach, takich jak PRINTS (PR00147). Chociaż członkowie mniejszych grup są zgodni, filogeneza może się znacznie różnić między autorami ze względu na różnice w metodologii, co prowadzi do pewnego zamieszania z autorami, którzy próbują dopasować wszystko (oszczędzając FeS-BCP) do klasyfikacji dwóch klas. Kryptochromy tworzą polifiletyczną obejmującą fotoliazy, które utraciły swoją aktywność naprawy DNA i zamiast tego kontrolują rytmy okołodobowe.
Aplikacja
Dodanie fotoliazy z niebiesko-zielonej algi Anacystis nidulans do komórek HeLa częściowo zmniejszyło uszkodzenia DNA spowodowane ekspozycją na UVB.
Białka ludzkie zawierające tę domenę
Nomenklatura
Systematyczna nazwa tej klasy enzymów to liaza pirymidynowa dezoksyrybocyklobutadipirymidyny . Inne powszechnie używane nazwy to enzym fotoreaktywujący , fotoliaza DNA , enzym fotoreaktywujący DNA , fotoliaza cyklobutano-dipirymidynowa , fotoliaza DNA , fotoliaza dezoksyrybonukleinowa , fotoliaza dezoksyrybodipirymidyny , fotoliaza , PRE , fotoliaza PhrB , dezoksyrybonukleinowa fotoliaza cyklobutano-dipirymidynowa , fotoliaza phr A , fotoliaza dipirymidynowa (światłoczuła) i dezoksyrybojądrowa dimer liaza pirymidynowa (światłoczuła) . Enzym ten należy do rodziny liaz , w szczególności do „catch-all” klasy liaz węgiel-węgiel.
Dalsza lektura
- Eker AP, Fichtinger-Schepman AM (1975). „Badania enzymu fotoreaktywującego DNA ze Streptomyces griseus II. Oczyszczanie enzymu”. Biochim. Biofiza. Akta . 378 (1): 54–63. doi : 10.1016/0005-2787(75)90136-7 . PMID 804322 .
- Sancar GB, Smith FW, Reid R, Payne G, Levy M, Sancar A (1987). „Mechanizm działania fotoliazy DNA Escherichia coli. I. Tworzenie kompleksu enzym-substrat” . J. Biol. chemia . 262 (1): 478–85. doi : 10.1016/S0021-9258(19)75952-3 . PMID 3539939 .
- Setlow JK, Bollum FJ (1968). „Minimalna wielkość substratu dla enzymu fotoreaktywującego drożdże”. Biochim. Biofiza. Akta . 157 (2): 233-7. doi : 10.1016/0005-2787(68)90077-4 . PMID 5649902 .
Linki zewnętrzne
-
 Media związane z Photolyase w Wikimedia Commons
Media związane z Photolyase w Wikimedia Commons