DEPDC5
| Identyfikatory | |||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| DEPDC5 | |||||||||||||||||||||||||||||||||||||||||||||||
| , DEP.5, FFEVF, domena DEP zawierająca 5, FFEVF1, domena DEP zawierająca 5, podjednostka podzłożona GATOR1 | |||||||||||||||||||||||||||||||||||||||||||||||
| Identyfikatory zewnętrzne | |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
| Wikidane | |||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||
DEPDC5 (lub domena DEP zawierająca 5 ) jest ludzkim białkiem o słabo poznanej funkcji, ale w kilku badaniach wiązano go z rakiem . Jest kodowana przez gen o tej samej nazwie, zlokalizowany na chromosomie 22 .
Funkcjonować
Funkcja DEPDC5 nie jest jeszcze znana, ale bierze się udział w transdukcji sygnału wewnątrzkomórkowego w oparciu o homologię między domenami DEP DEPDC5 i Dishevelled-1 ( DVL1 ).
Mutacje w tym genie zostały powiązane z przypadkami padaczki ogniskowej (doi:10.1038/ng.2601).
Gen
U Homo sapiens gen DEPDC5 został zlokalizowany na długim ramieniu chromosomu 22, 22q12.2-q12.3, pomiędzy genami PRRL14 i YWHAH . Kliniczne znaczenie tego genu obejmuje intronowy SNP (rs1012068), który jest związany z 2-krotnym wzrostem ryzyka raka wątrobowokomórkowego .
Struktura
Domeny
DEP
Domena DEP wywodzi swoją nazwę od białek Disheveled , Egl-10 i Pleckstrin , z których każde zawiera wariant tej domeny. Obejmuje 82 reszty i ma 343 aminokwasy od C-końca . MODEL SZWAJCARSKI przewiduje dwa arkusze beta i trzy helisy alfa zawarte w domenie.
Chociaż jego dokładna funkcja nie jest znana, domena DEPDC5 DEP ma największe podobieństwo strukturalne do domeny DEP DVL1 podczas wykonywania CBLAST w NCBI . Dopasowanie daje wartość E 1,00e-08 i wskazuje 30% identyczności między domenami DEP dwóch białek. W DVL1 domena DEP bierze udział w lokalizacji białka w błonie komórkowej jako część szlaku sygnałowego Wnt .
DUF 3608
Domena DUF 3608 znajduje się 99 aminokwasów od N-końca i sama obejmuje 280 aminokwasów. PELE przewiduje co najmniej jeden arkusz beta i dwie helisy alfa w tej domenie. Zawiera również 26 wysoce konserwatywnych reszt i kilka modyfikacji potranslacyjnych. Oba wystąpienia zostały omówione w dalszej części tego artykułu.
Dowody na działanie DUF 3608 zostały odkryte w homologu drożdży Iml1p. Uważa się, że DUF 3608 Imlp1 pomaga w wiązaniu z dwoma partnerami białkowymi, Npr2 i Npr3. autofagii „głodu bez azotu” . Naukowcy, którzy to odkryli, proponują zmianę nazwy DUF 3608 na RANS (wymagane do autofagii indukowanej w warunkach głodu bez azotu).
Struktura drugorzędowa
W oparciu o jednomyślny konsensus narzędzia PELE do przewidywania struktury drugorzędowej, DEPDC5 zawiera co najmniej dziesięć helis alfa i dziewięć arkuszy beta. Lokalizacje tych drugorzędowych struktur pokazano na poniższym obrazku: czerwone światła to helisy alfa, a niebieskie światła to arkusze beta.
Homologia
ortologi
Grzyby są najbardziej spokrewnionymi organizmami zawierającymi białko ortologiczne do ludzkiego DEPDC5, w tym Saccharomyces cerevisiae i Albugo laibachii . U grzybów nazwa białka to Iml1p lub białko związane z błoną wakuolową Iml1. Odchylenia nazw u innych organizmów obejmują CG12090 ( Drosophila ) i AGAP007010 ( komar ). Ochrona jest wysoka między ludźmi a innymi kręgowców , od 74% identyczności u pielęgnic do 99% identyczności u szympansów .
Poniższa tabela podsumowuje analizę 20 białek ortologicznych do ludzkiego DEPDC5.
| Gatunek | Nazwa zwyczajowa | Numer dostępu do NCBI | Nazwa NCBI | Długość | Tożsamość sekwencji | Podobieństwo sekwencji | Lata od odejścia od człowieka (mya) |
| Panowie troglodyci | Szympans | XP_003317262 | DEPDC5 | 1572 aa | 99% | 99% | 6.4 |
| Nomascus leucogenys | Gibon | XP_003258163 | DEPDC5 | 1602 aa | 99% | 99% | 20.4 |
| Mus musculus | Mysz | NP_001164038 | DEPDC5 | 1591 aa | 94% | 96% | 92,4 |
| Bos Taurus | Krowa | XP_002694678 | DEPDC5 | 1593 aa | 94% | 96% | 94,4 |
| Sorex krzyżak | Jędza | ACE77702 | DEPDC5 | 1570 aa | 94% | 96% | 94,4 |
| Monodelphis domestica | Opos | XP_001378772 | DEPDC5 | 1522 aa | 89% | 93% | 163,9 |
| Gallus gallus | Kurczak | XP_415249 | DEPDC5 | 1592 aa | 88% | 93% | 301.7 |
| Meleagris gallopavo | Indyk | XP_003211073 | DEPDC5 | 1592 aa | 88% | 93% | 301.7 |
| Taeniopygia guttata | Zięba zebry | XP_002199825 | DEPDC5 | 1572 aa | 87% | 92% | 301.7 |
| Xenopus tropicalis | Żaba | XP_002931964 | podobny do DEPDC5 | 1574 aa | 79% | 86% | 371,2 |
| Danio Rerio | Zebry | XP_691450 | podobny do DEPDC5 | 1590 aa | 75% | 84% | 400.1 |
| Oreochromis niloticus | Pielęgnica | XP_003459226 | DEPDC5 | 1577 aa | 74% | 82% | 400.1 |
| Strongylocentrotus purpuratus | Jeżowiec | XP_794020 | podobny do DEPDC5 | 1608 aa | 43% | 57% | 742,9 |
| muszka owocowa | Drosophila | NP_647618 | GC12090 | 1471 aa | 41% | 57% | 782,7 |
| Pediculus humanus corporis | Wesz | XP_002429401 | DEPDC, domniemany | 1538 aa | 38% | 53% | 782,7 |
| Anopheles gambiae | Komar | XP_308760 | AGAP007010-PA | 1640 aa | 36% | 51% | 782,7 |
| Ascaris suum | Ascaris | ADY40551 | DEPDCp5 | 1359 aa | 31% | 51% | 937,5 |
| Ustilago maydis | Grzyb kukurydziany | XP_757759 | białko związane z wakuolami Iml1 | 1867 r | 23% | 52% | 1215,8 |
| Saccharomyces cerevisiae | Drożdże | NP_012672 | Iml1p | 1584 aa | 20% | 50% | 1215,8 |
| Albugo laibachii | Biała rdza | CCA27519 | domniemane białko związane z błoną wakuolową | 1591 aa | 20% | 46% | 1362 |
30 reszt zostało zachowanych od czasu rozejścia się zwierząt i grzybów, z czego 26 znajduje się w domenie DUF 3608. Poniższe dopasowanie wielu sekwencji ilustruje zachowanie domeny DUF ; przedstawiciele kladów bezkręgowców i grzybów są wyrównani do ludzkiego DUF 3608 z całkowicie konserwowanymi pozostałościami w kolorze zielonym.
Paralogi
Nie są znane ludzkie paralogi DEPDC5 , ale istnieje 64 ludzkich białek zawierających homologiczną domenę DEP. Nie ma również zidentyfikowanych paralogów dla białka drożdży Iml1, najbardziej odległego spokrewnionego ortologu ludzkiego DEPDC5.
Wyrażenie
Ekspresja DEPDC5 została scharakteryzowana jako wszechobecna w tkance ludzkiej za pomocą analizy RT-PCR i badań mikromacierzy DNA , jak pokazano na poniższym wykresie. 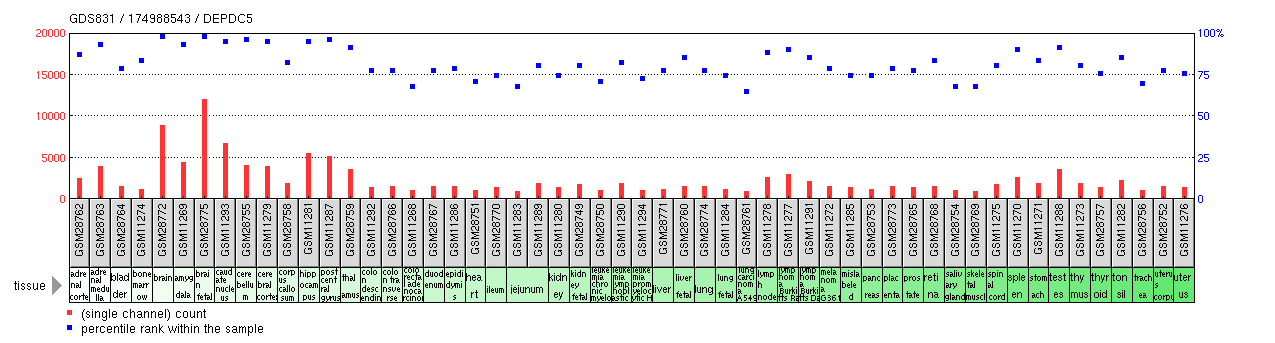
W jednym badaniu na pacjentach z rakiem wątrobowokomórkowym stwierdzono wyższą ekspresję DEPDC5 w tkance nowotworowej niż w tkance nienowotworowej. I odwrotnie, homozygotyczną delecję trzech genów, z których jeden to DEPDC5, stwierdzono w dwóch przypadkach glejaka wielopostaciowego . Inne anomalie ekspresji obejmują zerową ekspresję w linii komórkowej raka sutka MDA-MB-231 i niską ekspresję w linii komórkowej P116 ( ZAP70 ujemnej).
Modyfikacje potranslacyjne
Następujące modyfikacje potranslacyjne zostały przewidziane za pomocą narzędzi proteomicznych skompilowanych w ExPASy i PhosphoSite Plus dla ludzkiego białka DEPDC5.
| Modyfikacja potranslacyjna | Liczba/miejsce | Źródło |
| Fosforylacja | 133/(Ser: 87 Thr: 23 Tyr: 23) | NetPhos |
| 6/S579, S582, S1499, Y1515, Y1519, Y1543 | PhosphoSite Plus | |
| glikacja | 29/5, 8, 13, 14, 28, 34, 56, 59, 64, 93, 131, 147, 229, 247, 256, 319, 436, 528, 609, 710, 862, 878, 1008, 1185, 1233, 1387, 1408, 1499, 1567, 1597 | NetGlycate |
| Miejsce N-glikozylacji | 9/N201, N298, N311, N384, N684, N1157, N1377, N1444, N1529 | NetNGlyc |
| Zasiarczenie | 3/Y397, Y459, Y462 | Sulfinator |
| Sumoilacja | 2/K59, K147 | SUMOsp |
| Rozszczepienie propeptydu | 2/R1004-M1005, R1528-N1529 | Rekwizyt |
| O-glikozylacja | 0 | NetOGlyc |
| C-mannozylacja | 0 | NetCGlyc |
| mirystoilacja | 0 | mirystoilacja |
| Prenylacja | 0 | PrePS zarchiwizowane 2012-02-08 w Wayback Machine |
| Acetylacja | 0 | NetAcet |
Interakcja
DEPDC5 może prawdopodobnie oddziaływać z podjednostką proteasomu PSMA3 , o czym świadczy koimmunoprecypitacja i czynnik transkrypcyjny MYC . DEPDC5 jest w kompleksie „GATOR1” z NPRL2 i NPRL3 .







