Kuracyna A
 | |
| Identyfikatory | |
|---|---|
| |
| Numer CAS | |
| Identyfikator klienta PubChem | |
| ChemSpider | |
| UNII | |
| KEGG | |
| CHEBI | |
| CHEMBL | |
| Dane chemiczne i fizyczne | |
| Formuła | C 23 H 35 N O S |
| Masa cząsteczkowa | 373,60 g·mol -1 |
| Model 3D ( JSmol ) | |
| |
| |
Curacin A to hybryda syntazy poliketydowej (PKS) / nierybosomalnej syntazy peptydowej (NRPS), pochodzący z naturalnego produktu, wytwarzanego w izolacji z cyjanobakterii Lyngbya majuscula . Curacin A należy do rodziny produktów naturalnych, w tym jamajkamid, mupirocyna i pederyna , które mają niezwykły końcowy alken . Dodatkowo Curacin A zawiera znaczący tiazolinowy i unikalne ugrupowanie cyklopropylowe , które jest niezbędne dla aktywności biologicznej związku . Kuracyna A została scharakteryzowana jako silny antyproliferacyjny cytotoksyczny o znaczącej aktywności przeciwnowotworowej dla kilku linii raka , w tym raka nerki, okrężnicy i piersi. Wykazano, że kuracyna A oddziałuje z miejscami wiązania kolchicyny na tubulinie, co hamuje polimeryzację mikrotubul, niezbędny proces podziału i proliferacji komórek.
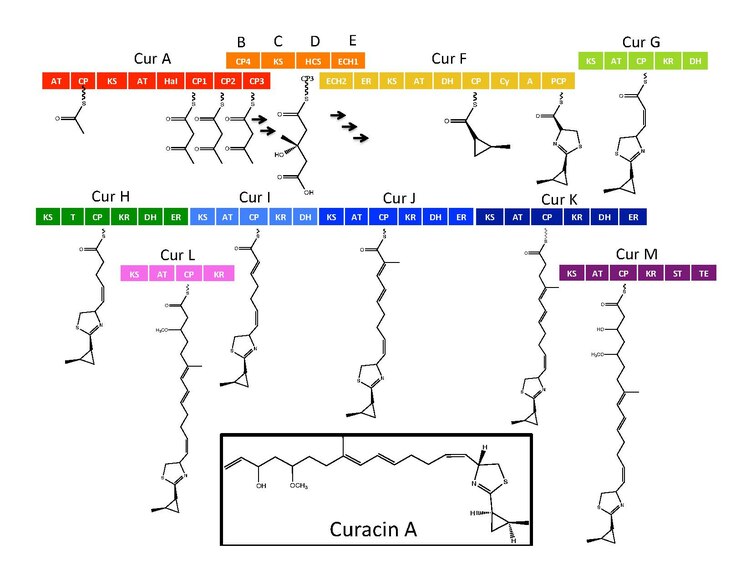
Biosynteza
Syntetyczne enzymy dla kuracyny A znajdują się w klastrze genów z 14 otwartymi ramkami odczytu (ORF) z nazewnictwem od CurA do CurN. Analiza szlaku wykazała obecność jednego modułu hybrydowego NRPS/PKS zlokalizowanego na CurF, jednej kasety syntazy HMG-CoA zlokalizowanej na CurD i siedmiu monomodułowych modułów PKS. CurA zawiera unikalną domenę ładującą N -acetylotransferazę (GNAT) związaną z GCN5 i związane z nią acylowe białko nośnikowe (ACP). Moduł ładujący wiąże grupę acetylową z ACP, która następnie kondensuje z jednym z trzech tandemowych ACP obecnych w sąsiednim module CurA. Kaseta syntazy hydroksymetyloglutarylo-CoA (szlak mewalonianu) katalizuje tworzenie kwasu hydroksymetyloglutarylowego przez dodanie jednostki malonylo-CoA do końcowego ketydu ugrupowania aceto-acetylo-ACP ACP1, ACP2 lub ACP3. kolejne enzymy, w tym unikalna halogenaza niezależna od hemu (HaI), katalizują tworzenie pierścienia cyklopropylowego. Specyficzny dla cysteiny moduł NRPS zlokalizowany na CurF następuje po utworzeniu pierścienia cyklopropylowego i ze względu na aktywność cyklizującej domeny kondensacyjnej tworzy pierścień tiazolowy przyłączony do ugrupowania cyklopropylowego z poprzednich reakcji na szlaku. Następnie następuje siedem samodzielnych modułów PKS, aby rozszerzyć rosnący łańcuch poliketydowy o metylacje zależne od S -adenozylometioniny (SAM) występujące w pozycjach 10 i 13. Ostatni moduł syntazy kuracyny wykorzystuje rzadką strategię odciążania obejmującą sulfotransferazę. Sulfotransferaza siarczanuje grupę hydroksylową węgla 15, co aktywuje cząsteczkę do dekarboksylacji i tworzenia końcowego alkenu.
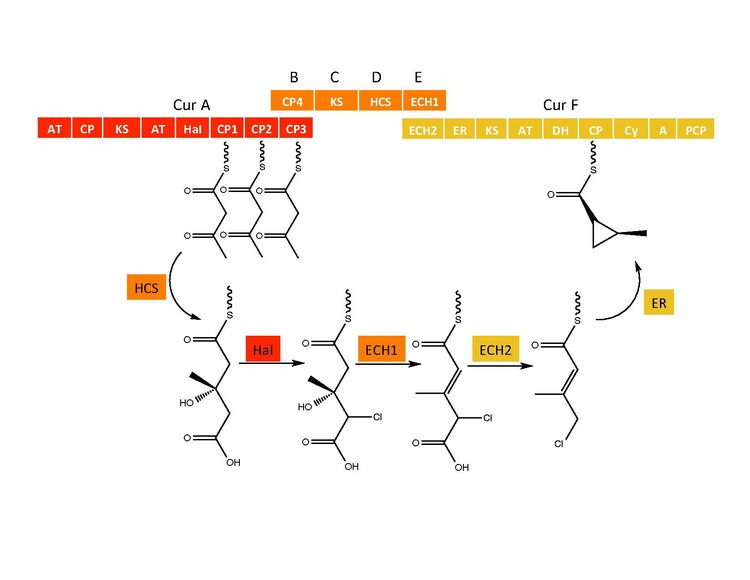
Tworzenie pierścienia cyklopropylowego
CurB (ACP), CurC (ketosyntaza) i CurD (reduktaza HMG-CoA) są odpowiedzialne za tworzenie (S) HMG-ACP3. HaI, z genu CurA, jest unikalną halogenazą niehemową, która przechodzi przez rzekomy związek pośredni Fe(IV)=O w celu dodania atomu chloru do nieaktywowanego atomu węgla. Po chlorowaniu ECH1 działając jako odwadniacz HMG-ACP3 do 3-metylogultakonylo-ACP3, a ECH2 przeprowadza wymaganą dekarboksylację. Wreszcie, niezwykła reakcja cyklizacji katalizowana przez ER, rzekomo przechodząca przez mechanizm podobny do podstawienia, tworzy pierścień cyklopropanu. Dodany atom chloru pomaga w etapie dekarboksylacji i prawdopodobnie służy jako grupa opuszczająca podczas tworzenia pierścienia cyklopropanu.