Międzynarodowy identyfikator chemiczny
| Deweloperzy | Zaufanie InChI |
|---|---|
| Pierwsze wydanie | 15 kwietnia 2005 |
| Wersja stabilna | 1.06 / 15 grudnia 2020
|
| System operacyjny | Microsoft Windows i Unix |
| Platforma | IA-32 i x86-64 |
| Dostępne w | język angielski |
| Licencja | Licencja zaufania IUPAC / InChI |
| Strona internetowa |
Międzynarodowy identyfikator chemiczny ( InChI / sposobu ɪ n tʃ iː / IN -chee lub / ) ɪ ŋ k iː / ING -kee to tekstowy identyfikator substancji chemicznych , zaprojektowany w celu zapewnienia standardowego kodowania informacji molekularnych i ułatwienia szukaj takich informacji w bazach danych iw sieci. Początkowo opracowany przez Międzynarodową Unię Chemii Czystej i Stosowanej (IUPAC) i National Institute of Standards and Technology (NIST) od 2000 do 2005, format i algorytmy nie są zastrzeżone. Od maja 2009 r. jest rozwijany przez InChI Trust, organizację charytatywną non-profit z Wielkiej Brytanii , która działa na rzecz wdrażania i promowania korzystania z InChI.
Identyfikatory opisują substancje chemiczne w kategoriach warstw informacji — atomy i ich połączenia wiązań, informacje o tautomerach , informacje o izotopach , stereochemia i informacje o ładunku elektronicznym. Nie wszystkie warstwy muszą być zapewnione; na przykład warstwę tautomeru można pominąć, jeśli ten rodzaj informacji nie jest odpowiedni dla konkretnego zastosowania. Algorytm InChI przekształca wejściowe informacje strukturalne w unikalny identyfikator InChI w trzyetapowym procesie: normalizacji (w celu usunięcia zbędnych informacji), kanonizacji (w celu wygenerowania unikalnej etykiety numerycznej dla każdego atomu) i serializacji (w celu nadania ciągu znaków) .
InChI różnią się od powszechnie używanych numerów rejestracyjnych CAS pod trzema względami: po pierwsze, można ich swobodnie używać i nie są one zastrzeżone; po drugie, mogą być obliczone na podstawie informacji strukturalnych i nie muszą być przypisywane przez jakąś organizację; i po trzecie, większość informacji w InChI jest czytelna dla człowieka (z praktyką). InChIs można zatem postrzegać jako pokrewne ogólnej i niezwykle sformalizowanej wersji nazw IUPAC . Mogą przekazać więcej informacji niż prostsze UŚMIECHY notacji i różnią się tym, że każda struktura ma unikalny ciąg InChI, co jest ważne w aplikacjach bazodanowych. Informacje o trójwymiarowych współrzędnych atomów nie są reprezentowane w InChI; można użyć formatu takiego jak PDB .
InChIKey, czasami określany jako zaszyfrowany InChI, to skondensowana cyfrowa reprezentacja InChI o stałej długości (27 znaków), która nie jest zrozumiała dla człowieka. Specyfikacja InChIKey została wydana we wrześniu 2007 roku w celu ułatwienia wyszukiwania w Internecie związków chemicznych, ponieważ były one problematyczne w przypadku pełnej długości InChI. W przeciwieństwie do InChI, InChIKey nie jest unikalny: chociaż można obliczyć, że kolizje są bardzo rzadkie, zdarzają się.
W styczniu 2009 została wydana wersja 1.02 oprogramowania InChI. Zapewniło to środki do generowania tak zwanego standardowego InChI, który nie pozwala na wybór przez użytkownika opcji dotyczących stereochemii i warstw tautomerycznych łańcucha InChI. Standardowy InChIKey jest wtedy zaszyfrowaną wersją standardowego ciągu InChI. Standard InChI uprości porównywanie ciągów i kluczy InChI generowanych przez różne grupy, a następnie udostępnianych za pośrednictwem różnych źródeł, takich jak bazy danych i zasoby internetowe.
Ciągły rozwój standardu jest wspierany od 2010 roku przez organizację non-profit InChI Trust , której IUPAC jest członkiem. Obecna wersja oprogramowania to 1.06 i została wydana w grudniu 2020 r. Przed 1.04 oprogramowanie było dostępne bezpłatnie na LGPL typu open source , ale teraz korzysta z niestandardowej licencji o nazwie IUPAC-InChI Trust License.
Pokolenie
Aby uniknąć generowania różnych InChI dla struktur tautomerycznych, przed wygenerowaniem InChI, wejściowa struktura chemiczna jest normalizowana w celu zredukowania jej do tak zwanej podstawowej struktury macierzystej. Może to obejmować zmianę rzędów wiązań, zmianę układu ładunków formalnych i ewentualnie dodawanie i usuwanie protonów. Różne struktury danych wejściowych mogą dawać ten sam wynik; na przykład kwas octowy i octan dawałyby tę samą macierzystą strukturę rdzenia, strukturę kwasu octowego. Rdzeń struktury macierzystej może być odłączony, składający się z więcej niż jednego składnika, w którym to przypadku podwarstwy w InChI zwykle składają się z podwarstw dla każdego składnika, oddzielonych średnikami (okresy dla podwarstwy wzoru chemicznego). Jednym ze sposobów, w jaki może się to zdarzyć, jest to, że wszystkie atomy metali są odłączone podczas normalizacji; więc na przykład InChI dla tetraetyloołowiu będzie miał pięć składników, jeden dla ołowiu i cztery dla grup etylowych.
Pierwsza, główna warstwa InChI odnosi się do tej podstawowej struktury macierzystej, podając jej wzór chemiczny, łączność niewodorową bez kolejności wiązań (podwarstwa /c) i łączność wodorową ( podwarstwa /h ). Część /q warstwy ładunku daje swój ładunek, a część / p warstwy ładunku mówi, ile protonów (jonów wodoru) należy dodać lub usunąć, aby zregenerować pierwotną strukturę. Jeśli występuje, warstwa stereochemiczna z podwarstwami /b , /t , /m i /s , dostarcza informacji stereochemicznych, a warstwa izotopowa /i (która może zawierać podwarstwy /h , /b , /t , /m i /s ) dostarcza informacji izotopowych. Są to jedyne warstwy, które mogą wystąpić w standardowym InChI.
może dołączyć stałą warstwę wodoru /f , która może zawierać różne dodatkowe podwarstwy; nie można tego jednak zrobić w standardowym InChI, więc różne tautomery będą miały ten sam standardowy InChI (na przykład alanina da ten sam standardowy InChI, niezależnie od tego, czy wejście jest w formie neutralnej, czy obojnaczej). Wreszcie, niestandardowa ponownie połączona / r warstwa może być dodano, co skutecznie daje nowy InChI generowany bez zrywania wiązań z atomami metali. Może to zawierać różne warstwy podrzędne, w tym /f .
Formatowanie i warstwy
| Rodzaj mediów internetowych |
chemiczny/x-inchi
|
|---|---|
| Typ formatu | format pliku chemicznego |
Każdy InChI zaczyna się ciągiem znaków „ InChI= ”, po którym następuje numer wersji, obecnie 1 . Jeśli InChI jest standardem, po nim następuje litera S dla standardowego InChIs , który jest w pełni znormalizowanym smakiem InChI, zachowującym ten sam poziom dbałości o szczegóły struktury i te same konwencje dotyczące percepcji rysunkowej. Pozostałe informacje mają strukturę sekwencji warstw i podwarstw, przy czym każda warstwa zapewnia jeden określony typ informacji. Warstwy i podwarstwy są oddzielone ogranicznikiem " / ” i rozpoczynają się charakterystycznym przedrostkiem (z wyjątkiem podwarstwy warstwy głównej o wzorze chemicznym). Sześć warstw z ważnymi podwarstwami to:
- Warstwa główna
- Wzór chemiczny (bez przedrostka). Jest to jedyna podwarstwa, która musi wystąpić w każdym InChI. Liczby używane w całym InChI są podane w kolejności elementów we wzorze z wyłączeniem atomów wodoru. Na przykład „/C10H16N5O13P3” oznacza, że atomy o numerach 1–10 to atomy węgla, 11–15 to atomy azotu, 16–28 to tlen, a 29–31 to fosfor.
- Połączenia atomowe (przedrostek: „
c”). Atomy we wzorze chemicznym (z wyjątkiem wodorów) są ponumerowane w kolejności; ta podwarstwa opisuje, które atomy są połączone wiązaniami, z którymi innymi. -
wodoru (przedrostek: „
h”). Opisuje, ile atomów wodoru jest połączonych z każdym z pozostałych atomów.
-
Warstwa ładunku
- podwarstwa ładunku (przedrostek: „
q”) - podwarstwa protonów (przedrostek: „
p” dla „protonów”)
- podwarstwa ładunku (przedrostek: „
- Warstwa stereochemiczna
-
izotopowa (przedrostki: „
i”, „h”, a także „b”, „t”, „m”, „s” dla stereochemii izotopowej) - Warstwa Fixed-H (przedrostek: „
f”); zawiera niektóre lub wszystkie z powyższych rodzajów warstw z wyjątkiem połączeń atomowych; może kończyć się podwarstwą „o”; nigdy nie zawarte w standardowym InChI - Ponownie połączona warstwa (przedrostek: „
r”); zawiera całe InChI struktury z ponownie połączonymi atomami metalu; nigdy nie zawarte w standardowym InChI
Format ogranicznika-prefiksu ma tę zaletę, że użytkownik może łatwo użyć wyszukiwania wieloznacznego , aby znaleźć identyfikatory pasujące tylko w niektórych warstwach.
| Formuła strukturalna | standardowe InChI |
|---|---|
InChI=1S/C2H6O/c1-2-3/h3H,2H2,1H3
|
|

L - kwas askorbinowy z InChI
|
InChI=1S/C6H8O6/c7-1-2(8)5-3(9)4(10)6(11)12-5/h2,5,7-10H,1H2/t2-,5+/m0/ s1
|
InChIKey
Skondensowany, 27-znakowy InChIKey to zaszyfrowana wersja pełnego InChI (przy użyciu algorytmu SHA-256 ), zaprojektowana w celu umożliwienia łatwego wyszukiwania w Internecie związków chemicznych. Standardowy InChIKey jest mieszanym odpowiednikiem standardowego InChI . Większość struktur chemicznych w Internecie do 2007 roku była reprezentowana jako pliki GIF , których nie można przeszukiwać pod kątem zawartości substancji chemicznych. Pełny InChI okazał się zbyt długi, aby można go było łatwo przeszukiwać, dlatego opracowano InChIKey. Istnieje bardzo mała, ale różna od zera szansa, że dwie różne cząsteczki będą miały ten sam InChIKey, ale prawdopodobieństwo zduplikowania tylko pierwszych 14 znaków zostało oszacowane jako tylko jedna duplikacja w 75 bazach danych, z których każda zawiera miliard unikalnych struktur. Ponieważ wszystkie bazy danych mają obecnie mniej niż 50 milionów struktur, taka duplikacja wydaje się obecnie mało prawdopodobna. Niedawne badania dokładniej badają częstość kolizji, stwierdzając, że eksperymentalna częstość kolizji jest zgodna z teoretycznymi oczekiwaniami.
InChIKey obecnie składa się z trzech części oddzielonych myślnikami, odpowiednio 14, 10 i jednego znaku, np. XXXXXXXXXXXXXX-YYYYYYYYFV-P . Pierwsze 14 znaków wynika z skrótu SHA-256 informacji o łączności (warstwa główna i /q warstwy ładunku) InChI. Druga część składa się z 8 znaków wynikających z haszowania pozostałych warstw InChI, pojedynczego znaku wskazującego rodzaj InChIKey ( S dla standardowego i N dla niestandardowego) oraz znaku wskazującego wersję używanej InChI (obecnie A dla wersji 1.) Wreszcie pojedynczy znak na końcu wskazuje protonację macierzystej struktury rdzenia, odpowiadającą podwarstwie / p warstwy ładunku ( N oznacza brak protonowania, O , P , ... jeśli należy dodać protony i M , L , ... jeśli powinny zostać usunięte.)
Przykład
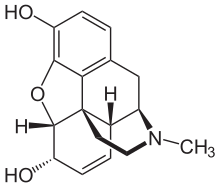
Morfina ma strukturę pokazaną po prawej stronie. Standardowy InChI dla morfiny to InChI=1S/C17H19NO3/c1-18-7-6-17-10-3-5-13(20)16(17)21-15-12(19)4-2-9( 14(15)17)8-11(10)18/h2-5,10-11,13,16,19-20H,6-8H2,1H3/t10-,11+,13-,16-,17- /m0/s1 , a standardowy InChIKey dla morfiny to BQJCRHHNABKAKU-KBQPJGBKSA-N .
resolwery InChI
Ponieważ InChI nie można zrekonstruować z InChIKey, InChIKey zawsze musi być połączony z oryginalnym InChI, aby powrócić do pierwotnej struktury. InChI Resolvers działają jako usługa wyszukiwania w celu utworzenia tych linków, a prototypowe usługi są dostępne w National Cancer Institute , usłudze UniChem w Europejskim Instytucie Bioinformatyki oraz PubChem . ChemSpider miał resolwer do lipca 2015 roku, kiedy został wycofany z eksploatacji.
Nazwa
Format pierwotnie nosił nazwę IChI (IUPAC Chemical Identifier), a następnie w lipcu 2004 r. przemianowano go na INChI (IUPAC-NIST Chemical Identifier), aw listopadzie 2004 r. ponownie zmieniono nazwę na InChI (IUPAC International Chemical Identifier), znak towarowy IUPAC.
Ciągły rozwój
Kierownictwo naukowe standardu InChI jest prowadzone przez IUPAC Division VIII Subcommitte, a finansowanie podgrup badających i definiujących rozszerzenie standardu jest prowadzone zarówno przez IUPAC , jak i InChI Trust . InChI Trust finansuje rozwój, testowanie i dokumentację InChI. Obecne rozszerzenia są definiowane w celu obsługi polimerów i mieszanin , struktur Markusha , reakcji i metaloorganicznych , a po zaakceptowaniu przez Podkomisję Wydziału VIII zostaną dodane do algorytmu.
Oprogramowanie
InChI Trust opracowało oprogramowanie do generowania InChI, InChIKey i innych identyfikatorów. Poniżej przedstawiono historię wydań tego oprogramowania.
| Oprogramowanie i wersja | Data | Licencja | Uwagi |
|---|---|---|---|
| InChI w. 1 | kwiecień 2005 | ||
| InChI w. 1.01 | sierpień 2006 | ||
| InChI v. 1.02beta | wrzesień 2007 | LGPL 2.1 | Dodaje funkcjonalność InChIKey. |
| InChI w. 1.02 | styczeń 2009 r | LGPL 2.1 |
Zmieniono format InChIKey. Wprowadza standardowe InChI. |
| InChI w. 1.03 | czerwiec 2010 | LGPL 2.1 | |
| Dokumentacja kodu źródłowego InChI v. 1.03 | marzec 2011 r | ||
| InChI w. 1.04 | wrzesień 2011 r | Licencja IUPAC/InChI Trust InChI 1.0 |
Nowa licencja. Dodano obsługę elementów 105-112. Usunięto obsługę CML. |
| InChI w. 1.05 | styczeń 2017 r | Licencja IUPAC/InChI Trust InChI 1.0 |
Dodano obsługę elementów 113-118. Eksperymentalne podłoże polimerowe. Eksperymentalne wsparcie dla dużych cząsteczek. |
| RInChI w. 1.00 | marzec 2017 r | Licencja IUPAC/InChI Trust InChI 1.0 i w stylu BSD | Oblicza reakcję InChIs. |
| InChI w. 1.06 | grudzień 2020 r | Licencja IUPAC/InChI Trust InChI 1.0 | Zmieniony wspornik polimerowy. |
Przyjęcie
InChI został przyjęty przez wiele większych i mniejszych baz danych, w tym ChemSpider , ChEMBL , Golm Metabolome Database , OpenPHACTS i PubChem . Jednak przyjęcie nie jest proste, a wiele baz danych wykazuje rozbieżności między strukturami chemicznymi a zawartym w nich InChI, co stanowi problem przy łączeniu baz danych.
Zobacz też
- Molekularny język zapytań
- Uproszczony system wprowadzania danych molekularnych (SMILES)
- Edytor cząsteczek
- Notacja liniowa SYBYL
- Bioclipse generuje InChI i InChIKeys dla narysowanych struktur lub otwartych plików
- Chemistry Development Kit wykorzystuje JNI-InChI do generowania InChI, może przekształcać InChI w struktury i generować tautomery w oparciu o algorytmy InChI
Uwagi i odniesienia
Linki zewnętrzne
- Witryna IUPAC InChI
- Opis algorytmu kanonizacji
- Googlowanie dla InCh to prezentacja dla W3C.
- InChI wydanie 1.02 ostateczna wersja InChI 1.02 i wyjaśnienie standardu InChI, styczeń 2009
- NCI/CADD Chemical Identifier Resolver Generuje i rozwiązuje InChI/InChIKeys i wiele innych identyfikatorów chemicznych
- Internetowy edytor cząsteczek PubChem obsługujący SMILES / SMARTS i InChI
- ChemSpider Compound API ChemSpider REST API , który umożliwia generowanie InChI i konwersję InChI do struktury (również SMILES i generowanie innych właściwości)
- MarvinSketch z ChemAxon , implementacja do rysowania struktur (lub otwierania innych formatów plików) i wyjścia do formatu pliku InChI
- BKchem implementuje własny parser InChI i wykorzystuje implementację IUPAC do generowania łańcuchów InChI
- CompoundSearch implementuje wyszukiwanie bibliotek widmowych InChI i InChI Key
- SpectraBase implementuje wyszukiwanie bibliotek spektralnych za pomocą InChI i InChI Key
- JSME Archived 2015-01-06 at the Wayback Machine to darmowy edytor molekularny oparty na JavaScript, który generuje InChI i InChI Key w przeglądarce internetowej, co pozwala na łatwe wyszukiwanie w Internecie związków chemicznych