Wirus Bacillus phi29

|
|
| Wirus Bacillus Φ29 | |
|---|---|
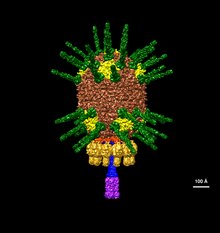
| Ilustracja przedstawiająca głowę Φ29 na podstawie danych z mikroskopii elektronowej EMDB-2162 | |
| Klasyfikacja wirusów | |
| (nierankingowe): | Wirus |
| królestwo : | Duplodnaviria |
| Królestwo: | Heunggongvirae |
| Gromada: | Urowiricota |
| Klasa: | Caudoviricetes |
| Zamówienie: | Caudovirales |
| Rodzina: | Salasmaviridae |
| Rodzaj: | Salaswirus |
| Gatunek: |
Wirus Bacillus Φ29
|
Wirus Bacillus Φ29 (bakteriofag Φ29) to bakteriofag z dwuniciowym DNA (dsDNA) z wydłużoną dwudziestościenną głową i krótkim ogonem, który należy do rodzaju Salasvirus , rzędu Caudovirales i rodziny Salasmaviridae . Są w tej samej kolejności co fagi PZA, Φ15, BS32, B103, M2Y (M2), Nf i GA-1. Fag Φ29, odkryty po raz pierwszy w 1965 roku, jest najmniejszym Bacillus wyizolowanym do tej pory i jednym z najmniejszych znanych fagów dsDNA.
Φ29 ma unikalną strukturę motoryczną pakującą DNA, która wykorzystuje RNA pakujący prohead (pRNA) do kierowania translokacją genomu faga podczas replikacji . Ten nowatorski system strukturalny zainspirował trwające badania w dziedzinie nanotechnologii , dostarczania leków i terapii .
W naturze fag Φ29 infekuje Bacillus subtilis , gatunek bakterii Gram-dodatnich , tworzących przetrwalniki, które znajdują się w glebie , a także w przewodach pokarmowych różnych organizmów morskich i lądowych , w tym ludzi .
Historia
W 1965 roku amerykański mikrobiolog dr Bernard Reilly odkrył faga Φ29 w laboratorium dr Johna Spizizena na Uniwersytecie w Minnesocie . Ze względu na niewielkie rozmiary i złożoną morfologię stał się idealnym modelem do badania wielu procesów w biologii molekularnej , takich jak morfogeneza , pakowanie wirusowego DNA , replikacja wirusa i transkrypcja .
Struktura
Struktura Φ29 składa się z siedmiu głównych białek : białka końcowego (p3), białka główki lub kapsydu (p8), białka główki lub włókna kapsydu (p8.5), dystalnego guzka ogona (p9), portalu lub białko łączące (p10), białka rurki ogonowej lub dolnego kołnierza (p11) oraz włókna ogona lub białka przydatków (p12 *).
Główną różnicą między strukturą Φ29 a strukturą innych fagów jest wykorzystanie pRNA w silniku pakującym DNA.
Silnik pakujący DNA
Silnik pakujący DNA Φ29 pakuje genom faga do prokapsydu podczas replikacji wirusa. Silnik pakujący Φ29 składa się strukturalnie z prokapsydu i białek łączących, które oddziałują z pRNA, enzymem pakującym ( gp16) i substratem pakującym (genomowy DNA-gp3). Ponieważ proces pakowania genomu jest energochłonny , musi być wspomagany przez silnik napędzany ATP , który przekształca energię chemiczną w energię mechaniczną poprzez hydrolizę ATP . Silnik pakujący Φ29 jest w stanie wygenerować około 57 pikoniutonów (pN) siły , co czyni go jednym z najpotężniejszych dotychczas badanych biomotorów.
pRNA
pRNA Φ29 jest wysoce wszechstronną cząsteczką , która może polimeryzować do dimerów , trimerów , tetramerów , pentamerów i heksamerów . Wczesne badania, takie jak Anderson (1990) i Trottier (1998), postawiły hipotezę, że pRNA tworzy heksamery międzycząsteczkowe, ale badania te miały wyłącznie podstawę genetyczną , a nie podejście oparte na mikroskopii . W roku 2000 badanie przeprowadzone przez Simpsona i in. zastosowali mikroskopię krioelektronową , aby ustalić, że in vivo tylko pentamer lub mniejszy polimer może przestrzennie zmieścić się w wirusie. Ostatecznie zastosowano pojedyncze izomorficzne zastąpienie krystalografią z rozpraszaniem anomalnym (SIRAS) , aby określić, że struktura in vivo jest pierścieniem tetramerowym. To odkrycie było zgodne z wiedzą na temat geometrii strukturalnej i niezbędnej elastyczności trójdrożnego złącza silnika pakującego. Kiedy pRNA jest w tej formie pierścienia tetramerowego, działa jako część silnika pakującego DNA, który transportuje cząsteczki DNA do miejsca docelowego w kapsułce prohead. W szczególności domeny funkcjonalne pRNA wiążą się z enzymem pakującym gp16 i cząsteczką łącznika strukturalnego, aby pomóc w translokacji DNA przez kanał prohead. Po zakończeniu pakowania DNA pRNA dysocjuje i ulega degradacji.
Genom i replikacja
Fag Φ29 ma liniowy genom dsDNA składający się z 19 285 zasad . Oba końce 5' genomu są zakończone kowalencyjnie związanym końcowym białkiem (p3), które tworzy kompleks z polimerazą DNA podczas replikacji.
Φ29 jest jednym z wielu fagów z polimerazą DNA , która ma inną budowę i funkcję w porównaniu ze standardowymi polimerazami DNA w innych organizmach. Φ29 tworzy kompleks replikacyjny obejmujący końcowe białko p3, nukleotyd dAMP i własną polimerazę DNA w celu syntezy DNA w kierunku od 5' do 3' . Ten proces replikacji wykorzystuje również mechanizm cofania się w kierunku końca 3' genomu, który wykorzystuje powtarzający się motyw TTT do przesunięcia kompleksu replikacyjnego do tyłu bez zmiany sekwencji matrycy. na dokładniejsze zainicjowanie replikacji DNA dzięki sprawdzeniu przez kompleks polimerazy określonej sekwencji przed rozpoczęciem procesu wydłużania.
Aplikacje
Montaż nanocząstek
Wszechstronność struktury i funkcji RNA umożliwia łączenie nanocząstek do nanomedycznych środków terapeutycznych . pRNA w bakteriofagu Φ29 może wykorzystywać swoje trójdrożne połączenie do samodzielnego składania się w nanocząsteczki.
Jednym z głównych wyzwań związanych z wykorzystaniem nanocząstek pochodzących z pRNA jest produkcja na dużą skalę , ponieważ większość gałęzi przemysłu nie jest obecnie przystosowana do obsługi przemysłowej syntezy pRNA. Dzieje się tak przede wszystkim dlatego, że nanotechnologia RNA jest wciąż rozwijającą się dziedziną, w której brakuje zastosowań przemysłowych i optymalizacji produkcji małych RNA.
Dostawa narkotyków
System pakowania DNA Φ29, wykorzystujący pRNA, zawiera silnik do dostarczania cząsteczek terapeutycznych, takich jak rybozymy i aptamery . Niewielki rozmiar nanocząstek pochodzących z pRNA pomaga również dostarczać leki w ciasnych przestrzeniach, takich jak naczynia krwionośne .
Główną trudnością w stosowaniu dostarczania leków na bazie aptamerów jest pozyskiwanie unikalnych aptamerów i innych multimerów do określonych terapii chorób , które potencjalnie degradują multimery terapeutyczne i nanocząsteczki in vivo. Nanocząstki muszą być stabilizowane jako mechanizmy dostarczania, aby przystosować się do mikrośrodowisk, które mogą skutkować utratą ładunku terapeutycznego.
Leczenie potrójnie ujemnego raka piersi
Potrójnie ujemny rak piersi (TNBC) jest agresywną postacią raka piersi , która stanowi od dziesięciu do piętnastu procent wszystkich przypadków raka piersi. Chemioterapia jest obecnie jedynym realnym sposobem leczenia TNBC, ponieważ utrata docelowych receptorów nieodłącznie związana z chorobą powoduje, że komórki nowotworowe uodparniają się na środki farmaceutyczne .
Trójdrożne połączenie w silniku pakującym DNA Φ29 może pomóc uwrażliwić komórki TNBC na chemioterapię przy użyciu mechanizmu dostarczania leku siRNA w celu zahamowania wzrostu i objętości TNBC. Leczenie to można również łączyć z lekami przeciwnowotworowymi, takimi jak doksorubicyna , w celu wzmocnienia efektów terapeutycznych.