Dokowanie (molekularne)
| Glosariusz dokowania |
|---|
|
| edytować |
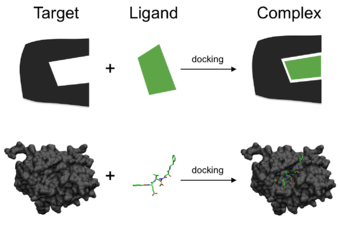
W dziedzinie modelowania molekularnego dokowanie to metoda przewidywania preferowanej orientacji jednej cząsteczki względem drugiej, gdy ligand i cel są ze sobą związane , tworząc stabilny kompleks . Znajomość preferowanej orientacji z kolei może być wykorzystana do przewidywania siły asocjacji lub powinowactwa wiązania między dwiema cząsteczkami przy użyciu, na przykład, funkcji oceny .
Powiązania między biologicznie istotnymi cząsteczkami, takimi jak białka , peptydy , kwasy nukleinowe , węglowodany i lipidy , odgrywają kluczową rolę w przekazywaniu sygnału . Ponadto względna orientacja dwóch oddziałujących partnerów może wpływać na rodzaj wytwarzanego sygnału (np. agonizm vs antagonizm ). Dlatego dokowanie jest przydatne do przewidywania zarówno siły, jak i rodzaju generowanego sygnału.
Dokowanie molekularne jest jedną z najczęściej stosowanych metod projektowania leków w oparciu o strukturę , ze względu na jego zdolność do przewidywania konformacji wiązania małocząsteczkowych ligandów z odpowiednim docelowym miejscem wiązania . Charakterystyka zachowania wiązania odgrywa ważną rolę w racjonalnym projektowaniu leków , a także w wyjaśnianiu podstawowych procesów biochemicznych.
Definicja problemu
Można myśleć o dokowaniu molekularnym jako o problemie „zamka i klucza” , w którym chce się znaleźć właściwą względną orientację „klucza” , który otworzy „zamek” (gdzie na powierzchni zamka znajduje się otwór klucza, w którą stronę należy przekręcić klucz po włożeniu itp.). W tym przypadku białko można traktować jako „zamek”, a ligand jako „klucz”. Dokowanie molekularne można zdefiniować jako problem optymalizacyjny, który opisuje „najlepiej dopasowaną” orientację ligandu, który wiąże się z określonym białkiem będącym przedmiotem zainteresowania. Jednakże, ponieważ zarówno ligand, jak i białko są elastyczne, „ręka w rękawiczce” jest bardziej odpowiednia niż „zamek i klucz” . W trakcie procesu dokowania, ligand i białko dostosowują swoją konformację, aby uzyskać ogólne „najlepsze dopasowanie”, a ten rodzaj dostosowania konformacyjnego skutkującego całkowitym wiązaniem jest określany jako „dopasowanie indukowane ” .
Badania nad dokowaniem molekularnym koncentrują się na komputerowej symulacji procesu rozpoznawania molekularnego . Ma na celu osiągnięcie zoptymalizowanej konformacji zarówno dla białka, jak i ligandu oraz względnej orientacji między białkiem i ligandem, tak aby energię swobodną całego systemu.
Podejścia do dokowania
W społeczności zajmującej się dokowaniem molekularnym szczególnie popularne są dwa podejścia. Jedno podejście wykorzystuje technikę dopasowywania, która opisuje białko i ligand jako powierzchnie komplementarne. Drugie podejście symuluje rzeczywisty proces dokowania, w którym obliczane są energie interakcji par ligand-białko. Oba podejścia mają istotne zalety, jak również pewne ograniczenia. Zostały one opisane poniżej.
Komplementarność kształtu
Metody dopasowywania geometrycznego/komplementarności kształtu opisują białko i ligand jako zestaw cech, które sprawiają, że można je dokować. Cechy te mogą obejmować powierzchni molekularnej /komplementarnych powierzchni. W tym przypadku powierzchnia cząsteczkowa receptora jest opisana w odniesieniu do jego powierzchni dostępnej dla rozpuszczalnika , a powierzchnia cząsteczkowa ligandu jest opisana w odniesieniu do opisu jego powierzchni pasującej. Komplementarność między dwiema powierzchniami sprowadza się do opisu dopasowania kształtu, który może pomóc w znalezieniu komplementarnej pozycji dokowania cząsteczek celu i ligandu. Innym podejściem jest opisanie właściwości hydrofobowych białka za pomocą skrętów w atomach łańcucha głównego. Jeszcze innym podejściem jest użycie techniki deskryptora kształtu Fouriera. Podczas gdy podejścia oparte na komplementarności kształtu są zazwyczaj szybkie i solidne, zwykle nie są w stanie dokładnie modelować ruchów lub dynamicznych zmian w konformacjach ligand/białko, chociaż ostatnie osiągnięcia pozwalają tym metodom badać elastyczność ligandu. Metody komplementarności kształtów mogą szybko przeskanować kilka tysięcy ligandów w ciągu kilku sekund i faktycznie dowiedzieć się, czy mogą one wiązać się w miejscu aktywnym białka i są zwykle skalowalne, aby nawet interakcje białko-białko. Są również znacznie bardziej podatne na farmakoforach , ponieważ wykorzystują geometryczne opisy ligandów, aby znaleźć optymalne wiązanie.
Symulacja
Symulowanie procesu dokowania jest znacznie bardziej skomplikowane. W tym podejściu białko i ligand są oddzielone pewną odległością fizyczną, a ligand znajduje swoją pozycję w miejscu aktywnym białka po określonej liczbie „ruchów” w swojej przestrzeni konformacyjnej. Ruchy obejmują transformacje ciała sztywnego, takie jak translacje i obroty, a także wewnętrzne zmiany w strukturze ligandu, w tym obroty pod kątem skrętu. Każdy z tych ruchów w przestrzeni konformacyjnej ligandu indukuje całkowity koszt energetyczny systemu. Stąd całkowita energia systemu jest obliczana po każdym ruchu.
Oczywistą zaletą symulacji dokowania jest to, że elastyczność ligandów jest łatwa do uwzględnienia, podczas gdy techniki komplementarności kształtu muszą wykorzystywać pomysłowe metody w celu uwzględnienia elastyczności ligandów. Ponadto dokładniej modeluje rzeczywistość, podczas gdy techniki uzupełniające kształty są bardziej abstrakcją.
Oczywiście symulacja jest kosztowna obliczeniowo i wymaga zbadania dużego krajobrazu energetycznego. Techniki oparte na siatce, metody optymalizacji i zwiększona prędkość komputera sprawiły, że symulacja dokowania stała się bardziej realistyczna.
Mechanika dokowania
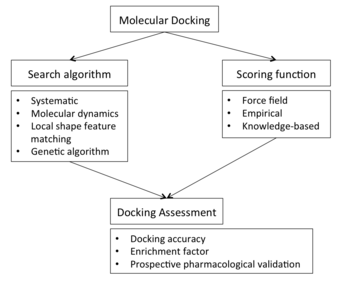
Aby wykonać ekran dokowania, pierwszym wymaganiem jest struktura białka będącego przedmiotem zainteresowania. Zwykle struktura została określona przy użyciu technik biofizycznych, takich jak krystalografia rentgenowska , spektroskopia NMR lub krio-mikroskopia elektronowa (cryo-EM) , ale może również pochodzić z modelowania homologii konstrukcji. Ta struktura białka i baza danych potencjalnych ligandów służą jako dane wejściowe do programu dokującego. Sukces programu dokującego zależy od dwóch elementów: algorytmu wyszukiwania i funkcji oceniania .
Algorytm wyszukiwania
Przestrzeń poszukiwań teoretycznie składa się ze wszystkich możliwych orientacji i konformacji białka sparowanego z ligandem. Jednak w praktyce przy obecnych zasobach obliczeniowych niemożliwe jest wyczerpujące zbadanie przestrzeni poszukiwań — wymagałoby to wyliczenia wszystkich możliwych zniekształceń każdej cząsteczki (cząsteczki są dynamiczne i występują w zespole stanów konformacyjnych) oraz wszystkich możliwych orientacji rotacyjnych i translacyjnych cząsteczek . ligand w stosunku do białka na danym poziomie ziarnistości . Większość używanych programów dokujących uwzględnia całą przestrzeń konformacyjną liganda (ligand elastyczny), a kilka próbuje modelować elastyczny receptor białkowy. Każda „migawka” pary jest określana jako poza .
Do ligandu i receptora zastosowano różne strategie wyszukiwania konformacyjnego. Obejmują one:
- systematyczne lub stochastyczne poszukiwania torsyjne dotyczące wiązań obrotowych
- symulacje dynamiki molekularnej
- algorytmy genetyczne do „ewoluowania” nowych konformacji o niskim zużyciu energii i gdzie wynik każdej pozy działa jako funkcja dopasowania używana do wybierania osobników do następnej iteracji.
Elastyczność liganda
Konformacje ligandu mogą być generowane pod nieobecność receptora, a następnie dokowane lub konformacje mogą być generowane w locie w obecności wnęki wiążącej receptor lub z pełną elastycznością obrotową każdego kąta dwuściennego przy użyciu dokowania opartego na fragmentach. Ocena energii pola siłowego jest najczęściej używana do wybierania konformacji rozsądnych energetycznie, ale stosowano również metody oparte na wiedzy.
Peptydy są zarówno bardzo elastycznymi, jak i stosunkowo dużymi cząsteczkami, co sprawia, że modelowanie ich elastyczności jest trudnym zadaniem. Opracowano szereg metod umożliwiających skuteczne modelowanie elastyczności peptydów podczas dokowania białko-peptyd.
Elastyczność receptora
Możliwości obliczeniowe znacznie wzrosły w ciągu ostatniej dekady, umożliwiając zastosowanie bardziej wyrafinowanych i wymagających dużej mocy obliczeniowej metod w wspomaganym komputerowo projektowaniu leków. Jednak radzenie sobie z elastycznością receptorów w metodologiach dokowania jest nadal drażliwą kwestią. Główną przyczyną tej trudności jest duża liczba stopni swobody, które należy uwzględnić w tego rodzaju obliczeniach. Zaniedbanie go jednak w niektórych przypadkach może prowadzić do słabych wyników dokowania pod względem przewidywania ułożenia wiązania.
Wiele struktur statycznych określonych eksperymentalnie dla tego samego białka w różnych konformacjach jest często używanych do naśladowania elastyczności receptora. Alternatywnie biblioteki rotamerów łańcuchów bocznych aminokwasów, które otaczają wnękę wiążącą, w celu wygenerowania alternatywnych, ale rozsądnych energetycznie konformacji białek.
Funkcja punktacji
Programy dokujące generują dużą liczbę potencjalnych pozycji ligandów, z których niektóre można natychmiast odrzucić z powodu kolizji z białkiem. Pozostałe są oceniane przy użyciu funkcji punktacji, która przyjmuje pozę jako dane wejściowe i zwraca liczbę wskazującą prawdopodobieństwo, że pozycja reprezentuje korzystną interakcję wiązania i klasyfikuje jeden ligand względem drugiego.
Większość funkcji punktacji to oparte na fizyce pola siłowe mechaniki molekularnej , które szacują energię pozy w miejscu wiązania. Różne wkłady w wiązanie można zapisać jako równanie addytywne:
Składowe składają się z efektów rozpuszczalnika, zmian konformacyjnych w białku i ligandzie, energii swobodnej wynikającej z interakcji białko-ligand, rotacji wewnętrznych, energii asocjacji ligandu i receptora w celu utworzenia pojedynczego kompleksu oraz energii swobodnej wynikającej ze zmian modów wibracyjnych. Niska (ujemna) energia wskazuje na stabilny system, a tym samym na prawdopodobną interakcję wiązania.
Alternatywne podejścia wykorzystują zmodyfikowane funkcje punktacji, aby uwzględnić ograniczenia oparte na znanych kluczowych interakcjach białko-ligand lub potencjały oparte na wiedzy pochodzące z interakcji obserwowanych w dużych bazach danych struktur białko-ligand (np. Protein Data Bank ) .
Istnieje duża liczba struktur z krystalografii rentgenowskiej dla kompleksów między białkami i ligandami o wysokim powinowactwie, ale stosunkowo mniej dla ligandów o niskim powinowactwie, ponieważ te ostatnie kompleksy są zwykle mniej stabilne, a zatem trudniejsze do krystalizacji. Funkcje oceniania wytrenowane na podstawie tych danych mogą prawidłowo dokować ligandy o wysokim powinowactwie, ale dadzą również wiarygodne zadokowane konformacje dla ligandów, które się nie wiążą. Daje to dużą liczbę fałszywie dodatnich , tj. ligandów, które zgodnie z przewidywaniami mają wiązać się z białkiem, a które w rzeczywistości nie wiążą się, gdy są umieszczane razem w probówce.
Jednym ze sposobów zmniejszenia liczby fałszywych trafień jest ponowne obliczenie energii najwyżej ocenianych póz przy użyciu (potencjalnie) dokładniejszych, ale bardziej intensywnych obliczeniowo technik, takich jak metody uogólnionego urodzenia lub metody Poissona-Boltzmanna .
Ocena dokowania
Współzależność między funkcją pobierania próbek a punktacją wpływa na zdolność dokowania w przewidywaniu prawdopodobnych pozycji lub powinowactwa wiązania dla nowych związków. W związku z tym generalnie wymagana jest ocena protokołu dokowania (gdy dostępne są dane eksperymentalne) w celu określenia jego zdolności predykcyjnej. Ocenę dokowania można przeprowadzić przy użyciu różnych strategii, takich jak:
- obliczenia dokładności dokowania (DA);
- korelacja między wynikiem dokowania a odpowiedzią eksperymentalną lub określeniem współczynnika wzbogacenia (EF);
- odległość między ugrupowaniem wiążącym jon a jonem w miejscu aktywnym;
- obecność modeli dopasowania indukowanego.
Dokładność dokowania
Dokładność dokowania stanowi jedną z miar ilościowego określenia sprawności programu dokowania poprzez racjonalizację zdolności przewidywania właściwej pozycji ligandu w odniesieniu do obserwowanej eksperymentalnie.
Czynnik wzbogacający
Ekrany dokujące można również ocenić poprzez wzbogacenie przypisanych ligandów znanych substancji wiążących spośród dużej bazy danych przypuszczalnie niewiążących cząsteczek „ wabików ”. W ten sposób sukces ekranu dokującego jest oceniany na podstawie jego zdolności do wzbogacenia niewielkiej liczby znanych związków aktywnych w najwyższych rzędach ekranu spośród znacznie większej liczby cząsteczek wabików w bazie danych. Obszar pod charakterystyki działania odbiornika (ROC) jest szeroko stosowany do oceny jego wydajności.
Spodziewany
IC50 są poddawane walidacji farmakologicznej (np. pomiary , powinowactwa lub siły działania ). Dopiero badania prospektywne stanowią rozstrzygający dowód przydatności techniki do określonego celu. W przypadku receptorów sprzężonych z białkiem G (GPCR), które są celem ponad 30% leków dostępnych na rynku, dokowanie molekularne doprowadziło do odkrycia ponad 500 ligandów GPCR.
Analiza porównawcza
Potencjał programów dokujących do odtwarzania trybów wiązania, jak określono za pomocą krystalografii rentgenowskiej, można ocenić za pomocą szeregu zestawów wzorcowych dokowania.
W przypadku małych cząsteczek istnieje kilka zestawów danych wzorcowych do dokowania i wirtualnych badań przesiewowych, np. Astex Diverse Set składający się z wysokiej jakości rentgenowskich struktur krystalicznych białek-ligandów lub Directory of Useful Decoys (DUD) do oceny wydajności wirtualnego skriningu.
Ocenę programów dokujących pod kątem ich potencjału do odtwarzania trybów wiązania peptydów można ocenić za pomocą Lessons for Efficiency Assessment of Docking and Scoring (LEADS-PEP).
Aplikacje
Wiążąca interakcja między małocząsteczkowym ligandem a białkiem enzymu może skutkować aktywacją lub hamowaniem enzymu. Jeśli białko jest receptorem, wiązanie ligandu może skutkować agonizmem lub antagonizmem . Dokowanie jest najczęściej stosowane w dziedzinie projektowania leków — większość leków to małe cząsteczki organiczne , a dokowanie można zastosować do:
- identyfikacja trafień – dokowanie w połączeniu z funkcją oceniania może być wykorzystane do szybkiego przeszukiwania dużych baz danych potencjalnych leków in silico w celu identyfikacji cząsteczek, które prawdopodobnie zwiążą się z docelowym białkiem będącym przedmiotem zainteresowania (patrz skrining wirtualny ). Farmakologia odwrotna rutynowo wykorzystuje dokowanie do identyfikacji celu.
- optymalizacja ołowiu - dokowanie można wykorzystać do przewidywania, gdzie iw jakiej względnej orientacji ligand wiąże się z białkiem (nazywany również trybem wiązania lub pozy). Informacje te mogą z kolei zostać wykorzystane do zaprojektowania silniejszych i selektywnych analogów.
- bioremediacja - dokowanie ligandów białkowych może być również wykorzystywane do przewidywania zanieczyszczeń, które mogą być rozkładane przez enzymy.
Zobacz też
- Projekt leku
- Algorytm Katchalskiego-Katzira
- Lista systemów grafiki molekularnej
- Dokowanie makromolekularne
- Mechanika molekularna
- Struktura białka
- Projekt białka
- Oprogramowanie do modelowania mechaniki molekularnej
- Lista oprogramowania dokującego białko-ligand
- Oprogramowanie do projektowania molekularnego
- Dokowanie@Dom
- Ibercivis
- Baza danych ZINC
- Wyszukiwarka leadów
- Wirtualny pokaz
- Funkcje punktacji dla dokowania
Linki zewnętrzne
-
Bikadi Z, Kovacs S, Demko L, Hazai E. „Molecular Docking Server - Ligand Protein Docking & Molecular Modeling” . Firma Virtua Drug Sp . Źródło 2008-07-15 .
Serwis internetowy, który oblicza miejsce, geometrię i energię małych cząsteczek oddziałujących z białkami
- Malinauskas T. „Instalacja krok po kroku MGLTools 1.5.2 (AutoDockTools, Python Molecular Viewer i Visual Programming Environment) w systemie Ubuntu Linux 8.04” . Zarchiwizowane od oryginału w dniu 2009-02-26 . Źródło 2008-07-15 .
- Docking@GRID Project of Conformational Sampling and Docking on Grids: jednym z celów jest wdrożenie pewnych wewnętrznych rozproszonych algorytmów dokowania w sieciach obliczeniowych, pobierz Docking@GRID open-source w wersji Linux
- Click2Drug.org - Katalog narzędzi do obliczeniowego projektowania leków.
- Ligand: Dokowanie receptora z MOE (molekularne środowisko operacyjne)