Kryminalistyczna analiza DNA
| Część serii poświęconej |
| kryminalistyce |
|---|
 |
Profilowanie DNA to określenie profilu DNA do celów prawnych i dochodzeniowych. Metody analizy DNA zmieniały się wiele razy na przestrzeni lat wraz z rozwojem technologii i pozwalają na określenie większej ilości informacji przy mniejszej ilości materiału wyjściowego. Współczesna analiza DNA opiera się na statystycznym obliczeniu rzadkości wytworzonego profilu w populacji.
Choć najbardziej znane jako narzędzie w dochodzeniach kryminalistycznych, profilowanie DNA może być również wykorzystywane do celów niezwiązanych z kryminalistyką, takich jak testy na ojcostwo i badania genealogiczne ludzi .
Historia
Metody tworzenia profilu DNA zostały opracowane przez Aleca Jeffreysa i jego zespół w 1984 roku.
Metody
Emerytowane metody
Analiza RFLP
Pierwszą prawdziwą metodą profilowania DNA była analiza polimorfizmu długości fragmentów restrykcyjnych. Pierwsze zastosowanie analizy RFLP w sprawach kryminalistycznych miało miejsce w 1985 roku w Wielkiej Brytanii. Ten typ analizy wykorzystywał powtórzenia tandemowe o zmiennej liczbie (VNTR) do rozróżniania osobników. VNTR są powszechne w całym genomie i składają się z tej samej sekwencji DNA powtarzanej wielokrotnie. Różne osoby mogą mieć różną liczbę powtórzeń w określonym miejscu w genomie. Na przykład osoba A może mieć cztery, podczas gdy osoba B może mieć 5 powtórzeń. Różnice uwidoczniono za pomocą procesu zwanego elektroforezą żelową . Mniejsze fragmenty przemieszczałyby się dalej przez żel niż oddzielające je większe fragmenty. Różnice te wykorzystano do rozróżnienia poszczególnych osób, a gdy przeprowadzono wiele ośrodków VNTR razem, analiza RFLP ma wysoki stopień indywidualizacji.
Proces analizy RFLP był niezwykle czasochłonny, a ze względu na długość użytych powtórzeń, od 9 do 100 par zasad, nie można było zastosować metod amplifikacji, takich jak reakcja łańcuchowa polimerazy . To ograniczyło RFLP do próbek, które miały już dostępną większą ilość DNA na początek i nie radziły sobie dobrze z zdegradowanymi próbkami. Analiza RFLP była podstawowym typem analizy przeprowadzanej w większości laboratoriów kryminalistycznych, zanim ostatecznie została wycofana i zastąpiona nowszymi metodami. Został całkowicie porzucony przez FBI w 2000 roku i zastąpiony analizą STR.
Testy alfa DQ
Opracowany w 1991 roku test DQ alfa był pierwszą techniką kryminalistyczną DNA, która wykorzystywała reakcję łańcuchową polimerazy. Ta technika pozwoliła na użycie znacznie mniejszej liczby komórek niż analiza RFLP, co czyni ją bardziej przydatną w przypadku miejsc zbrodni, które nie zawierały dużych ilości materiału DNA, który był wcześniej wymagany. Locus DQ alfa 1 (lub lokalizacja) również był polimorficzny i miał wiele różnych alleli , które można było wykorzystać do ograniczenia puli osobników, które mogły dać taki wynik i zwiększyć prawdopodobieństwo wykluczenia.
Locus DQ alfa połączono z innymi loci w komercyjnie dostępnym zestawie o nazwie Polymarker w 1993 roku. Polymarker był prekursorem nowoczesnych zestawów multipleksujących i umożliwił badanie wielu różnych loci za pomocą jednego produktu. Chociaż bardziej czuły niż analiza RFLP, Polymarker nie zawierał takiej samej mocy dyskryminacyjnej jak starsze testy RFLP. Do 1995 roku naukowcy próbowali powrócić do analizy opartej na VNTR połączonej z technologią PCR, zwaną polimorfizmem długości amplifikowanych fragmentów (AmpFLP).
AmFLP

AmpFLP był pierwszą próbą połączenia analizy VNTR z PCR na potrzeby analizy kryminalistycznej. Ta metoda wykorzystywała krótsze VNTR niż analiza RFLP, od 8 do 16 par zasad. Krótsze rozmiary par zasad AmpFLP zostały zaprojektowane tak, aby lepiej współpracowały z procesem amplifikacji PCR. Oczekiwano, że ta technika pozwoli na moc dyskryminacyjną analizy RFLP z możliwością przetwarzania próbek, które mają mniej matrycowego DNA do pracy lub które zostały zdegradowane w inny sposób. Jednak tylko kilka loci zostało zweryfikowanych pod kątem współpracy aplikacji kryminalistycznych z analizą AmpFLP, ponieważ laboratoria kryminalistyczne szybko przestawiły się na inne techniki, co ograniczyło ich zdolność do rozróżniania próbek kryminalistycznych.
Technika ta ostatecznie nigdy nie była szeroko stosowana, chociaż nadal jest używana w mniejszych krajach ze względu na niższy koszt i prostszą konfigurację w porównaniu z nowszymi metodami. Pod koniec lat 90. laboratoria zaczęły przechodzić na nowsze metody, w tym analizę STR. Wykorzystywały one jeszcze krótsze fragmenty DNA i mogły być bardziej niezawodnie amplifikowane przy użyciu PCR, przy jednoczesnym zachowaniu i poprawie mocy dyskryminacyjnej starszych metod.
Obecne metody
analiza STR

Analiza krótkich powtórzeń tandemowych (STR) jest podstawowym rodzajem kryminalistycznej analizy DNA przeprowadzanej w nowoczesnych laboratoriach DNA. Analiza STR opiera się na RFLP i AmpFLP stosowanych w przeszłości, zmniejszając rozmiar powtarzających się jednostek do 2 do 6 par zasad i łącząc wiele różnych loci w jednej reakcji PCR. Te zestawy testów multipleksujących mogą wytwarzać wartości alleli dla dziesiątek różnych loci w całym genomie, jednocześnie ograniczając ilość czasu potrzebnego do uzyskania pełnego, indywidualizującego profilu. Analiza STR stała się złotym standardem profilowania DNA i jest szeroko stosowana w kryminalistyce.
STR można również ograniczyć tylko do chromosomu Y. Analiza Y-STR może być stosowana w przypadkach dotyczących ojcostwa lub w badaniach rodzinnych , ponieważ chromosom Y jest identyczny w linii ojcowskiej (z wyjątkiem przypadków, w których wystąpiła mutacja ). Niektóre zestawy multipleksujące łączą loci autosomalne i Y-STR w jeden zestaw, co jeszcze bardziej skraca czas potrzebny do uzyskania dużej ilości danych.
Obecnie analiza STR wymaga wielu komórek do stworzenia pełnego profilu DNA. Jednak nauka jest coraz bliżej stworzenia pełnego profilu DNA za pomocą analizy STR na pojedynczych komórkach.
sekwencjonowanie mtDNA
Sekwencjonowanie mitochondrialnego DNA to specjalistyczna technika wykorzystująca oddzielne mitochondrialne DNA obecne w większości komórek. To DNA jest przekazywane w linii matczynej i nie jest unikalne między osobnikami. Jednak ze względu na liczbę mitochondriów obecnych w komórkach analizę mtDNA można zastosować do próbek silnie zdegradowanych lub próbek, w których analiza STR nie dostarczyłaby wystarczającej ilości danych, aby była użyteczna. mtDNA jest również obecny w miejscach, w których nie występuje autosomalny DNA, na przykład w łodygach włosów.
Ze względu na zwiększone ryzyko zanieczyszczenia w przypadku mtDNA niewiele laboratoriów przetwarza próbki mitochondriów. Te, które mają specjalne protokoły, które dodatkowo oddzielają od siebie różne próbki, aby uniknąć zanieczyszczenia krzyżowego.
Szybkie DNA
Rapid DNA to technologia „swab in-profile out”, która całkowicie automatyzuje cały proces ekstrakcji, amplifikacji i analizy DNA. Instrumenty Rapid DNA są w stanie przejść od wymazu do profilu DNA w zaledwie 90 minut i eliminują potrzebę przeprowadzania tego procesu przez przeszkolonych naukowców. Narzędzia te są rozważane pod kątem wykorzystania w procesie rezerwacji sprawcy, umożliwiając funkcjonariuszom policji uzyskanie profilu DNA osoby aresztowanej.
Niedawno w Stanach Zjednoczonych uchwalono ustawę Rapid DNA Act z 2017 r., która nakazuje FBI stworzenie protokołów wdrożenia tej technologii na terenie całego kraju. Obecnie DNA uzyskane z tych instrumentów nie kwalifikuje się do przesłania do krajowych baz danych DNA, ponieważ nie analizują one wystarczającej liczby loci, aby spełnić standardowy próg. Jednak wiele agencji policyjnych już korzysta z narzędzi Rapid DNA do pobierania próbek od osób aresztowanych na ich obszarze. Te lokalne bazy danych DNA nie podlegają przepisom federalnym ani stanowym.
Masowo równoległe sekwencjonowanie
Znane również jako sekwencjonowanie nowej generacji, sekwencjonowanie masowo równoległe (MPS) opiera się na analizie STR poprzez wprowadzenie bezpośredniego sekwencjonowania loci. Zamiast liczby powtórzeń obecnych w każdym miejscu, MPS dałoby naukowcowi rzeczywistą sekwencję par zasad. Teoretycznie MPS ma zdolność rozróżniania bliźniaków jednojajowych, ponieważ przypadkowe mutacje punktowe byłyby widoczne w powtarzających się segmentach, które nie zostałyby wykryte przez tradycyjną analizę STR.
Rzadkość profilu
Kiedy profil DNA jest używany w sposób dowodowy, dostarczana jest statystyka dopasowania, która wyjaśnia, jak rzadki jest profil w populacji. W szczególności ta statystyka to prawdopodobieństwo, że osoba wybrana losowo z populacji miałaby ten specyficzny profil DNA. Nie jest to prawdopodobieństwo, że profil „pasuje” do kogoś . Istnieje wiele różnych metod określania tej statystyki, a każda z nich jest używana przez różne laboratoria w oparciu o ich doświadczenie i preferencje. Jednak obliczenia ilorazu wiarygodności stają się metodą preferowaną w stosunku do pozostałych dwóch najczęściej stosowanych metod, przypadkowego człowieka bez wykluczenia i połączonego prawdopodobieństwa włączenia. Statystyki dopasowania są szczególnie ważne w interpretacji mieszanin, w których istnieje więcej niż jeden współautor profilu DNA. Kiedy te statystyki są podawane na sali sądowej lub w raporcie laboratoryjnym, zwykle dotyczą trzech najpopularniejszych ras z tego konkretnego obszaru. Dzieje się tak, ponieważ częstości alleli w różnych loci zmieniały się w zależności od pochodzenia danej osoby. https://strbase.nist.gov/training/6_Mixture-Statistics.pdf
Przypadkowy mężczyzna nie wykluczony
Prawdopodobieństwo uzyskane tą metodą to prawdopodobieństwo, że osoby losowo wybranej z populacji nie można wykluczyć z analizowanych danych. Ten rodzaj statystyki dopasowania jest łatwy do wyjaśnienia na sali sądowej osobom, które nie mają żadnego wykształcenia naukowego, ale traci również wiele mocy dyskryminacyjnej, ponieważ nie bierze pod uwagę genotypu podejrzanego. To podejście jest powszechnie stosowane, gdy próbka jest zdegradowana lub zawiera tak wielu czynników, że nie można określić pojedynczego profilu. Jest to również przydatne w wyjaśnianiu laikom, ponieważ metoda uzyskiwania statystyki jest prosta. Jednak ze względu na ograniczoną moc dyskryminacyjną RMNE nie jest generalnie przeprowadzana, chyba że nie można zastosować innej metody. Nie zaleca się stosowania RMNE w danych wskazujących na obecność mieszaniny.
Połączone prawdopodobieństwo włączenia/wyłączenia
Połączone prawdopodobieństwo włączenia lub wykluczenia oblicza prawdopodobieństwo, że przypadkowa, niepowiązana osoba byłaby współtwórcą profilu DNA lub mieszaniny DNA. W tej metodzie statystyki dla każdego pojedynczego locus są określane za pomocą statystyk populacji, a następnie łączone w celu uzyskania całkowitego CPI lub CPE. Obliczenia te są powtarzane dla wszystkich dostępnych loci ze wszystkimi dostępnymi danymi, a następnie każda wartość jest mnożona razem, aby uzyskać całkowite połączone prawdopodobieństwo włączenia lub wyłączenia. Ponieważ wartości są mnożone razem, za pomocą CPI można uzyskać bardzo małe liczby. CPI lub CPE uważa się za dopuszczalne obliczenie statystyczne, gdy wskazana jest mieszanina. https://www.promega.com/-/media/files/resources/conference-proceedings/ishi-15/parentage-and-mixture-statistics-workshop/generalpopulationstats.pdf?la=en
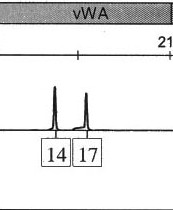
Przykładowe obliczenie dla pojedynczego profilu źródłowego
Prawdopodobieństwo rasy kaukaskiej posiadającej allel 16 przy vWA = 0,10204
Prawdopodobieństwo, że osoba rasy kaukaskiej ma allel 17 przy vWA = 0,26276
Prawdopodobieństwo, że osoba rasy kaukaskiej ma zarówno allel 14, jak i 17 (P) = 0,10204 + 0,26276 = 0,3648
Prawdopodobieństwo obecności wszystkich innych alleli (Q) = 1 - P lub 1 - 0,3648 = 0,6352
Prawdopodobieństwo wykluczenia dla vWA = Q 2 + 2Q(1-Q) lub .6352 2 + 2(.6352)(1 - .6352) = .86692096 ≈ 86,69%
Prawdopodobieństwo włączenia dla vWA = 1 - CPE lub 1 - 0,86692096 = 0,13307904 ≈ 13,31%
Przykładowe obliczenie profilu mieszanki
Prawdopodobieństwo, że osoba rasy kaukaskiej ma allel 14 przy vWA = 0,10204
Prawdopodobieństwo, że osoba rasy kaukaskiej ma allel 15 przy vWA = 0,11224
Prawdopodobieństwo, że osoba rasy kaukaskiej ma allel 16 przy vWA = 0,20153
Prawdopodobieństwo rasy kaukaskiej posiadającej allel 19 przy vWA = 0,08418
Prawdopodobieństwo rasy kaukaskiej posiadającej allel 14, 15, 16 lub 19 (P) = 0,10204 + 0,11224 + 0,20153 + 0,08418 = 0,49999
Prawdopodobieństwo obecności wszystkich innych alleli (Q) = 1 - P lub 1 - 0,49999 = 0,50001
Prawdopodobieństwo wykluczenia dla vWA = Q 2 + 2Q(1-Q) lub .50001 2 + 2(.50001)(1 - .50001) = .7500099999 ≈ 75%
Prawdopodobieństwo włączenia dla vWA = 1 - CPE lub 1 - 0,7500099999 = 0,2499900001 ≈ 25%
Współczynnik prawdopodobieństwa
Współczynniki wiarygodności (LR) to porównanie dwóch różnych prawdopodobieństw w celu określenia, które z nich jest bardziej prawdopodobne. Kiedy dotyczy procesu, LR jest prawdopodobieństwem argumentu oskarżenia w stosunku do prawdopodobieństwa argumentu obrony, biorąc pod uwagę ich początkowe założenia. W tym scenariuszu prawdopodobieństwo oskarżenia jest często równe 1, ponieważ zakłada się, że oskarżenie nie ściga podejrzanego, jeśli nie ma absolutnej pewności (100%), że ma właściwą osobę. Współczynniki wiarygodności stają się coraz bardziej powszechne w laboratoriach ze względu na ich przydatność w przedstawianiu statystyk dla danych wskazujących na wielu autorów, a także ich zastosowanie w oprogramowaniu do genotypowania probabilistycznego, które przewiduje najbardziej prawdopodobne kombinacje alleli na podstawie zestawu danych.
Wadą stosowania współczynników wiarygodności jest to, że bardzo trudno jest zrozumieć, w jaki sposób analitycy doszli do określonej wartości, a matematyka staje się bardzo skomplikowana, gdy do równań wprowadza się więcej danych. Aby zwalczyć te problemy na sali sądowej, niektóre laboratoria stworzyły „skalę słowną”, która zastępuje rzeczywistą wartość liczbową ilorazu wiarygodności.