Pirogronian, dikinaza fosforanowa
| Identyfikatory | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| pirogronianu, dikinazy fosforanowej | |||||||||
| nr WE | 2.7.9.1 | ||||||||
| Nr CAS | 9027-40-1 | ||||||||
| Bazy danych | |||||||||
| IntEnz | Widok IntEnz | ||||||||
| BRENDA | Wpis BRENDY | ||||||||
| EXPASY | Widok NiceZyme | ||||||||
| KEGG | Wpis KEGG | ||||||||
| MetaCyc | szlak metaboliczny | ||||||||
| PRYAM | profil | ||||||||
| Struktury WPD | RCSB PDB PDBe PDBsum | ||||||||
| Ontologia genów | AmiGO / QuickGO | ||||||||
| |||||||||
| Pirogronian, dikinaza fosforanowa | |
|---|---|
 Dimer dikinazy pirogronianowo-fosforanowej, Clostridium symbiosum ()
| |
| Identyfikatory | |
| Symbol | PPDK |
| InterPro | IPR010121 |
| Zobacz domeny poniżej. | |
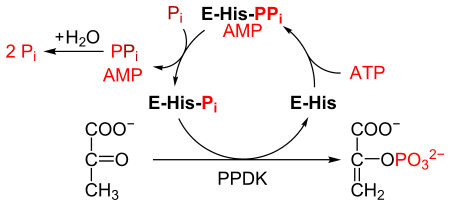
Pirogronian, dikinaza fosforanowa lub PPDK ( EC 2.7.9.1 ) to enzym z rodziny transferaz , który katalizuje reakcję chemiczną
- ATP + pirogronian + fosforan AMP + fosfoenolopirogronian + difosforan
Enzym ten badano głównie na roślinach, ale badano go również na niektórych bakteriach. Jest to kluczowy enzym w glukoneogenezie i fotosyntezie, odpowiedzialny za odwrócenie reakcji kinazy pirogronianowej w glikolizie Embdena-Meyerhofa-Parnasa. Nie należy go mylić z pirogronianem, dikinazą wodną .
Należy do rodziny transferaz , a konkretnie przenoszących grupy zawierające fosfor ( fosfotransferazy ) ze sparowanymi akceptorami ( dikinazy ). Enzym ten uczestniczy w metabolizmie pirogronianu i wiązaniu węgla .
Nomenklatura
Systematyczna nazwa tej klasy enzymów to ATP: pirogronian, fosfotransferaza fosforanowa. Inne powszechnie używane nazwy obejmują pirogronian, dikinaza ortofosforanowa, dikinaza pirogronianowo-fosforanowa (fosforylująca), dikinaza pirogronianowo-fosforanowa, nieorganiczna dikinaza pirogronianowo-fosforanowa, dikinaza pirogronianowo-fosforanowa, ligaza pirogronianowo-fosforanowa, dikinaza pirogronianowo-fosforanowa, ligaza pirogronianowo-fosforanowa, pirogronian , dikinaza Pi i PPDK.
Mechanizm reakcji
PPDK katalizuje konwersję pirogronianu do fosfoenolopirogronianu (PEP), zużywając 1 cząsteczkę ATP i wytwarzając w tym procesie jedną cząsteczkę AMP . Mechanizm składa się z 3 odwracalnych reakcji :
- Enzym PPDK wiąże się z ATP, tworząc AMP i difosforylowany PPDK.
- Difosforylowany PPDK wiąże się z nieorganicznym fosforanem , tworząc difosforan i (mono)fosforylowany PPDK.
- Fosforylowany PPDK wiąże się z pirogronianem, tworząc fosfoenolopirogronian i regenerując PPDK.
Reakcja jest podobna do reakcji katalizowanej przez kinazę pirogronianową , która również przekształca pirogronian w PEP. Jednakże kinaza pirogronianowa katalizuje nieodwracalną reakcję i nie zużywa ATP. Natomiast PPDK katalizuje odwracalną reakcję i zużywa 1 cząsteczkę ATP na każdą cząsteczkę przekształconego pirogronianu.
Obecnie szczegóły każdego etapu mechanicznego są nieznane
Struktura
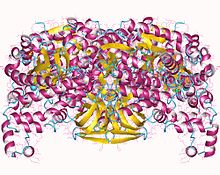
W swojej aktywnej postaci PPDK jest homotetramerem z podjednostkami o masie około 95 kDa
Istnieją dwa różne centra reakcji oddalone od siebie o około 45 Angstremów , w których wiążą się różne substraty . Miejsce wiązania nukleotydu (ATP) znajduje się na N-końcu , ma 240 aminokwasów i charakterystyczny uchwyt ATP. Miejsce wiązania pirogronianu/PEP znajduje się na C-końcu , ma 340 aminokwasów i fałd beczkowy α/β. Istnieje również domena centralna, która zawiera His 455, resztę pierwotną odpowiedzialny za katalizę. His455 jest resztą akceptora lub dawcy fosforylu. Struktura enzymu sugeruje, że ramię His455 podlega obrotowemu , aby przenieść grupę fosforylową pomiędzy dwoma centrami reakcji. Podczas tego obrotu domena centralna obraca się o co najmniej 92 stopnie i przesuwa się o 0,5 Angstremów.
Badania struktur krystalicznych PPDK pokazują, że domena centralna znajduje się w różnej odległości od dwóch pozostałych domen, w zależności od źródła enzymu. U kukurydzy znajduje się bliżej C-końca, natomiast u Clostridium symbiosum bliżej N-końca.
Badania wykazały, że mechanizmy wiązania PPDK są podobne do mechanizmów ligazy D-Ala-D-Ala i kinazy pirogronianowej . W szczególności PPDK jest bardzo podobny do kinazy pirogronianowej, która również katalizuje konwersję pirogronianu do fosfoenolopirogronianu; jednakże robi to bez pośredniego enzymu fosforylowanego. Chociaż ich sekwencje aminokwasów są różne, reszty kluczowe dla katalizy są zachowane w obu enzymach. z mutagenezą punktową wykazały, że reszty katalityczne obejmują Arg 561, Arg 617, Glu 745, Asn 768 i Cys 831 (numeracja w stosunku do C, białka symbiosum , ).
|
|
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Funkcja biologiczna i ewolucja
PPDK stosuje się w szlaku C4 w celu poprawy efektywności wiązania dwutlenku węgla . W środowiskach o dużej ilości światła tempo fotosyntezy u roślin jest ograniczone szybkością wchłaniania dwutlenku węgla (CO 2 ). Można to poprawić, stosując szereg reakcji chemicznych w celu transportu CO2 z komórek mezofilu (które znajdują się na zewnątrz liścia) do komórek osłonki (które znajdują się wewnątrz komórek). PPDK przekształca pirogronian w PEP, który reaguje z CO2 do produkcji szczawiooctanu . Kiedy CO2 jest uwalniany w komórkach osłonki wiązki, pirogronian ulega regeneracji i cykl jest kontynuowany.
Chociaż reakcja katalizowana przez PPDK jest odwracalna, PEP jest preferowanym produktem w warunkach biologicznych. Dzieje się tak za sprawą zasadowego pH w zrębie , gdzie zachodzi reakcja, a także wysokiego stężenia kinazy adenylanowej i pirofosfatazy . Ponieważ te dwa enzymy katalizują egzergoniczne z udziałem odpowiednio AMP i difosforanu, napędzają reakcję katalizowaną przez PPDK. Ponieważ PPDK zużywa ATP, szlak C4 jest niekorzystny dla roślin żyjących w środowiskach o małym dostępie do światła, ponieważ nie są one w stanie wytwarzać dużych ilości ATP.
PPDK występuje bardzo obficie w liściach C4 i zawiera do 10% całkowitego białka . Badania wykazały, że enzym jest identyczny w około 96% u różnych gatunków roślin. Eksperymenty hybrydyzacyjne wykazały, że różnice genetyczne korelują ze stopniem, w jakim rośliny realizują szlak C4 – w roślinach występują rzadkie sekwencje, które również wykazują cechy C3. PPDK występuje także w małych ilościach w roślinach C3. Historia ewolucji sugeruje, że kiedyś odgrywała ona rolę w glikolizie , podobnie jak podobna kinaza pirogronianowa , a ostatecznie ewoluowała w ścieżkę C4.
Oprócz roślin, PPDK występuje także w pasożytniczej ameobie Entamoeba histolytica () i bakterii Clostridium symbiosum (; a także w innych bakteriach). W tych dwóch organizmach PPDK działa podobnie (a czasami zamiast) kinazy pirogronianowej, katalizując reakcję w kierunku wytwarzania ATP w ramach glikolizy. Zaproponowano inhibitory Entamoeba PPDK jako amebicydy przeciwko temu organizmowi.
Rozporządzenie
Roślinne PPDK jest regulowane przez białko regulatorowe pirogronianu i dikinazy fosforanowej (PDRP). Gdy poziom światła jest wysoki, PDRP defosforyluje Thr 456 na PPDK przy użyciu AMP , aktywując w ten sposób enzym. PDRP dezaktywuje PPDK poprzez fosforylację tej samej reszty treoniny przy użyciu difosforanu . PDRP jest unikalnym regulatorem , ponieważ katalizuje zarówno aktywację, jak i dezaktywację PPDK poprzez dwa różne mechanizmy.
Badania nad PPDK kukurydzy sugerują, że introny , sekwencje terminatorowe i być może inne sekwencje wzmacniające działają wspólnie, zwiększając poziom funkcjonalnego i stabilnego mRNA . cDNA PPDK ulegało jedynie niewielkiej ekspresji w ryżu transgenicznym w porównaniu z nienaruszonym DNA, w którym zaobserwowano znaczącą ekspresję.
Badania strukturalne
Na początku 2018 r. rozwiązano 14 struktur dla tej klasy enzymów, z kodami dostępu PDB 1DIK , 1GGO , 1H6Z , 1JDE , 1KBL , 1KC7 , 1VBG , 1VBH , 2DIK , 2FM4 , 5JVJ , 5JVL , 5JVN , 5LU4 .
Dalsza lektura
- Hatch MD, Slack CR (styczeń 1968). „Nowy enzym do wzajemnej konwersji pirogronianu i fosfopirogronianu i jego rola w szlaku fotosyntezy kwasu dikarboksylowego C4” . Dziennik biochemiczny . 106 (1): 141–6. doi : 10.1042/bj1060141 . PMC 1198479 . PMID 4305612 .
- Reeves RE (czerwiec 1968). „Nowy enzym o funkcji glikolitycznej kinazy pirogronianowej” . Journal of Biological Chemistry . 243 (11): 3202–4. doi : 10.1016/S0021-9258(18)93395-8 . PMID 4297474 .
- Reeves RE (listopad 1971). „Pirogronian, dikinaza fosforanowa z Bacteroides symbiosus” . Dziennik biochemiczny . 125 (2): 531–9. doi : 10.1042/bj1250531 . PMC 1178089 . PMID 5144757 .
- Reeves RE, Menzies RA, Hsu DS (październik 1968). „Reakcja dikinazy pirogronianowo-fosforanowej. Losy fosforanów i równowaga” . Journal of Biological Chemistry . 243 (20): 5486–91. doi : 10.1016/S0021-9258(18)91972-1 . PMID 4302788 .