Technologia rekombinazy specyficznej dla miejsca
Technologie rekombinazy specyficznej dla miejsca to narzędzia inżynierii genomu , które zależą od enzymów rekombinazy w celu zastąpienia docelowych odcinków DNA.
Historia
Pod koniec lat 80. kierowanie genów do mysich embrionalnych komórek macierzystych (ESC) umożliwiło przenoszenie mutacji do mysiej linii zarodkowej i pojawiło się jako nowa opcja badania genetycznych podstaw sieci regulacyjnych istniejących w genomie. Nadal klasyczne celowanie w geny okazała się ograniczona na kilka sposobów, ponieważ funkcje genów zostały nieodwracalnie zniszczone przez gen markerowy, który musiał zostać wprowadzony do selekcji rekombinowanych ESC. Te wczesne etapy doprowadziły do powstania zwierząt, u których mutacja była obecna we wszystkich komórkach organizmu od samego początku, prowadząc do złożonych fenotypów i/lub wczesnej śmiertelności. Istniała wyraźna potrzeba opracowania metod ograniczających te mutacje do określonych punktów rozwoju i określonych typów komórek. To marzenie stało się rzeczywistością, gdy grupy w USA były w stanie wprowadzić bakteriofagi i pochodzące z drożdży systemy rekombinacji specyficznej dla miejsca (SSR-) do komórek ssaków, a także do myszy.
Klasyfikacja, właściwości i zastosowania dedykowane
Powszechne strategie inżynierii genetycznej wymagają trwałej modyfikacji docelowego genomu. W tym celu należy zainwestować wielkie wyrafinowanie w projektowanie dróg stosowanych do dostarczania transgenów. Chociaż dla celów biotechnologicznych integracja losowa jest nadal powszechna, może ona skutkować nieprzewidywalną ekspresją genów ze względu na zmienną liczbę kopii transgenu, brak kontroli nad miejscami integracji i związanymi z tym mutacjami. Wymagania molekularne w dziedzinie komórek macierzystych są znacznie bardziej rygorystyczne. Tutaj rekombinacja homologiczna (HR) może w zasadzie zapewniać specyficzność procesowi integracji, ale dla eukariotów jest to zagrożone przez wyjątkowo niską wydajność. Chociaż meganukleazy, nukleazy efektorowe typu palca cynkowego i aktywatory transkrypcji (ZFN i TALEN) są rzeczywistymi narzędziami wspierającymi HR, to dostępność rekombinaz specyficznych dla miejsca (SSR) zapoczątkowała racjonalną konstrukcję linii komórkowych o przewidywalnych właściwościach. Obecnie obie technologie, HR i SSR, można łączyć w wysoce wydajne technologie „tag-and-exchange”.
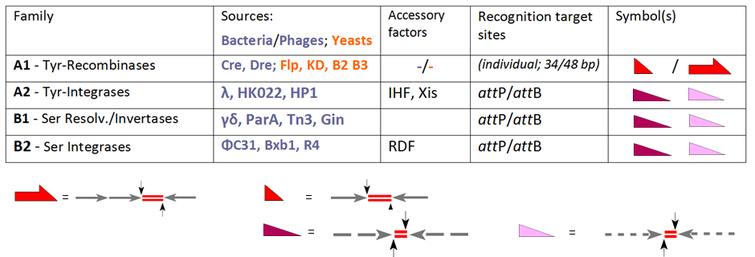
Zidentyfikowano wiele systemów rekombinacji specyficznych dla miejsca, które przeprowadzają te rearanżacje DNA do różnych celów, ale prawie wszystkie z nich należą do jednej z dwóch rodzin, rekombinaz tyrozynowych (YR) i rekombinaz serynowych (SR), w zależności od ich mechanizmu . Te dwie rodziny mogą pośredniczyć w maksymalnie trzech typach rearanżacji DNA (integracja, wycięcie/rozdzielczość i inwersja) wzdłuż różnych dróg reakcji w zależności od ich pochodzenia i architektury.
Członkiem założycielem rodziny YR jest integraza lambda, kodowana przez bakteriofaga λ , umożliwiająca integrację DNA faga z genomem bakteryjnym. Wspólną cechą tej klasy jest konserwatywny nukleofil tyrozyny atakujący rozszczepiony fosforan DNA, tworząc wiązanie 3'-fosfotyrozyny. Wcześni członkowie rodziny SR to blisko spokrewnione resolwazy / inwertazy DNA z bakteryjnych transpozonów Tn3 i γδ, które opierają się na katalitycznej serynie odpowiedzialnej za atakowanie fosforanu nożycowego w celu utworzenia wiązania 5'-fosfoseryny. Te niekwestionowane fakty zostały jednak naruszone przez spore zamieszanie w czasie, gdy inni członkowie weszli na scenę, na przykład rekombinazy YR Cre i Flp (zdolne do integracji, wycinania/rozdzielenia, jak również inwersji), które mimo to zostały powitane jako nowi członkowie „rodziny integraz”. Odwrotnymi przykładami są PhiC31 i pokrewne SR, które zostały pierwotnie wprowadzone jako resolwazy / inwertazy, chociaż przy braku czynników pomocniczych integracja jest ich jedyną funkcją. Obecnie standardowa aktywność każdego enzymu określa jego klasyfikację, zastrzegając ogólny termin „rekombinaza” dla członków rodziny, która jako taka obejmuje wszystkie trzy szlaki, INT, RES i INV:
Nasza tabela rozszerza wybór konwencjonalnych systemów SSR i grupuje je według ich wydajności. Wszystkie te enzymy rekombinują dwa miejsca docelowe, które są albo identyczne (podrodzina A1), albo różne (enzymy pochodzące z fagów w A2, B1 i B2). Podczas gdy dla A1 miejsca te mają indywidualne oznaczenia („ FRT ” w przypadku rekombinazy Flp, loxP dla rekombinazy Cre), terminy „ att P” i „ att B” (miejsca przyczepu odpowiednio na części fagowej i bakteryjnej) są ważne w pozostałych przypadkach. W przypadku podrodziny A1 mamy do czynienia z krótkimi (zwykle 34 pz-) miejscami składającymi się z dwóch (prawie) identycznych ramion 13 pz (strzałki) flankujące przerywnik 8 pz (obszar skrzyżowania, oznaczony dubletami czerwonej linii). Należy zauważyć, że dla Flp dostępne jest alternatywne miejsce 48 pz z trzema ramionami, z których każde zawiera jednostkę Flp (tak zwany „protomer” ) att P- i att Miejsca B podlegają podobnym zasadom architektonicznym, ale tutaj ramiona wykazują tylko częściową tożsamość (wskazaną przez przerywane linie) i różnią się w obu przypadkach. Te cechy odpowiadają za istotne różnice:
- rekombinacja dwóch identycznych miejsc eduktów prowadzi do miejsc produktów o tym samym składzie, chociaż zawierają ramiona z obu substratów; konwersje te są odwracalne;
- w przypadku rekombinacji att P x att B krzyżowanie może zachodzić tylko między tymi komplementarnymi partnerami w procesach prowadzących do dwóch różnych produktów ( att P x att B → att R + att L) w sposób nieodwracalny.
Aby usprawnić ten rozdział, kolejne implementacje skupią się na dwóch rekombinazach (Flp i Cre) i tylko jednej integrazie (PhiC31), ponieważ ich spektrum obejmuje narzędzia, które obecnie są najczęściej używane do ukierunkowanych modyfikacji genomu. Zostanie to zrobione w ramach poniższego przeglądu.

Drogi reakcji
Integracja / rozdzielczość trybu i inwersja (INT / RES i INV) zależą od orientacji miejsc docelowych rekombinazy (RTS), wśród tych par att P i att B. Sekcja C wskazuje, w usprawniony sposób, sposób, w jaki pośredniczy rekombinaza wymianę kasety (RMCE) można osiągnąć za pomocą synchronicznych podwójnych zwrotnic (zamiast integracji, po której następuje rozdzielczość).
Rekombinazy Tyr są odwracalne, podczas gdy Ser-Integraza jest jednokierunkowa. Godny uwagi jest sposób, w jaki odwracalna integracja/rozdzielczość Flp (rekombinaza Tyr) jest modulowana przez 48 pz (zamiast minimalnych 34 pz) FRT : dodatkowe ramię 13 pz służy jako „ścieżka lądowania” Flp, przyczyniając się do tworzenia kompleks synaptyczny, zarówno w kontekście funkcji Flp-INT, jak i Flp-RMCE (patrz odpowiednie sytuacje równowagi). Chociaż prawie niemożliwe jest zapobieżenie (napędzanemu entropią) odwróceniu integracji w sekcji A dla Cre i trudne do osiągnięcia dla Flp, RMCE można zakończyć, jeśli plazmid donorowy zostanie dostarczony w nadmiarze ze względu na dwucząsteczkowy charakter zarówno przedniego - i reakcja odwrotna. Pozowanie obu FRT w odwrotny sposób doprowadzą do równowagi obu orientacji wstawki (zielona strzałka). W przeciwieństwie do Flp, integraza Ser PhiC31 (reprezentacje na dole) prowadzi do integracji jednokierunkowej, przynajmniej przy braku czynnika kierunkowości rekombinazy (RDF-). W stosunku do Flp-RMCE, który wymaga dwóch różnych („heterospecyficznych”) FRT -spacer, partnera reakcji ( att B) pierwszego reagującego att Miejsce P jest uderzane arbitralnie, tak że nie ma kontroli nad kierunkiem, w którym kaseta dawcy wchodzi do celu (por. produkty alternatywne). Również w odróżnieniu od Flp-RMCE , kilka różnych celów RMCE nie może być montowanych równolegle, ze względu na brak heterospecyficznych (nieoddziałujących krzyżowo) kombinacji att P/ att B.
Rekombinaza Cre
Rekombinaza Cre (Cre) jest zdolna do rekombinacji określonych sekwencji DNA bez potrzeby stosowania kofaktorów . Enzym rozpoznaje sekwencje DNA o długości 34 par zasad zwane loxP („miejsce krzyżowania w fagu P1”). W zależności od orientacji miejsc docelowych względem siebie, Cre będzie integrować/wycinać lub odwracać sekwencje DNA. Po wycięciu (zwane „rozdzieleniem” w przypadku kolistego substratu) określonego regionu DNA normalna ekspresja genów jest znacznie upośledzona lub zakończona.
Ze względu na wyraźną aktywność rozdzielającą Cre, jednym z jego początkowych zastosowań było wycięcie genów lox z flankami P („floxed”), co doprowadziło do specyficznego dla komórki nokautu genu takiego floksowanego genu po tym, jak Cre ulegnie ekspresji w tkance będącej przedmiotem zainteresowania. Obecne technologie obejmują metody, które pozwalają zarówno na przestrzenną, jak i czasową kontrolę aktywności Cre. Powszechna metoda ułatwiająca przestrzenną kontrolę zmian genetycznych obejmuje wybór promotora specyficznego dla tkanki kierować ekspresją Cre. Umieszczenie Cre pod kontrolą takiego promotora skutkuje zlokalizowaną, tkankowo specyficzną ekspresją. Na przykład Leone i in. umieścili jednostkę transkrypcyjną pod kontrolą sekwencji regulatorowych genu mieliny (PLP), co doprowadziło do indukowanego usunięcia docelowych sekwencji genowych w oligodendrocytach i komórkach Schwanna . Specyficzny fragment DNA rozpoznawany przez Cre pozostaje nienaruszony w komórkach, które nie wykazują ekspresji genu PLP; to z kolei ułatwia empiryczną obserwację lokalnych skutków zmian genomu w osłonce mielinowej otaczającej włókna nerwowe w ośrodkowym układzie nerwowym (OUN) i obwodowym układzie nerwowym (PNS). Selektywną ekspresję Cre osiągnięto również w wielu innych typach komórek i tkanek.
opracowano formy Cre, które wykorzystują różne domeny wiążące ligandy . Jedna skuteczna strategia indukowania specyficznej czasowej aktywności Cre obejmuje fuzję enzymu ze zmutowaną domeną wiążącą ligand dla ludzkiego receptora estrogenowego (ERt). Po wprowadzeniu tamoksyfenu ( antagonisty receptora estrogenowego ) konstrukt Cre-ERt jest w stanie przeniknąć do jądra i wywołać ukierunkowaną mutację. ERt wiąże tamoksyfen z większym powinowactwem niż endogenne estrogeny , co pozwala Cre-ERt pozostać cytoplazmatycznym u zwierząt nieleczonych tamoksyfenem. Czasowa kontrola aktywności SSR przez tamoksyfen umożliwia indukowanie zmian genetycznych w późniejszej fazie embriogenezy i/lub w dorosłych tkankach. Pozwala to naukowcom ominąć śmiertelność embrionów, jednocześnie badając funkcję docelowych genów.
Niedawne rozszerzenie tych ogólnych koncepcji doprowadziło do wygenerowania „Cre-zoo”, tj. kolekcji setek szczepów myszy, dla których określone geny można usunąć przez ukierunkowaną ekspresję Cre.

rekombinaza Flp
W swoim naturalnym gospodarzu (S. cerevisiae) system Flp/ FRT umożliwia replikację „plazmidu 2 µ” przez odwrócenie segmentu, który jest flankowany przez dwa identyczne, ale przeciwnie zorientowane miejsca FRT (aktywność „flippazy”). Ta inwersja zmienia względną orientację widełek replikacyjnych w plazmidzie, umożliwiając „toczące się koło” - amplifikację okrągłej jednostki 2 μ, zanim multimeryczne związki pośrednie zostaną rozdzielone w celu uwolnienia wielu produktów monomerycznych. Podczas gdy minimalne miejsca FRT o wielkości 34 pz sprzyjają wycięciu/rozdzieleniu w podobnym stopniu jak lox analogowy Miejsca P dla Cre, naturalnych, bardziej rozciągniętych wariantów FRT o długości 48 bp, umożliwiają wyższy stopień integracji, przezwyciężając jednocześnie pewne rozwiązłe interakcje, jak opisano dla enzymów fagowych, takich jak Cre- i PhiC31. Dodatkową zaletą jest fakt, że można zastosować proste zasady do generowania heterospecyficznych FRT , które przechodzą krzyżowanie z równymi partnerami, ale nie z FRT typu dzikiego . Fakty te umożliwiły, od 1994 r., rozwój i ciągłe udoskonalanie wymiany kaset za pośrednictwem rekombinazy (RMCE-), umożliwiających czystą wymianę kasety docelowej na przychodzącą kasetę dawcy.
W oparciu o technologię RMCE, w ramach programu EUCOMM (European Conditional Mouse Mutagenesis) rozwinął się szczególny zasób wstępnie scharakteryzowanych szczepów ES, który nadaje się do dalszego opracowania, w oparciu o ustanowiony obecnie Cre- i/lub Flp- oparte na konfiguracjach „FlExing” (wycinanie / odwracanie za pośrednictwem Flp), obejmujące czynności wycinania i odwracania. Projekt ten, zainicjowany w 2005 r., koncentrował się najpierw na mutagenezie nasycenia, aby umożliwić pełną adnotację funkcjonalną genomu myszy (koordynowaną przez Międzynarodowe Konsorcjum Knockout-Mouse, IKMC), a ostatecznym celem było zmutowanie wszystkich genów białek poprzez pułapkowanie genów i celowanie u myszy komórki ES. Wysiłki te stanowią szczyt różnych strategii „oznacz i wymień”, które są przeznaczone do znakowania odrębnego miejsca genomowego, tak aby „znacznik” mógł służyć jako adres do wprowadzenia nowej (lub zmiany istniejącej) informacji genetycznej. Etap tagowania per se może odnosić się do pewnych klas miejsc integracyjnych, wykorzystując preferencje integracyjne retrowirusów lub nawet integraz specyficznych dla miejsca, takich jak PhiC31, z których oba działają w zasadniczo jednokierunkowy sposób.
Tradycyjne, pracochłonne procedury „znakowania i wymiany” opierały się na dwóch kolejnych etapach rekombinacji homologicznej (HR-), z których pierwszy („HR1”) polegał na wprowadzeniu znacznika składającego się z genu markera selekcyjnego. Następnie użyto „HR2” do zastąpienia markera przez „GOI. W pierwszej („knock-out” -) reakcji gen znakowano markerem selekcyjnym, zwykle przez wstawienie kasety hygtk ([+/-]) zapewniając oporność na G418. W następnym etapie „knock-in", znakowana sekwencja genomowa została zastąpiona homologicznymi sekwencjami genomowymi z pewnymi mutacjami. Klony komórkowe można było następnie wyizolować na podstawie ich oporności na gancyklowir z powodu utraty genu HSV-tk, tj. („selekcja negatywna”). Ta konwencjonalna dwuetapowa procedura oznaczania i wymiany mogłaby zostać usprawniona po pojawieniu się RMCE, który mógłby przejąć i zwiększyć wydajność etapu dobijania.
integraza PhiC31
Bez wątpienia integrazy Ser są obecnie wybieranymi narzędziami do integracji transgenów z ograniczoną liczbą dobrze poznanych genomowych miejsc akceptorowych, które w większości (ale nie zawsze) naśladują miejsce att P faga, ponieważ przyciągają wektor donorowy zawierający att B . W tej chwili najbardziej znanym członkiem jest PhiC31-INT o udowodnionym potencjale w kontekście ludzkich i mysich genomów.
W przeciwieństwie do powyższych rekombinaz Tyr, PhiC31-INT jako taki działa w sposób jednokierunkowy, mocno blokując wektor donorowy na genomowo zakotwiczonym celu. Oczywistą zaletą tego systemu jest to, że może on polegać na niezmodyfikowanych, natywnych att P (akceptor) i att B. Dodatkowe korzyści (wraz z pewnymi komplikacjami) mogą wynikać z faktu, że genomy myszy i ludzi per se zawierają ograniczoną liczbę endogennych celów (tzw . P-pseudosites”). Dostępne informacje sugerują, że znaczne wymagania dotyczące sekwencji DNA pozwalają integrazie rozpoznawać mniej miejsc niż systemy integracji oparte na retrowirusach lub nawet transpozazach, otwierając jej karierę jako doskonałego nośnika do transportu i insercji w wielu dobrze znanych miejscach genomowych , z których niektóre mają tak zwane właściwości „bezpiecznej przystani”.
Wykorzystując fakt specyficznych ( att P x att B) dróg rekombinacji, RMCE staje się możliwe bez wymagań dla syntetycznych, heterospecyficznych miejsc att . Ta oczywista zaleta odbywa się jednak kosztem pewnych niedociągnięć, takich jak brak kontroli nad rodzajem lub kierunkowością kasety wejściowej (dawcy). Dalsze ograniczenia wynikają z faktu, że nieodwracalność nie pozwala na standardowe multipleksowania-RMCE , w tym reakcje „szeregowego RMCE”, tj. wielokrotne wymiany kaset w danym locus genomowym .
Perspektywy i perspektywy
Adnotacja genomów człowieka i myszy doprowadziła do zidentyfikowania >20 000 genów kodujących białka i >3000 genów niekodujących RNA, które kierują rozwojem organizmu od zapłodnienia przez embriogenezę do dorosłego życia. Chociaż odnotowano dramatyczny postęp, znaczenie rzadkich wariantów genów pozostaje głównym tematem badań.
Jako jedna z najważniejszych platform zajmujących się funkcjami genów kręgowców na dużą skalę, ustalono zasoby genetyczne całego genomu zmutowanych mysich komórek ES. W tym celu w Europie i Ameryce Północnej powołano cztery międzynarodowe programy ukierunkowane na mutagenezę wysycającą genomu myszy (EUCOMM, KOMP, NorCOMM i TIGM). Koordynowane przez Międzynarodowe Konsorcjum Knockout Mouse (IKSC) te repozytoria komórek ES są dostępne do wymiany między międzynarodowymi jednostkami badawczymi. Obecne zasoby obejmują mutacje w 11 539 unikalnych genach, w tym 4 414 warunkowych.
Odpowiednie technologie osiągnęły obecnie poziom pozwalający na ich rozszerzenie na inne gatunki ssaków i ludzkie komórki macierzyste, przede wszystkim te o statusie iPS (indukowany pluripotencjalny) .
Zobacz też
- Rekombinacja specyficzna dla miejsca
- Wymiana kaset za pośrednictwem rekombinazy
- Rekombinaza Cre
- Rekombinacja Cre-Lox
- Rekombinacja FLP-FRT
- Rekombinacja genetyczna
- Rekombinacja homologiczna
Linki zewnętrzne
- http://www.knockoutmouse.org/
- Emes, RD; Goodstadt, L; zima, EE; Ponting, CP (2003). „Porównanie genomów człowieka i myszy stanowi podstawę zoologii genomu” . Hum Mol Genet . 12 (7): 701–9. doi : 10.1093/hmg/ddg078 . PMID 12651866 .